预约演示
更新于:2025-05-07
Jiangsu Baidu Medical Technology Co., Ltd.
注销
| 私营公司|2013|中国江苏省注销
| 私营公司|2013|中国江苏省更新于:2025-05-07
概览
关联
100 项与 Jiangsu Baidu Medical Technology Co., Ltd. 相关的临床结果
登录后查看更多信息
0 项与 Jiangsu Baidu Medical Technology Co., Ltd. 相关的专利(医药)
登录后查看更多信息
294
项与 Jiangsu Baidu Medical Technology Co., Ltd. 相关的新闻(医药)2025-04-29
一、2025 年 3 月 28 日,诺奖得主、蛋白质设计先驱 David Baker 教授在 Nature Methods 期刊发表了题为:Atomic context-conditioned protein sequence design using LigandMPNN 的研究论文。该研究开发了一种新型深度学习方法——LigandMPNN,该方法明确地对生物分子系统中的所有非蛋白质成分进行了建模,预计 LigandMPNN 将在设计新的结合蛋白、传感器和酶方面得到广泛应用。比Rosetta快250倍,亲和力提升百倍。蛋白质的从头设计,能够创造出具有新功能的新型蛋白质,例如催化作用、与 DNA、小分子和金属的结合以及蛋白质间的相互作用。二、在生物信息学领域,传统纯生信研究依赖大量实验,成本高、周期长,限制了研究进展。而随着人工智能技术飞速发展,机器学习算法展现出强大的数据处理与分析能力,为纯生信研究开辟新路径。将机器学习引入纯生信,有望突破实验瓶颈,高效挖掘生物数据价值,助力科研人员更深入理解生命奥秘,在疾病筛查、药物研发等方面发挥关键作用,这一融合趋势成为科研界关注焦点。shop十大热门专题助你发顶刊01AI蛋白质设计02合成生物与基因电路设计03CADD计算机辅助药物设计04AIDD人工智能药物设计与发现05机器学习代谢组学06机器学习微生物组学07深度学习基因组学08深度学习质谱蛋白组学09CRISPR-Cas9基因编辑技术10AI智慧医疗影像技术shop以下为具体内容介绍01AI蛋白质设计内容可向下滑动第一天蛋白质结构及分子动力学基础a)蛋白质设计概述b)蛋白质结构基础 pdb文件格式详解数据库详解c)分子动力学基础分子-蛋白对接蛋白-蛋白对接可视化软件pymol使用AMBER分子动力学模拟高斯加速分子动力学模拟模拟轨迹分析MM-GB/PBSA方法计算结合自由能分子动力学模拟在酶改造中的应用第二天a)Python、Numpy基础b)经典模型线性及非线性映射:线性回归、逻辑回归聚类:K-近邻聚类核方法:支持向量机树方法:决策树、随机森林神经网络:多层感知机scikit-learn 基本调用实操c)深度学习深度神经网络DNN卷积神经网络CNN循环神经网络RNNd)前沿架构及复现TransformerBERTGPTViT第三天蛋白结构预测及其下游应用a)蛋白结构预测背景介绍b)AlphafoldAF和AF2的差异与创新AF3的差异与创新Alphafold使用详解c)基于Alphafold的下游应用基于AF2的蛋白-蛋白对接/蛋白-多肽对接利用AF2做多构象预测和功能发现基于AF2的环肽设计d)trrosetta幻想设计e)基于ProteinMPNN的蛋白质设计MPNN模型简介设计流程方法比较第四天蛋白质的AI生成模型扩散模型a)RFdiffusion实现蛋白结构设计蛋白Binder生成蛋白骨架设计单体蛋白从头生成多聚体蛋白从头生成RFdiffusion在酶改造中的应用b)Chroma的基本构架与实现蛋白构象空间全空间采样c)ProteinGenerator蛋白质骨架与序列设计与rfdiffusion的异同蛋白语言模型a)ProGEN与ProGEN2模型构架讲解与基于结构方法的比较性能与改进 b)ESM-foldESM-fold的基本架构ESMfold网络讲解与alphafold方法的对比ESM-fold的性能评估c) 基于大语言模型的下游应用孤儿蛋白结构的预测大型蛋白复合物结构预测基于蛋白语言模型的酶设计第五天基于深度学习的蛋白质挖掘与改造a)基于alphafold的酶挖掘b)通过预测蛋白复合物发现生命过程新机制c)基于大语言模型的新酶挖掘d)深度学习从基因组挖掘全新蛋白e)综合:结合多种深度学习与计算工具的蛋白改造流程02合成生物与基因电路设计内容可向下滑动第一天合成生物学基础概念与应用领域剖析1.深度解读合成生物学精准定义,系统阐述其涵盖研究内容,沿着发展历程脉络回溯,展望未来趋势,全方位扫描应用领域。2.通过详实案例分析,将合成生物学在医药、农业、工业等实际场景的应用具象化,让学员感知其变革力量。生物元件功能精讲与标准化设计准则1.聚焦生物元件,如启动子精准调控转录起始、终止子界定转录终点、RBS 驱动核糖体结合开启翻译。2.结合 BioBrick 元件设计范式与应用实例,传授生物元件模块化设计黄金法则,助力学员掌握构建标准化生物模块的精髓。第二天基因线路逻辑架构搭建与实例演练1.开启基因线路逻辑世界大门,详细拆解与、或、非基本逻辑门运作原理及设计技巧。2.引入 iGEM 竞赛中经典逻辑门线路应用案例,手把手指导学员实践操作,使其能独立构思简单基因线路并洞悉其功能。复合元件整合与电路深度设计以前两天所学生物元件、基因线路知识为基石,引导学员梳理合成生物学核心玩法。亲手设计、搭建、分析经典电路,实现知识融会贯通,掌握复杂电路中各模块协同运作奥秘。第三天代谢途径精细构建与优化策略1.深入讲解代谢途径搭建步骤,剖析定向进化、代谢工程等优化策略。2.以青蒿素生物合成途径优化为典型案例,传授从理论到实践的代谢途径设计方法,助力学员提升生物合成效率。基因组合成与编辑技术前沿1.系统讲解基因组合成底层原理、多元方法,深度剖析 CRISPR/Cas9 等基因组编辑技术原理。2.结合海量案例,呈现基因组合成与编辑技术在定制生物、精准医疗等前沿领域的实际应用。第四天底盘生物特性解析与应用1.引入底盘生物概念,剖析常见底盘生物(如大肠杆菌、酵母菌等)特性。2.讲解其在基因线路搭载、基因表达中的关键作用,结合实例探讨如何依据项目需求选择适配底盘生物,实现高效基因操作。合成生物系统搭建与优化实践1.全方位讲解合成生物系统定义、分类,通过案例剖析系统构建流程,传授优化策略。2.组织实践操作,让学员亲身体验合成生物系统的构建与优化全过程,学会依据反馈调控系统参数。第五天合成生物学数学建模与性能分析1.开启生物系统数学建模之旅,介绍常微分方程、逻辑模型等常用建模方法。2.结合基因线路动力学模拟案例,指导学员运用数学模型精准分析生物系统性能,为系统设计与优化提供量化支撑。合成生物学全景总结与未来瞻望1.回溯课程所学知识,通过多领域案例分析,展现合成生物学应用广度与深度。2.组织学员热烈讨论未来发展趋势、潜在影响,着重介绍伦理、生物安全与生物安保等关键议题,培养学员全面视角。03CADD计算机辅助药物设计内容可向下滑动第一天1.CADD药物设计概论1.1计算机辅助药物设计的发源和基本概念1.2计算机辅助药物设计中常用的计算方法2. 基于结构的药物设计方法1.1 基于结构的药物设计方法概论1.2 蛋白质数据库的介绍:uniprot、PDB、AlphafldDB3. 高质量画图软件pymol 使用详解1.1 蛋白小分子相互作用、蛋白-蛋白相互作用绘制等1..2 蛋白及小分子表面图、静电势表示等第二天1.蛋白质结构处理方法1.1蛋白质结构预测方法理论介绍及使用1.2蛋白质缺失残基处理、残基质子态确定、单点突变等处理方法2.小分子结构处理方法1.1常用的小分子数据库介绍:zinc,chembl、pubchem、神农Alpha等1.2Chemdraw的使用及小分子性质计算第三天1. 蛋白-小分子对接的基本流程与技巧1.1 分子对接的原理1.2 基于autudock和autodock vina的分子对接步骤详解1.3 对接结果的分析与挑选技巧第四天1.特殊的分子对接的基本流程与技巧1.1蛋白-小分子柔性对接步骤详解1.2蛋白-蛋白/DNA/RNA,小分子-小分子对接方法详解2. 基于分子对接的虚拟筛选详解1.1 虚拟筛选的流程与处理1.2 虚拟筛选的结果分析与技巧第五天1.Openbael的使用1.1小分子不同的表示形式、文件格式介绍1.2Openbabel的文件格式转换、分子结构生成介绍2.ADMET介绍和反向找靶介绍3.QSAR模型介绍及操作1.1 QSAR模型的基本原理1.2 3DQSAR--COMFA和COMSIA模型的构建与结果分析第六天1.Linux系统操作1.1Linux系统介绍1.2Linux常用命令操作2.分子动力学模拟介绍1.1分子动力学模拟理论介绍1.2常用软件和一般流程介绍第七天1.MD实践1.1溶剂化下蛋白质分子动力学模拟1.2 溶剂化下蛋白质-配体的分子动力学模拟1.3 MD结果分析2. 答疑:1.1 课程知识答疑1.2 学员实际课题思路答疑部分模型案例图片04AIDD人工智能药物设计与发现内容可向下滑动第1天:环境搭建与深度学习基本知识讲解1.AIDD概述:从CADD到AIDD2.软件安装与环境搭建(1)anaconda(2)vscode(3)环境变量的配置(4)切换pip和conda镜像源(5)虚拟环境的创建3.RDKIT工具包的使用(1)基于RDKit的分子读写(2)基于RDKit的分子绘制(3)基于RDKit的分子指纹与分子描述符(4)基于RDKit的化合物相似性与子结构4.药物综合数据库的获取方法(1)基于requests的基本爬虫操作(2)小分子数据库PubChem数据获取(pubchempy / requests)(3)蛋白质数据库PDB、UniProt数据获取5.深度学习辅助药物设计(1)神经网络基本概念与sklearn工具包介绍(2)图神经网络与消息传递机制基本知识(3)Transformer模型基本知识:分词、位置编码、注意力机制、编码器、解码器、预训练-微调框架、huggingface 生态介绍(4)模型的评估与验证:准确率、精确率、召回率、F1分数、ROC曲线、AUC计算,平均绝对误差、均方差、R2分数、可释方差分数,交叉验证等第2天:顶刊复现专题1——分子与生化反应的表示学习与性质预测助力药物发现培训背景:在人工智能辅助药物发现(AIDD)中,分子与生化反应的表示学习与性质预测是整个研究流程的基石。分子的结构决定其功能,如何将复杂的分子结构和生化反应过程有效地表示为计算模型能够理解的形式,是实现高效预测和优化的前提。通过构建合理的分子表示(如图神经网络、SMILES编码、指纹等),我们可以让AI模型捕捉关键的化学特征,进而用于预测分子的物理化学性质、生物活性、毒性等,为后续的虚拟筛选、分子生成与反应设计提供可靠基础。因此,本专题不仅奠定了AIDD中建模与预测能力的核心能力框架,也为整个药物发现过程中的智能决策打下了坚实基础。培训内容1:Nature Machine Intelligence|基于注意力的神经网络在化学反应空间映射中的应用《Mapping the space of chemical reactions using attention-based neural networks》1.数据集1.1.Pistachio数据集:包含260万化学反应,来自专利数据,涵盖792个反应类别。数据经过去重和有效性过滤(使用RDKit)。1.2.USPTO 1k TPL数据集:基于USPTO专利数据,包含44.5万反应,通过原子映射和模板提取生成1,000个反应模板类别。1.3.Schneider 50k数据集:公开数据集,包含5万反应,50个类别,用于与传统指纹方法对比。2.模型。研究对比了两种Transformer架构:2.1.BERT分类器:基于编码器的模型,通过掩码语言建模预训练后,在分类任务上微调,使用[CLS]标记的嵌入作为反应指纹(rxnfp)。2.2.Seq2Seq模型:编码器-解码器结构,将分类任务分解为超类、类别和具体反应的层级预测。两者均采用简化版BERT(隐藏层256维),输入为未标注的SMILES序列,无需反应物-试剂区分或原子映射。3.训练。模型训练分为两步:3.1.预训练:BERT通过掩码SMILES令牌预测任务进行自监督学习,学习反应通用表示。3.2.微调:在分类任务上优化模型,使用交叉熵损失,学习率2×10⁻⁵,序列长度512。评估采用混淆熵(CEN)和马修斯相关系数(MCC)以处理数据不平衡。培训内容2:TOP期刊|基于深度学习的生化反应产量预测《Prediction of chemical reaction yields using deep learning》 1.数据。研究使用了三类数据:1.1.Buchwald-Hartwig HTE数据集:包含3955个Pd催化C-N偶联反应,涵盖15种卤化物、4种配体、3种碱和23种添加剂组合,产率通过统一实验测量,数据质量高。 1.2.Suzuki-Miyaura HTE数据集:包含5760个反应,涉及15对亲电/亲核试剂、12种配体、8种碱和4种溶剂的组合,产率分布均匀。 1.3.USPTO专利数据集:从公开专利中提取,包含不同规模(克级与亚克级)的反应产率,数据噪声大且分布不一致,需通过邻近反应产率平滑处理以提升模型表现。2.模型。核心模型基于预训练的rxnfp(反应指纹)BERT架构,新增回归层构成Yield-BERT。输入为标准化反应SMILES,通过自注意力机制捕捉反应中心及关键试剂的上下文信息。模型无需手工特征(如DFT计算描述符),直接端到端预测产率。实验表明,其性能优于传统方法(如随机森林和分子指纹拼接),尤其在HTE数据上接近化学描述符的预测水平,且参数鲁棒性高(超参数调整影响小)。3.训练。训练分为两步:3.1.预训练:BERT通过掩码语言任务学习SMILES的通用表示。 3.2.微调:采用简单Transformers库和PyTorch框架,以MSE损失优化回归层,学习率(2×10⁻⁵)和dropout率(0.1–0.8)为主要调参对象。HTE数据采用随机/时间划分验证,USPTO数据通过邻近反应产率平滑缓解噪声影响。小样本实验(5%训练数据)显示模型能快速筛选高产反应,指导合成优化。培训内容3:TOP期刊|基于T5Chem模型的生化反应表示学习与性质预测: 《Unified Deep Learning Model for Multitask Reaction Predictions with Explanation》1.数据来源和处理。通过自监督预训练与PubChem分子数据集进行训练,以实现对四种不同类型的化学反应预测任务的优异性能。模型处理包括反应类型分类、正向反应预测、单步逆合成和反应产率预测。2.模型架构和原理。T5Chem模型是基于自然语言处理中的“Text-to-Text Transfer Transformer”(T5)框架开发的统一深度学习模型,该模型通过适应T5框架来处理多种化学反应预测任务。T5Chem模型包含编码器-解码器结构,并根据任务类型引入了任务特定的提示和不同的输出层,如分子生成头、分类头和回归头,以处理序列到序列的任务、反应类型分类和产品产率预测。3.训练过程和细节。3.1.T5Chem模型首先在PubChem的97 million分子上进行自监督预训练,使用BERT类似的“masked language modeling”目标。3.2.在预训练阶段,源序列中的tokens被随机掩蔽,模型的目标是预测被掩蔽的正确的tokens。3.3.预训练完成后,模型在下游的监督任务中进行微调,使用不同的任务特定提示和输出层。3.4.模型在测试阶段通过生成分子token by token的方式进行预测,直到生成“句子结束标记”或达到最大预测长度。通过培训可以掌握的内容:1.分子与化学反应的表示方法。学习如何将分子和化学反应编码为机器可处理的格式,如SMILES(Simplified Molecular-Input Line-Entry System)和反应SMILES。理解分子指纹(如Morgan指纹)和反应指纹(如rxnfp)的构建方式,以及它们在化学信息学中的应用。掌握Transformer架构(如BERT)如何用于化学反应的特征提取,并生成具有化学意义的向量表示。2.深度学习在化学反应预测中的应用。了解如何利用序列到序列(Seq2Seq)模型和BERT进行化学反应分类(如反应类型识别)和产率预测。学习如何通过自注意力机制分析化学反应的关键部分(如反应中心、试剂影响),提高模型的可解释性。掌握如何利用预训练+微调策略,使模型在少量标注数据下仍能取得良好性能。3.数据驱动的化学研究范式。认识不同数据来源(如高通量实验HTE、专利数据USPTO)的特点及其对模型训练的影响。学习如何处理数据噪声,并通过数据平滑、邻近分析等方法优化模型表现。了解小样本学习在化学中的应用,例如如何用5%-10%的训练数据筛选高产率反应,指导实验优化。第3天:顶刊复现专题2——蛋白质的表示学习与性质预测助力药物发现培训背景:在AIDD中,蛋白质是药物作用的主要靶标,其结构与功能的复杂性决定了药物设计的成败。蛋白质的表示学习与性质预测是理解分子-靶点相互作用、发现候选药物的重要环节。蛋白质,尤其是酶,作为药物的主要作用靶点,其功能、结构与动力学性质直接影响药物的设计与效果。本专题通过两篇前沿研究工作展开讲解:*《Enzyme function prediction using contrastive learning》展示了如何利用对比学习从蛋白质序列中提取高质量的功能表征,实现对酶功能的精确预测;《CatPred》*则提出了一个整合性深度学习框架,用于体外酶动力学参数(如Km、kcat等)的预测,这对于建立药效模型与优化先导化合物至关重要。这些方法显著提升了蛋白质建模的准确性与泛化能力,为AI驱动的靶点发现、机制理解及候选药物筛选提供了强有力的支持。培训内容1: Nature Communication|体外酶动力学参数深度学习的综合框架《CatPred: a comprehensive framework for deep learning in vitro enzyme kinetic parameters》CatPred 提出了一种全面的深度学习框架,用于预测体外酶动力学参数(kcat、Km、Ki),以解决实验测定成本高、数据稀疏和泛化能力差的问题。该方法不仅提供了准确的预测,还引入了对预测不确定性的量化,支持对训练集外(out-of-distribution)酶序列的稳健预测。此外,作者还构建了新的标准化数据集(CatPred-DB),并对多种酶表示方法进行了系统比较。1.数据:CatPred 使用的数据集来自 BRENDA 和 SABIO-RK 数据库,作者构建了 CatPred-DB,包括:23197 条 kcat,41174 条 Km和11929 条 Ki 数据,每条记录都包含酶的氨基酸序列、AlphaFold 或 ESMFold 预测的结构、底物的 SMILES 表达式。数据经过清洗和标准化处理,去除缺失值和重复值,并对参数取对数转换以符合正态分布。2.模型:CatPred 采用模块化设计,酶和底物分别通过不同的神经网络模块进行表征学习,并采用 概率回归 输出(高斯分布形式的均值和方差),允许进行 不确定性估计(aleatoric + epistemic)。3.训练3.1.所有模型采用负对数似然损失函数(NLL)训练,以同时预测参数均值和不确定性。3.2.使用训练-验证-测试三分法(80%-10%-10%),并设立“训练集外”的测试子集用于泛化能力评估。3.3.为了评估不确定性,CatPred 使用 10个模型的集成,通过不同初始参数训练,以此量化 epistemic uncertainty。3.4.模型训练时考虑了不同相似性(序列identity<99%、80%、60%、40%)的测试集,体现其鲁棒性。培训内容2:Science|基于对比学习的蛋白质分类属性预测《Enzyme function prediction using contrastive learning》1.数据来源和处理: CLEAN模型的训练基于UniProt数据库中的高质量数据,该数据库收录了约1.9亿个蛋白质序列。CLEAN模型以氨基酸序列作为输入,输出按可能性排序的酶功能列表(以EC编号为例)。为了验证CLEAN的准确性和鲁棒性,作者进行了广泛的in silico实验,并将CLEAN应用于内部收集的未表征的卤酶数据库(共36个)进行EC编号注释,随后通过案例研究进行体外实验验证。2.模型架构和原理: CLEAN模型采用了对比学习框架,目标是学习一个酶的嵌入空间,其中欧几里得距离反映了功能相似性。嵌入是指蛋白质序列的数值表示,它由机器可读,同时保留了酶携带的重要特征和信息。在CLEAN的任务中,具有相同EC编号的氨基酸序列具有较小的欧几里得距离,而具有不同EC编号的序列则具有较大的距离。3.训练过程和细节:3.1.在训练过程中,CLEAN模型使用对比损失函数进行监督训练,通过优先选择与锚点(anchor)嵌入具有小欧几里得距离的负序列,以提高训练效率。3.2.模型使用语言模型ESM1b获得的蛋白质表示作为前馈神经网络的输入,输出层产生细化的、功能感知的输入蛋白质嵌入。3.3.预测时,通过计算查询序列与所有EC编号聚类中心之间的成对距离来预测输入蛋白质的EC编号。3.4.CLEAN还开发了两种方法来从输出排名中预测自信的EC编号:一种是贪婪方法,另一种是基于P值的方法。通过培训可以掌握的内容:1. 蛋白质表示学习的基本方法:理解蛋白质序列、结构信息如何被编码为适用于深度学习模型的向量表示,包括基于序列的预训练模型(如ESM、ProtBERT)和结构感知模型的原理与应用。2. 对比学习在生物功能预测中的应用:深入学习对比学习策略,掌握如何通过正负样本构建来提升蛋白质功能分类模型的判别能力。3. 酶动力学参数预测建模框架:学员将理解如何结合序列、结构及辅助特征,利用深度学习模型预测关键的酶学参数(如Km、kcat),并掌握模型架构设计与性能评估的思路。4. 评估与可解释性分析方法:学习如何分析模型预测结果,评估性能指标,并探索特征重要性等可解释性技术,帮助理解模型的决策依据。5. 应用于真实药物研发场景的思维框架:建立从蛋白质建模到下游任务(如药物筛选、作用机制分析)的系统性理解,增强将AI方法应用于实际生物医药问题的能力。第4天:顶刊复现专题3——基于深度学习的分子生成助力药物发现培训背景:分子生成是化学、生物学和材料科学等领域的关键技术,对于新药开发、新材料设计和化学反应预测具有重要意义。传统的分子生成方法依赖于专家知识和试错实验,耗时且成本高昂。随着人工智能技术的发展,特别是自然语言处理和扩散模型在分子生成中的应用,我们现在能够利用计算模型来加速这一过程。本课程将介绍从NLP到扩散模型的设计模式,这些模型能够理解和生成分子结构,从而提高分子设计的效率和准确性。通过本课程的学习,参与者将能够掌握分子生成的最新技术和方法,以及如何将这些技术应用于实际问题。培训内容1:Nature Communication|基于端到端的图生成框架的分子生成:《Retrosynthesis prediction using an end-to-end graph generative architecture for molecular graph editing》1.数据来源和处理:Graph2Edits模型使用了公开可用的基准数据集USPTO-50k,包含50016个反应,这些反应被正确地原子映射并分类为10种不同的反应类型。数据集被分为40k、5k、5k的反应用于训练、验证和测试集。2.模型架构和原理:Graph2Edits模型是一个端到端的图生成架构,基于图神经网络(GNN)预测产品图的编辑序列,并根据预测的编辑序列顺序生成中间体和最终反应物。该模型将半模板方法的两阶段过程(识别反应中心和完成合成子)合并为一锅学习,提高了在复杂反应中的适用性,并使预测结果更易于解释。模型的核心是图编码器和自回归模型,用于生成编辑序列,并应用这些编辑来推断中间体和反应物。3.训练过程和细节:3.1.Graph2Edits模型使用有向消息传递神经网络(D-MPNN)作为图编码器,以获取原子表示和全局图特征,并预测原子/键编辑和终止符号。3.2.模型训练使用教师强制策略,即使用真实的编辑序列作为模型输入。在每个编辑步骤中,模型会计算所有可能的编辑的概率,并选择最高分的k个编辑,将这些编辑应用于输入图以获得k个中间体。3.3.在生成过程中,如果达到最大步骤数或图表示指示终止,则生成分支将停止。3.4.最终,根据可能性对前k个编辑序列和图进行排名,收集为最终预测结果。培训内容2Nature Computational Science|基于等变扩散模型的分子生成网络《Structure-based drug design with equivariant diffusion models》1.简单介绍。这篇文献提出了一种基于结构的药物设计方法(SBDD),利用SE(3)-等变扩散模型(DiffSBDD)生成与蛋白质结合口条件匹配的新颖小分子配体。该方法通过将SBDD问题建模为三维条件生成任务,能够一次性生成所有原子位置,克服了传统自回归方法因顺序生成而丢失全局上下文的局限性。DiffSBDD不仅支持从头分子设计,还能通过属性优化、负向设计和分子局部修饰(inpainting)等多种任务灵活应用。2.数据总结。该研究使用了CrossDocked和Binding MOAD两个数据集进行训练和评估。2.1.CrossDocked数据集包含40,344个训练蛋白-配体对和130个测试对,验证集规模为246个,确保不同集合中的蛋白质来自不同的酶分类主类以避免过拟合。2.2.Binding MOAD数据集经过筛选后用于测试,分析限于所有方法均能生成样本的78个CrossDocked和119个Binding MOAD目标。此外,数据集处理涉及移除损坏条目,并通过Zenodo公开提供处理后的数据和采样分子,确保研究可重复性。3.模型总结。DiffSBDD是一个SE(3)-等变扩散模型,以蛋白质结合口为条件生成三维分子结构,采用3D图表示(原子坐标和类型),避免了传统方法中从密度图回推分子结构的复杂后处理。模型设计尊重三维空间的旋转和平通过培训可以掌握的内容:1.自然语言处理(NLP)在分子生成中的应用:掌握如何使用NLP技术来理解和生成分子结构。学习如何将自然语言描述转换为分子结构(SMILES字符串)。2.扩散模型在分子生成中的应用:理解扩散模型的基本原理及其在分子生成中的优势。学习如何使用扩散模型来优化分子生成过程。3.数据预处理和特征工程:学习如何处理和准备用于训练分子生成模型的数据集。掌握如何从原始数据中提取有用的特征以提高模型性能。4.模型架构和原理:深入理解MolT5,TGM-DLM和GraphEdits模型的架构和工作原理。学习如何设计和实现这些模型以处理复杂的分子生成任务。5.训练过程和细节:掌握模型训练的全过程,包括预训练和微调。学习如何调整模型参数和训练策略以优化性能。6.评估和验证:学习如何使用各种指标(如BLEU分数、Tanimoto相似性等)来评估生成的分子。掌握如何验证模型生成的分子的有效性和准确性。7.模型解释和可视化:学习如何解释模型的预测结果,以及如何使用可视化工具来理解分子生成过程。8.最新研究进展和技术趋势:了解分子生成领域的最新研究进展和技术趋势。学习如何将最新的研究成果应用于实际工作。第5天:顶刊复现专题4: 结合分子动力学的蛋白质-配体复合物相互作用动态预测培训背景:蛋白质-配体相互作用的预测是现代药物发现和生物工程领域的核心任务之一,其重要性不言而喻。在药物开发过程中,准确预测蛋白质与小分子配体的结合位点、三维结构以及亲和力,不仅能够揭示分子间相互作用的机制,还能显著加速候选药物的筛选与优化,降低研发成本和时间。传统实验方法如X射线晶体学和核磁共振虽然精确,但耗时长、成本高,且难以应对大规模筛选需求。而随着深度学习和人工智能技术的快速发展,计算方法在蛋白质-配体预测中展现出巨大潜力。研究内容1: Nature Communication|交互作用感知的蛋白质-配体对接和亲和力预测模型《Interformer: an interaction-aware model for protein-ligand docking and affinity prediction》1.简要介绍:本研究提出了一种名为Interformer的基于Graph-Transformer架构的统一模型,用于蛋白-配体对接和亲和力预测。针对现有深度学习模型忽略蛋白与配体原子间非共价相互作用建模的不足,Interformer引入了交互感知混合密度网络(MDN)来明确捕捉氢键和疏水相互作用,并结合负采样策略和伪Huber损失函数,通过对比学习优化相互作用分布,提升对接姿势的准确性和亲和力预测的鲁棒性。2.数据集:研究使用了PDBBind时间分割测试集(333个样本)评估对接准确性,Posebusters基准测试验证物理合理性,以及内部真实世界数据集测试泛化能力。训练数据来源于PDBBind晶体结构数据库。3.模型:Interformer基于Graph-Transformer架构,包括:(1) 图表示模块,将原子作为节点、邻近关系作为边;(2) 掩码自注意力(MSA)机制,通过Intra-Blocks和Inter-Blocks分别捕捉配体/蛋白内部及两者间的相互作用;(3) 交互感知MDN,融合四种高斯分布模拟常规力、疏水作用和氢键;(4) 边缘输出层整合节点和边特征预测能量;(5) 姿势评分和亲和力模块基于虚拟节点预测正确姿势和实验亲和力值。4.训练细节:训练分两阶段:首先基于晶体结构训练能量模型生成负样本,随后联合正负样本训练姿势评分和亲和力模型。采用负对数似然损失优化MDN,二元交叉熵损失优化姿势评分,伪Huber损失(σ=4)优化亲和力预测(单位IC50、Kd、KI,经负对数归一化)。蒙特卡洛采样生成候选姿势,研究内容2:Nature Communication|分子动力学驱动的蛋白质-配体复合物结构动态预测《DynamicBind: predicting ligand-specific protein-ligand complex structure with a deep equivariant generative model》1.简单介绍:本研究提出了一种名为DynamicBind的深度学习方法,用于预测配体特异性的蛋白-配体复合物结构。传统分子对接方法通常将蛋白视为刚性或仅部分柔性,难以处理蛋白的大尺度构象变化,而分子动力学模拟虽然能捕捉动态构象,但计算成本高昂。DynamicBind通过等变几何扩散网络构建平滑的能量景观,高效模拟蛋白从无配体(apo)状态到配体结合(holo)状态的构象转变,无需依赖holo结构或大量采样。2.数据集:研究基于PDBbind2020数据库(19,443个蛋白-配体复合物晶体结构),按时间划分:2019年前的数据用于训练和验证,2019年的数据用于测试。额外构建了Major Drug Targets (MDT)测试集(599对),聚焦激酶、GPCR等主要药物靶点,要求AlphaFold预测结构与晶体结构的pocket RMSD>2Å,确保测试难度。训练中通过AlphaFold预测结构与晶体结构插值生成蛋白部分的样本。3.模型:DynamicBind是一个基于图神经网络的等变生成模型,使用粗粒化表示(蛋白以Cα节点和侧链二面角表示,配体以重原子节点表示),输出包括蛋白和配体的平移、旋转、扭转角更新,以及结合亲和力和cLDDT置信度评分。模型通过学习从apo到holo的“morph-like”变换,优化能量景观,包含63.67百万参数。4.训练细节:训练在8块Nvidia A100 80GB GPU上进行5天,输入为添加morph变换的蛋白decoy构象和加高斯噪声的配体构象,目标是去噪操作。损失函数包括八项(配体和蛋白的平移、旋转、扭转等),通过Kabsch算法对齐apo和holo结构,结合扩散噪声调整构象过渡。推理时迭代20次更新初始结构。通过培训可以掌握的内容:1.蛋白质-配体复合物结构预测:学员将学习如何利用深度学习方法(如NeuralPLexer)从蛋白序列和配体分子图预测复合物的三维结构,理解多尺度几何建模和扩散过程在捕捉原子级分辨率结构及构象变化中的作用,并掌握其在盲对接和柔性结合位点恢复中的应用。2.对接姿势生成与优化:掌握基于Graph-Transformer架构和蒙特卡洛采样生成对接姿势的技术,学习如何通过姿势评分和对比学习(如伪Huber损失)优化姿势选择,提升对接准确性(如RMSD<2Å的成功率)。3.亲和力预测的计算方法:学员将了解如何从对接姿势预测实验亲和力值(如IC50、Kd、KI),掌握基于虚拟节点和对比学习的姿势敏感性训练策略,以提高亲和力预测的鲁棒性和实际应用价值。4.模型评估与基准测试:熟悉常用基准数据集(如PDBBind)和评价指标(如RMSD、lDDT-BS、TM-score)的使用,理解如何通过时间分割测试集和物理合理性检查评估模型的泛化能力和性能。5.实际药物设计的应用:通过案例分析(如Interformer筛选出高亲和力小分子),学习如何将这些预测技术应用于酶工程和药物发现,加速候选分子的筛选和优化过程。05机器学习代谢组学 内容可向下滑动第一天A1 代谢物及代谢组学的发展与应用(1) 代谢与生理过程;(2) 代谢与疾病;(3) 非靶向与靶向代谢组学;(4) 空间代谢组学与质谱成像(MSI);(5) 代谢组学与药物和生物标志物;(6) 代谢流与机制研究。A2 代谢通路及代谢数据库(1) 几种经典代谢通路简介;(2) 三大常见代谢物库:HMDB 、METLIN 和 KEGG;(3) 代谢组学原始数据库:Metabolomics Workbench 和 Metabolights. A3 参考资料推荐A4 代谢组学实验流程简介A5 色谱 、质谱硬件与原理解析(1) 色谱分析原理与构造;(2) 色谱仪和色谱柱的选择;(3) 色谱的流动相:梯度洗脱法;(4) 离子源、质量分析器与质量检测器解析;(5) 质谱分析原理及动画演示;(6) 色谱质谱联用技术(LC-MS);第二天B1 代谢物样本处理与抽提(1) 各种组织、血液和体液等样本的提取流程与注意事项;(2) 代谢物抽提流程与注意事项;(3) 样本及代谢物的运输与保存问题;B2 LC-MS 数据质控与搜库(1) LC-MS 实验过程中 QC 和 Blank 样本的设置方法;(2) LC-MS 上机过程的数据质控监测和分析;(3) 代谢组学上游分析原理——基于 Compound Discoverer 与 Xcms 软件;(4) Xcms 软件数据转换、提峰、峰对齐与搜库;B3 R 软件基础(1) R 和 Rstudio 的安装;(2) Rstudio 的界面配置;(3) R 中的基础运算和统计计算;(4) R 中的包:包,函数与参数的使用;(5) R 语言语法,数据类型与数据结构;(6) R 基础画图;B4 ggplot2(1) ggplot2 简介(2) ggplot2 的画图哲学;(3) ggplot2 的配色系统;(4) ggplot2 数据挖掘与作图实战;第三天机器学习C1 有监督式机器学习在代谢组学数据处理中的应用(1) 人工智能、机器学习、深度学习的关系;(2) 回归算法:从线性回归、Logistic 回归与 Cox 回归讲起;(3) PLS-DA 算法:PCA 降维后没有差异的数据还有救吗?(4) VIP score 的意义及选择;(5) 分类算法:决策树,随机森林和贝叶斯网络模型;C2 一组代谢组学数据的分类算法实现的 R 演练(1) 数据解读;(2) 演练与操作;C3 无监督式机器学习在代谢组学数据处理中的应用(1) 大数据处理中的降维;(2) PCA 分析作图;(3) 三种常见的聚类分析:K-means、层次分析与 SOM(4) 热图和 hcluster 图的 R 语言实现;C4 一组代谢组学数据的降维与聚类分析的 R 演练(1) 数据解析;(2) 演练与操作;第四天D1 在线代谢组分析网页 Metaboanalyst 操作(1) 用 R 将数据清洗成网页需要的格式;(2) 独立组、配对组和多组的数据格式问题;(3) Metaboanalyst 中的上游分析(原始数据峰提取、峰对齐与搜库)(4) Metaboanalyst 的 pipeline 以及参数设置和注意事项;(5) Metaboanalyst 的结果查看和导出;(6) Metaboanalyst 的数据编辑;(7) 全流程演练与操作。D2 代谢组学数据清洗与 R 语言进阶(1) 代谢组学中的 t、fold-change 和响应值;(2) 数据清洗流程;(3) R 语言 tidyverse;(4) 数据预处理:数据过滤与数据标准化(样本的 Normalization 和代谢物的 Scaling);(5) 代谢组学数据清洗演练;第五天E1 文献数据分析部分复现(1 篇)(1) 文献深度解读;(2) 实操:从原始数据下载到图片复现;(3) 学员实操。E2 机器学习与代谢组学顶刊解读(3 篇);(1) Signal Transduction and Targeted Therapy 一篇有关饥饿对不同脑区代谢组学影响变化的小鼠脑组织代谢图谱类的文献;(数据库型)(2) Cell 一篇代谢组学孕妇全程血液代谢组学分析得出对孕周和孕产期预测的代谢标志物的文献;(生物标志物型)(3) Nature 一篇对胰腺癌患者肠道菌群的代谢组学分析找到可以提高化疗效果的代谢物的文献。(机制研究型)06机器学习微生物组学 内容可向下滑动第一天1. 微生物学基础知识回顾2. 机器学习基本概念介绍a. 什么是机器学习b. 监督学习、无监督学习c. 常用机器学习模型介绍3. 混淆矩阵4. ROC 曲线第二天R 语言简介与实操1. R 语言概述2. R studio 软件与 R 包安装3. R 语言语法及数据类型4. 条件语句和循环Linux 实操1. Linux 操作系统2. Linux 操作系统的安装与设置3. 网络配置与服务进程管理4. Linux 的远程登录管理5. 常用的 Linux 命令6. 在 Linux 下获取基因数据7. Shell script 与 Vim 编辑器第三天微生物组常用分析方法(实操)1. 微生物丰度分析2. 转录组丰度分析3. 进化树分析4. 降维分析第四天机器学习在微生物组学中的应用案例分享1. 疾病预测应用:利用机器学习基于微生物组学数据预测疾病状态2. 肠道菌群研究:机器学习研究饮食对肠道微生物的影响第五天机器学习模型训练和分析(实操)1. 加载数据及数据归一化2. 构建训练模型(GLM, RF, SVM)3. 模型参数优化4. 模型错误率曲线绘制5. 混淆矩阵计算6. 重要特征筛选7. 模型验证,ROC 曲线绘制利用模型进行预测利用机器学习基于微生物组学数据预测宿主表型1. 加载数据2. 数据归一化3. OUT 特征处理4. 机器学习模型构建(RF, KNN, SVM, Lasso 等多种机器学习方法)5. 绘制 ROC 曲线,比较不同机器学习模型模型性能评估利用机器学习基于临床特征和肠道菌群预测疾病风险1. 加载数据2. 机器学习模型构建(RF, gbm, SVM 等等)3. 交叉验证4. 模型性能评估07深度学习基因组学内容可向下滑动第一天理论部分深度学习算法介绍1.有监督学习的神经网络算法1.1 全连接深度神经网络 DNN 在基因组学中的应用举例1.2 卷积神经网络 CNN 在基因组学中的应用举例1.3 循环神经网络 RNN 在基因组学中的应用举例1.4 图卷积神经网络 GCN 在基因组学中的应用举例2.无监督的神经网络算法2.1 自动编码器 AE 在基因组学中的应用举例2.2 生成对抗网络 GAN 在基因组学中的应用举例基因组常用深度学习框架1. 介绍深度学习工具包 tensorflow, keras,pytorch2. 在工具包中识别深度学习模型要素2.1.数据表示2.2.张量运算2.3.神经网络中的“层”2.4.由层构成的模型2.5.损失函数与优化器2.6.数据集分割2.7.过拟合与欠拟合基因组学基础1. 基因组数据库2. 表观基因组3. 转录基因组4. 蛋白质组5. 功能基因组实操内容1.Linux 操作系统1.1 常用的 Linux 命令1.2 Vim 编辑器1.3 基因组数据文件管理, 修改文件权限1.4 查看探索基因组区域2.Python 语言基础2.1.Python 包安装和环境搭建2.2.常见的数据结构和数据类型3. 安装深度学习工具包 tensorflow, keras,pytorch,在工具包中识别深度学习模型要素第二天理论部分1. 介绍 keras_dna 平台,搭建基因组学常用深度学习应用案例2. 深度学习模型 DeepG4 从 Chip-Seq 及 DnaseSeq 中识别基序特征 G4实操内容1.基因组数据处理搭建深度学习模型1.1 安装并使用 keras_dna 处理各种基因序列数据如 BED、 GFF、GTF、BIGWIG、BEDGRAPH、WIG 等1.2 使用 keras_dna 设计深度学习模型1.3 使用 keras_dna 分割训练集、测试集1.4 使用 keras_dna 选取特定染色体的基因序列等2.使用 keras_dna 平台复现 DeepG4 模型,从 Chip-Seq 中识别 G4 特征第三天理论部分深度学习在基因调控预测中的应用1. selene_sdk 预测 DNA 甲基化及转录调控因子等 DeepSEA2. 循环神经网络 RNN 从 RNA 序列中预测 pre-miRNA,dnnMiRPre实操内容复现卷积神经网络 CNN 识别基序特征 DeepG4、基因调控因子 DeepSEA,1. 安装 selene_sdk,复现 DeepSEA 预测 DNA 甲基化,非编码基因变异等基因调控因子2. 复现循环神经网络 RNN 工具 dnnMiRPre,从 RNA-Seq 中预测 pre-miRNA第四天理论部分深度学习在预测疾病表型及生物标志物上的应用1. 从高维基因表达数据中识别乳腺癌分型的自动编码机深度学习工具DeepType2. 深度学习在识别拷贝数变异 DeepCNV 模型实操内容1. 复现 DeepType,从 METABRIC 乳腺癌数据中区分乳腺癌亚型2. 解析 DeepType 中新的乳腺癌亚型的标志基因3. 复现 DeepCNV 利用 SNP 微阵列联合图像分析识别拷贝数变异第五天理论部分深度学习在预测药物反应机制上的应用1. 联合肿瘤基因标记及药物分子结构预测药物反应机制的深度学习工具 SWnet实操内容1. 预处理药物分子结构信息2. 计算药物相似性3. 在不同数据集上构建 self-attention SWnet4. 评估 self-attention SWnet5. 构建多任务的 SWnet6. 构建单层 SWnet7. 构建带权值层的 SWnet08深度学习质谱蛋白组学内容可向下滑动第一天蛋白质组学测序技术及数据库理论讲解1.蛋白质组学测序质谱技术2.介绍蛋白质组学数据库3.深度学习解析蛋白质组学模型介绍GPU服务器上机实操1.Linux操作系统1.1常用的Linux命令1.2 Vim编辑器1.3基因组数据文件管理, 修改文件权限1.4查看探索基因组区域2.Python语言基础2.1.Python包安装和环境搭建2.2.常见的数据结构和数据类型第二天深度学习识别质谱测序中蛋白质肽的理化性质上午 理论讲解1.深度学习模型预测色谱法保留时间及碎片离子浓度Prosit2.深度学习预测质谱测序中截面碰撞CCS工具DeepCollisionalCrossSection3.深度学习预测单细胞蛋白组学覆盖率DeepSCP模型下午 深度学习模型Python代码解析及GPU服务器上机实操1.复现深度学习模型预测色谱法保留时间及碎片离子浓度Prosit模型2.复现深度学习预测质谱测序中截面碰撞工具DeepCollisionalCrossSection3.复现深度学习预测单细胞蛋白组学覆盖率DeepSCP模型第三天深度学习识别肽及肽组装上午 理论讲解1.深度学习从宏蛋白组学中识别肽 DeepFilter模型2.深度学习从蛋白质数据库中识别肽DeepDIA模型3.深度学习实现肽组装DeepNovo 及DeepNovo-DIA模型下午 深度学习模型Python代码解析及GPU服务器上机实操1.复现深度学习从宏蛋白组学中识别肽 DeepFilter模型2.复现深度学习从蛋白质数据库识别肽DeepDIA模型复现深度学习实现肽组装DeepNovo及DeepNovo-DIA模型第四天深度学习识别翻译后修饰结合位点识别疾病及药物靶点上午 理论讲解 1.胶囊网络深度学习模型预测翻译后修饰结合位点模型CapsNet_PTM2.注意力机制深度学习预测MHC I 结合位点ACME模型 3.深度学习模型PUFFIN量化Peptide-MHC结合不确定性提升药物设计中高亲和力肽筛选4.深度学习模型预测癌症抗原ACP-MHCNN 模型下午 深度学习模型Python代码解析及GPU服务器上机实操1.复现胶囊网络深度学习模型预测翻译后修饰结合位点模型CapsNet_PTM2.复现注意力机制深度学习预测pan-specific MHC I 结合位点ACME模型 3.复现深度学习模型PUFFIN量化Peptide-MHC结合不确定性提升药物设计中高亲和力肽筛选4.复现深度学习模型预测癌症抗原ACP-MHCNN模型第五天深度学习识别蛋白质功能上午 理论讲解1.深度学习模型3D卷积网络预测蛋白质-蛋白质相互作用DeepRank2.深度学习模型量化蛋白质表达DLNetworkForProteinAbundance3.基于自然语言注意力机制深度学习模型预测蛋白质功能SPROF-GO4.深度学习模型PCfun 预测蛋白质复合物Gene Ontology功能下午 深度学习模型Python代码解析及GPU服务器上机实操1.复现深度学习模型3D卷积网络预测蛋白质-蛋白质相互作用DeepRank2.复现深度学习模型量化蛋白质表达DLNetworkForProteinAbundance3.复现基于自然语言注意力机制深度学习模型预测蛋白质功能SPROF-GO4.复现深度学习模型PCfun 预测蛋白质复合物Gene Ontology功能09CRISPR-Cas9基因编辑技术内容可向下滑动第一天绪论1.课程简介与学习目标2.基因编辑技术概述2.1 基因编辑的定义、核心原理与技术分类2.2 基因编辑与合成生物学的交叉3.技术应用领域全景图3.1基础研究3.2农业育种3.3疾病治疗3.4生物制造4.伦理与安全问题初探4.1 脱靶效应与基因驱动4.2 人类胚胎编辑的伦理边界第二天基因编辑技术发展简史1.1 ZFN1.2 TALENs 1.3 局限性CRISPR技术的革命性突破2.1 原核生物免疫机制的发现历程 2.2 Cas9系统在真核细胞的应用验证 2.3里程碑事件与诺贝尔奖解读2.4中国科学家在基因编辑领域的突出贡献第三天CRISPR常用工具与实操CRISPR-Cas系统1.1 CRISPR系统的起源与机制1.2 主要工具酶的特征与选择(实操)1.3 sgRNA的设计与优化(实操)1.4 CRISPR 筛选(CRISPR Screnning)1.5 CRISPR-Cas系统在模式生物中的应用1.6 CRISPR-Cas系统与CAR-T细胞治疗碱基编辑器(Base Editing)2.1 腺嘌呤碱基编辑器(ABE)2.2 脱氨酶的活性优化及对脱靶效应的控制2.3 胞嘧啶碱基编辑器(CBE)2.4 C-to-T编辑的特异性与效率平衡2.5 利用碱基编辑器构建动物模型2.6 单核苷酸突变矫正先导编辑(Prime Editing)3.1 逆转录酶的特点与选择3.2 pegRNA的设计与优化(实操)3.3 双pegRNA编辑系统3.4 PM359治疗慢性肉芽肿病CRISPR激活与抑制系统(CRISPRa/i)4.1 dCas94.2 dCas9与转录激活因子、转录抑制因子的融合4.3 CRISPRa在干细胞重编程中的应用4.4 CRISPRa/i研究癌症相关基因的功能网络其他基因编辑工具5.1 大片段DNA精准操纵工具5.2 Cre-loxP系统在模式动物中的应用第四天递送系统CRISPR递送系统概述1.1 CRISPR技术的基本原理和发展历程1.2 CRISPR递送系统的重要性和挑战病毒载体递送系统2.1 病毒载体的类型和特点(如腺相关病毒AAV)2.3病毒载体的构建和优化2.3 病毒载体在CRISPR递送中的应用和案例分析非病毒递送系统3.1 纳米颗粒(如脂质纳米颗粒LNP)的设计与应用3.2 电穿孔技术的原理和应用3.3 非病毒递送系统的优缺点分析植物病毒递送系统4.1 植物-弹状病毒在CRISPR递送中的应用4.2 植物病毒递送系统的优化和挑战第五天CRISPR应用CRISPR在基础研究中的应用1.1 CRISPR在基因功能研究中的应用1.2 CRISPR在疾病模型创建中的应用1.3 CRISPR在基因调控研究中的应用CRISPR在遗传病治疗中的应用2.1 遗传病数据库的建立和应用2.2 CRISPR治疗遗传病的案例分析2.3FDA批准的CRISPR疗法介绍(镰状细胞贫血、杜氏肌肉营养不良、癌症的免疫疗法)小结与展望基本内容小结+当前技术瓶颈分析1.1 递送效率与组织靶向性难题1.2复杂性状的多基因协同编辑新兴技术发展方向2.1 DNA 聚合酶编辑器2.2 CRISPR 引导的重组酶和转座子2.3 表观基因组编辑2.4 RNA编辑2.5 AI与基因编辑新型CRISPR工具临床转化路线图3.1 体内编辑与体外编辑的产业化路径3.2 基因编辑疗法监管体系的国际比较基因编辑的道德考量和安全性10AI智慧医疗影像技术内容可向下滑动第一天智慧医疗中的图像处理与分析概论1、阐述智慧医疗定义,回溯其发展历程,剖析当下智慧医疗多元应用场景,如远程诊断、智能辅助决策等,展望未来在精准医疗、个性化治疗方面的潜力。2、深入讲解医疗图像在智慧医疗体系里的重要性,从辅助诊断、治疗规划到疗效评估全流程解析,明确课程知识、技能双目标,梳理学习计划,强调结合实际案例的实践导向。医疗图像的基本类型和获取方法1、详细科普医疗图像主要类型,简要介绍X射线成像原理、CT断层扫描机制、MRI磁共振成像特色、超声成像的声波利用,每种类型搭配实际应用场景,如X射线在骨折检测,MRI在脑部病变诊断的应用。第二天图像处理和分析的基本技术和算法1、实操演示图像预处理技术,展示去噪算法应用、增强视觉效果手段、对比度调整技巧的实际效果,如使用OpenCV进行高斯模糊去噪、中值滤波去除椒盐噪声,直方图均衡化增强对比度,每种方法结合具体医学图像案例,如去除X光图像中的噪声,增强CT图像的对比度,展示实际的OpenCV代码示例。2、简要讲解边缘检测原理,对比常用边缘检测算子如Sobel、Canny算子的效果,解读形态学操作如腐蚀、膨胀在图像处理中的功效,展示如何用OpenCV实现这些操作。3、拆解图像分割技术,简要介绍阈值分割法、区域分割策略、边缘分割原理,结合医学图像案例,如用阈值分割法分离MRI图像中的脑组织,用区域生长算法标记肿瘤区域,用边缘分割勾勒器官轮廓,展示如何用Python和OpenCV实现这些分割方法,提供相关的代码示例和实际的图像处理结果。深度学习在医疗图像分析中的应用1、从基础概念入手,介绍深度学习框架,简要讲解CNN卷积神经网络架构,说明卷积层、池化层、全连接层的作用,结合医学图像识别中的应用案例,讲解如何用深度学习模型实现图像分类、目标检测和分割。2、结合实际案例,剖析深度学习在图像分类、目标检测、分割中的应用流程,实操演示常用深度学习模型YOLOv5在医疗图像里的调用与优化,提供实际的模型定义和训练代码示例,如用YOLOv5检测X光图像中的肺结节,展示如何配置模型参数、加载预训练模型、训练和优化模型,实现医学图像的目标检测任务。第三天Transformer 技术在医疗图像分析中的应用1、讲解Transformer基本原理,简要介绍架构里自注意力机制运作逻辑,说明自注意力机制如何捕捉图像中不同区域之间的长距离依赖关系,提升模型对上下文的理解。2、分享Transformer在医疗图像分析中的前沿应用案例,如基于Transformer的模型在医学图像分割、分类和目标检测中的应用,讲解如何利用Transformer架构的全局特征提取能力提升诊断准确性,以脑肿瘤分割为例,演示Transformer如何更准确地识别肿瘤边界。3、指导学员动手实践,使用Transformer模型进行医疗图像分析任务,提供相关的代码示例和开源项目链接,如用Vision Transformer(ViT)实现医学图像分类,展示如何将医学图像分割成patches并输入到Transformer中,实现端到端的分类任务。多模态数据融合在医疗图像分析中的应用1、阐述多模态数据概念,列举常见类型,如影像、文本报告、生理数据融合,讲解多模态数据在医学诊断中的互补优势,如影像数据提供组织结构信息,文本报告包含症状和病史,生理数据反映身体机能状态。 2、介绍多模态数据融合方法,如特征级融合、决策级融合技术,结合实际案例,如融合CT图像和临床指标对疾病分期,演示如何用Python实现特征级融合,将不同模态的数据拼接成多维特征向量,输入到机器学习模型中进行分类或回归任务。 3、分析多模态融合在提升医疗图像分析准确性上的作用,展示实际的多模态数据融合代码示例,如用Keras或PyTorch实现多个神经网络分支分别处理不同模态的数据,最后在决策层进行融合。第四天医疗图像分析在疾病诊断和治疗中的实际案例1、以肺结节筛查诊断为切入点,展示图像分析辅助医生精准定位、良恶性判断流程,通过超声、CT、MRI 等检查手段对病情进行综合评估和诊断,提供实际的肺结节检测代码示例和数据集,讲解如何用深度学习模型在胸部CT图像中自动检测肺结节,并根据结节特征判断良恶性。2、延伸至肿瘤识别与分割,讲解如何利用图像处理技术勾勒肿瘤边界,探讨心血管疾病诊断评估,通过血管影像分析病情,展示实际的心血管疾病诊断代码示例和数据集,如用U-Net模型分割脑部MRI图像中的肿瘤区域,用血管成像技术分析冠状动脉狭窄程度。图像处理工具与实践与深度学习模型的训练1、介绍常用图像处理工具OpenCV、MATLAB 功能模块,实操演示图像处理算法实现与优化技巧,提供实际的图像处理代码示例和优化方法,如用OpenCV实现医学图像的量化、特征分析等功能,用MATLAB进行快速傅里叶变换和小波变换。2、指导学员使用OpenCV完成图像预处理和分析任务,上手深度学习框架训练医疗图像分类模型,掌握模型调参方法,提供实际的模型训练代码示例和调参技巧,如在ResNet模型的基础上进行微调,优化学习率、批量大小、正则化参数等。第五天医疗图像分析项目设计与实施1、引导学员进行项目需求分析,规划项目整体架构,涵盖数据收集、处理、模型选择到结果评估全流程,强调项目的临床实用性和快速部署,鼓励学员结合实际工作中的问题设计项目主题。2、提供结构化的课程学习计划,围绕主题进行拓展学习和实践,推荐相关书籍、学术论文、在线课程和实操项目资源,辅助学习者深化理解课程知识,提升专业技能。学习目标AI蛋白质设计课程旨在为学生提供深度学习与蛋白质设计领域的全面知识。通过讲授深度学习的基本概念和前沿技术,学生将理解深度学习在生物信息学特别是蛋白质设计中的具体应用。学生将了解如何使用主流深度学习框架PyTorch进行模型构建与优化,并通过实践操作掌握蛋白质结构预测、蛋白质功能预测和分子对接等关键技术。课程将介绍AlphaFold等先进模型,并探讨其在药物发现中的重要性。同时通过多肽设计、逆向中心法则等专题,学生将全面了解从功能推导结构和从零开始设计蛋白质的策略。合成生物与基因电路设计通过理论与实践结合,掌握合成生物学基础、基因电路设计、代谢途径优化、基因编辑技术及数学建模,培养学员在合成生物领域的创新能力和系统思维,为未来研究与应用奠定基础。CADD计算机辅助药物设计掌握包括PDB数据库、靶点蛋白、蛋白质-配体、蛋白-配体小分子、蛋白-配体结构、notepad的介绍和使用、分子对接、蛋白-配体对接、虚拟筛选、蛋白-蛋白对接、蛋白-多糖分子对接、蛋白-水合对接、Linux安装、gromacs分 子动力学全程实操、溶剂化分子动力学模拟AIDD人工智能药物设计与发现本课程让学员了解药物发现的前沿背景,学习人工智能领域的各类常见算法,熟悉工具包的安装与使用,掌握一定的算法编程能力,能够运用计算机方法研究药物相关问题。通过大量的案例讲解和实践操作,具备一定的AIDD模型构建和数据分析能力机器学习代谢组学熟悉代谢组学和机器学习相关硬件和软件;熟悉代谢组学从样本处理到数据分析的全流程;能复现至少1篇CNS或子刊级别的代谢组学文章图片。机器学习微生物组学课程将涵盖机器学习技术在微生物数据分析中的应用,包括基因组序列分析、基因调控网络构建和多组学数据整合等,并带领学员们深度使用R语言,Python语言实地操作演示。深度学习基因组学课程深入学习与了解深度学习基本框架与逻辑,同时掌握基本的生物信息学软件(Linux、R、python等)的使用,让学员能更好的应对基因组数据,挖掘出超越已有知识的新知识。而构建好的深度学习模型去探求新的研究思路和寻找新的潜在生物学机制,更好的服务于自身的科学研究和探索的过程中。深度学习质谱蛋白组学课程通过对这些深度学习在蛋白组学中的应用案例进行深度讲解和实操,让学员能够掌握深度学习分析蛋白组学数据流程,系统学习深度学习及蛋白组学理论知识及熟悉软件代码实操,熟练掌握这些前沿的分析工具的使用以及研究创新深度学习算法解决生物学及临床疾病问题与需求。CRISPR-Cas9基因编辑技术课程从全局出发,由浅入深,课程通过基础入门+应用案例实操演练的方式,从最初的原理讲解到最后的应用实战,学完本课程你将掌握基因编辑技术的相关原理及其应用,此外可以学到基因编辑系统的优化策略,可以学到如何操作常用的生物学软件。能够快速运用到自己的科研项目和课题上。AI智慧医疗影像技术课程掌握智慧医疗图像处理与分析技术,包括图像预处理、深度学习应用、多模态数据融合及项目实践,培养学员在医疗影像分析中的技术应用能力和问题解决能力讲师介绍AI蛋白设计授课老师来自北大人工智能学院,作为核心成员参与过清华某初创公司大语言模型项目,主导过北大长沙研究院的核心项目智能体的开发,此外和包括百度,字节等多个公司和北大的合作中作为主要负责人,累计参与项目金额超过千万。目前发过多篇ACL等计算机顶会和sci论文。合成生物与基因电路设计两位授课老师均来自清华大学,干湿结合分别引领本课程的实验设计和建模分析,研究方向涉及植物生物学、合成生物学与生物信息学。在对应领域中科院一区有多篇产出,同时曾作为队长和评委多次参加过合成生物学(iGEM)顶级赛事,曾获得全球十佳项目(TOP10)和多个单项奖及提名。CADD计算机辅助药物设计杨教授在计算机/人工智能辅助药物设计(CADD/AIDD)领域具有多年的研究经验,熟悉分子对接和虚拟筛选,分子动力学模拟、先导物优化、人工智能等计算工具和方法,在J. Med. Chem, J. Chem. Inf. Model,J. Phys. Chem. B, Frontiers in Pharmacology 等国际主流期刊发表学术论文20余篇,主持或参与国家自然科学基金、河南省重点研发、企业横向项目十余项。AIDD人工智能药物设计与发现AIDD授课老师曹老师,有十余年的计算机算法研究和程序设计经验。研究方向涉及生物信息学,深度学习,药物合成路径设计,药物不良反应等。发明专利5项,参与国家重点科研项目4项,发表SCI高水平论文10篇,包括BMC Bioinformatics, Journal of Biomedical Informatics, International Journal of Molecular Sciences等知名期刊。机器学习代谢组学讲老师来自985高校神经科学博士,主要利用代谢组学、转录组学和分子生物学等技术研究神经内科慢性病的发病机制和生物标志物。擅长高效液相色谱-质谱联用(LC-MS)技术进行非靶向和靶向代谢组学从样本制备到数据分析的全流程研究,以及多组学大数据的生物信息学整合分析。5年内在J Clin Invest, EBioMedicine, Cell Death Dis, Cell Death Discov, Nanotoxicology等杂志发表SCI论文10篇。机器学习微生物组学主讲老师来自清华大学,研究方向包括生物信息学、机器学习与微生物基因组学,大模型与蛋白质定向进化等。同时他在图神经网络和疾病药物靶向等知识图谱技术方面有丰富的经验,带领并指导多次团队在国际基因工程竞赛(iGEM)中获得国际金牌,并一作发表了多篇一区高水平SCI论文。深度学习基因组学主讲老师刘老师,生物信息学PI,有十余年的测序数据分析经验。研究领域涉及人工智能、自然语言处理、功能基因组学、转录组学、miRNA及靶基因网络分析,单细胞测序数据分析,基因调控网络时序分析,蛋白质互作网络分析,多组学联合分析等。主持省自然科学基金等项目4项,发表SCI论文23篇,论著一部深度学习质谱蛋白组学主讲老师刘老师,生物信息学博士,从事生物信息及医学人工智能研究 15 年,开发过数个生物信息学工具,发表 SCI 论文 20 余篇,其中人工智能算法文章近 10 篇,编著医学数据分析实用教材一部,研究致力于医学人工智能在复杂疾病诊疗中的应用。CRISPR-Cas9基因编辑技术主讲老师均来自清华大学、浙江大学、西湖大学等国内顶尖高校,他们在基因编辑及相关领域拥有深厚的学术背景和丰富的研究经验。在博士期间深入研究基因编辑技术,发表了多篇高水平论文(包括子刊和多篇一区文章)并有各类系统扎实的设计实操经验,助力学员们在基因编辑领域取得更大的进步和发展。AI智慧医疗影像技术授课老师来自清华大学, 拥有生物医学信息学与信息工程处理等丰富的经验。在对应领域中科院一区有多篇产出,也有过多篇CCF-A类同领域,如ICLR的产出和审稿经验,熟悉计算机领域算法和生物医学图像的加工和处理方式。授课时间01AI蛋白质设计2025.06.14全天(9点-11点半-下午1点半-5点)2025.06.15全天(9点-11点半-下午1点半-5点)2025.06.16晚上授课(19:00-22:00)2025.06.18晚上授课(19:00-22:00)2025.06.19晚上授课(19:00-22:00)2025.06.20晚上授课(19:00-22:00)2025.06.21全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放02合成生物与基因电路设计2025.06.14全天(9点-11点半-下午1点半-5点)2025.06.15全天(9点-11点半-下午1点半-5点)2025.06.17晚上授课(晚19:00—晚22:00)2025.06.18晚上授课(晚19:00—晚22:00)2025.06.19晚上授课(晚19:00—晚22:00)2025.06.20晚上授课(晚19:00—晚22:00)2025.06.21全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放03CADD计算机辅助药物设计2025.06.10晚上授课(19:00-22:00)2025.06.11晚上授课(19:00-22:00)2025.06.12晚上授课(19:00-22:00)2025.06.13晚上授课(19:00-22:00)2025.06.15全天(9点-11点半-下午1点半-5点)2005.06.16晚上授课(19:00-22:00)2005.06.17晚上授课(19:00-22:00)2005.06.18晚上授课(19:00-22:00)2005.06.19晚上授课(19:00-22:00)2025.06.21全天(9点-11点半-下午1点半-5点)2025.06.22全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放04AIDD人工智能药物设计与发现2025.06.14全天(9点-11点半-下午1点半-5点)2025.06.15全天(9点-11点半-下午1点半-5点)2025.06.17晚上授课(晚19:00—晚22:00)2025.06.18晚上授课(晚19:00—晚22:00)2025.06.19晚上授课(晚19:00—晚22:00)2025.06.20晚上授课(晚19:00—晚22:00)2025.06.22全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放05机器学习代谢组学2025.06.21全天(9点-11点半-下午1点半-5点)2025.06.22全天(9点-11点半-下午1点半-5点)2025.06.23晚上授课(晚19:00—晚22:00)2025.06.24晚上授课(晚19:00—晚22:00)2025.06.25晚上授课(晚19:00—晚22:00)2025.06.26晚上授课(晚19:00—晚22:00)2025.06.28全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放06机器学习微生物组学2025.06.22全天(9点-11点半-下午1点半-5点)2025.06.23晚上授课(晚19:00—晚22:00)2025.06.24晚上授课(晚19:00—晚22:00)2025.06.25晚上授课(晚19:00—晚22:00)2025.06.26晚上授课(晚19:00—晚22:00)2025.06.28全天(9点-11点半-下午1点半-5点)2025.06.29全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放07深度学习基因组学2025.06.14全天(9点-11点半-下午1点半-5点)2025.06.15全天(9点-11点半-下午1点半-5点)2025.06.17晚上授课(晚19:00—晚22:00)2025.06.18晚上授课(晚19:00—晚22:00)2025.06.19晚上授课(晚19:00—晚22:00)2025.06.20晚上授课(晚19:00—晚22:00)2025.06.21全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放08深度学习质谱蛋白组学2025.06.22全天(9点-11点半-下午1点半-5点)2025.06.23晚上授课(晚19:00—晚22:00)2025.06.24晚上授课(晚19:00—晚22:00)2025.06.25晚上授课(晚19:00—晚22:00)2025.06.26晚上授课(晚19:00—晚22:00)2025.06.28全天(9点-11点半-下午1点半-5点)2025.06.29全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放09CRISPR-Cas9基因编辑技术2025.06.22全天(9点-11点半-下午1点半-5点)2025.06.23晚上授课(晚19:00—晚22:00)2025.06.24晚上授课(晚19:00—晚22:00)2025.06.25晚上授课(晚19:00—晚22:00)2025.06.26晚上授课(晚19:00—晚22:00)2025.06.28全天(9点-11点半-下午1点半-5点)2025.06.29全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放10AI智慧医疗影像技术2025.06.14全天(9点-11点半-下午1点半-5点)2025.06.15全天(9点-11点半-下午1点半-5点)2025.06.17晚上授课(晚19:00—晚22:00)2025.06.18晚上授课(晚19:00—晚22:00)2025.06.19晚上授课(晚19:00—晚22:00)2025.06.20晚上授课(晚19:00—晚22:00)2025.06.21全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放报名费用及福利1.AI蛋白质设计 公费价:每人每班¥6380 元 自费价:每人每班¥5880元2.CADD计算机辅助药物设计3.AIDD人工智能药物发现4.机器学习代谢组学5.深度学习在基因组学中的应用6.合成生物与基因电路设计7.深度学习在质谱蛋白组学中的应用8.机器学习微生物组学9.CRISPR-Cas9基因编辑技术公费价:每人每班¥5880自费价:每人每班¥548010.AI智慧医疗影像技术公费价:每人每班¥4680自费价:每人每班¥4380优惠一报二赠一:10880(赠送课程任选)优惠二报三赠一:13880(赠送课程任选)报四赠二:18880元(赠送课程任选)特惠三全报:25880两年内可免费参加本公司举办的任何课程(不限次数及课程,包括之后的新开课)限时福利:报名成功后转发朋友圈或转发50人以上群聊即可获得300元现金红包(只限前15名)特惠福利:报一送一可额外送的回放(包含全套课程回放和课件资料ppt)机器学习生物医学单细胞测序与空间转录组深度学习宏基因组学蛋白晶体结构解析报名费用可开具正规报销发票及提供相关缴费证明、邀请函,可提前开具报销发票、文件用于报销 。报名缴费后即可获得全套预习资料供大家课前准备证书:参加培训并通过考试的学员,可以申请获得工业和信息化部工业文化发展中心颁发的“工业强国建设素质素养提升尚工行动”岗位能力适应评测证书。该证书可在中心官网查询,可作为能力评价,考核和任职的重要依据。评测证书查询网址:www.miit-icdc.org(自愿申请,须另行缴纳考试费500元/人)SIMPLICITY官方联系人联系人:苏老师,报名咨询电话:13783571273(同V)往期学员好评截图:
基因疗法核酸药物
2025-04-26
·动脉网
近日,量子AI初创公司SandboxAQ宣布获得4.5亿美元E轮融资,本轮融资吸引了谷歌、英伟达以及法国巴黎银行等行业巨头的投资,总融资额9.5亿美元,公司估值达到57.5亿美元(约合人民币419.3亿元)。SandboxAQ是2022年从谷歌母公司Alphabet拆分出来的公司,利用量子计算技术开发人工智能模型,已成功研发了大型定量模型(Large Quantitative Models,LQMs)。其量子模型服务于生命科学、金融服务、导航等多个领域,可用于处理大量数值数据集、执行复杂计算和进行统计分析,正在不断打破技术边界,具有巨大的潜在应用价值。在AI蓬勃发展的时代浪潮下,SandboxAQ究竟凭借何种独特优势,不断吸引着投资者的目光?又在生命科学领域,采用了怎样的创新路径来实现技术突破,为医疗行业不断注入新动能?我们来一一解码。01谷歌前CEO创业,以量子AI破解药物研发难题SandboxAQ是一家量子AI企业,被看作是量子技术领域的先行者。自成立以来,SandboxAQ公司始终专注于量子AI的解决方案研发,致力于推动量子技术在商业领域的应用。量子计算凭借量子叠加、量子纠缠等特性,使其在处理复杂计算任务时具备指数级的加速能力。量子计算与AI的融合(即“量子AI”),能显著提升人工智能的学习能力和计算效率。SandboxAQ由前谷歌首席执行官Eric Schmidt和Alphabet前员工Jack Hidary共同创办。董事长Eric Schmidt担任过谷歌首席执行官和董事长,并在谷歌母公司Alphabet担任过要职,在科技、商业和政策方面拥有深厚影响力。首席执行官Jack Hidary拥有哥伦比亚大学神经科学背景,深耕功能性脑成像和人工神经网络研究,是量子计算领域的专家,其著作《量子计算:一种应用方法》更是成为本科和博士课程的重要教科书。此外,他还是一位连续创业者,成功带领EarthWeb/Dice公司上市,并联合创立VistaResearch,在企业运营与创新领域成绩斐然。在科技与数据智能领域,“沙盒”(Sandbox)是一种隔离且受限的运行环境,专门用于安全测试与技术创新。沙盒因此成为技术创新的摇篮,新软件、算法和系统得以在此环境中反复验证与迭代。SandboxAQ的命名正源于这一理念。公司以“沙盒”精神为指引,专注于量子计算模型的研发,旨在为技术创新搭建稳定、可靠的试验平台。就医疗领域而言,SandboxAQ便展现出强大的创新能力。利用量子与AI深度融合,其量子模型LQMs正在不断加速新药研发进程,攻克药物研发难题;同时,基于LQMs所研发的心磁图仪(MCG)设备正在解锁心脏病诊断领域的创新应用,提高诊断准度与精度。02运用定量大模型LQMs,快速筛选并生成药物分子传统药物发现过程中,往往需要采用虚拟筛选的方式对数以十亿计的化合物进行处理。这一过程依赖庞大的预枚举数据集,并需要运用多种方法对化合物进行评估和过滤,最终仅有数十到数百个化合物进入实验测试与验证阶段,不仅计算成本高昂,而且无法确定数据之外是否存在更优的配体,存在明显局限性。为了攻克药物研发难题,SandboxAQ成立生物制药分子模拟部门AQBioSim,依托定量大模型LQMs,为药物发现与开发全生命周期提供提质增效的解决方案。SandboxAQ推出的生成式AI应用程序IDOLpro,则有效破解了这一难题。作为SandboxAQ基于云的模拟软件包,IDOLpro是一种将公共数据与基于物理的模拟相结合的LQM。它借助AWS基础设施,将扩散模型与多目标优化相结合,以此指导、设计药物分子生成。从技术原理来看,LQMs在药物分子筛选与生成环节发挥关键作用。它通过对海量化学数据和分子结构库进行训练,能够模拟分子间的相互作用与动态行为,快速锁定符合预期作用机制(MoA)的目标分子,并在几分钟内优化3D药物分子,以AI算法创造定制特性分子,进一步提升药物研发的准确性与效率。在实际测试中,通过使用crosstocked和Binding MOAD等评估蛋白质-配体对接方法性能的基准数据集进行评估,IDOLpro所生成的模型在结合亲和力上达到了行业领先方法的3.4倍,首次产生的化合物性能甚至优于实验验证的分子。另外,在药物研发的筛选探索中,SandboxAQ基于LQMs研发出创新性的AQFEP(Advanced Quantum Free Energy Perturbation,先进量子自由能微扰)技术,也为药物筛选带来新突破。传统药物筛选方法开发并不完善。相对自由能微扰(RFEP)方法虽然广泛应用于结合亲和力预测,但计算成本高,且需要依赖已知活性的结构类似物,限制了筛选范围和灵活性。而绝对自由能微扰(AFEP)方法理论上能更准确地鉴定命中率,但处理速度迟缓,难以满足虚拟筛选的快速对接与评分的实际需求。AQFEP技术有效克服了上述弊端,它将主动学习与严格的基于物理的评分功能相结合,充分汲取RFEP和AFEP的优势,兼具高效性与准确性。该技术无需参考分子,在集成工作流程中,AQFEP能够率先“解锁”分子,实现对大型化学库的高效筛选,大幅提升虚拟筛选的命中率。在神经退行性疾病和肿瘤等领域的筛选工作中,AQFEP于命中识别和线索优化环节均表现卓越,具有极高的应用价值。目前,SandboxAQ已与两家顶级学术研究机构建立起了合作伙伴关系,并扩大了与大型生物制药公司的合作关系,包括阿斯利康、赛诺菲和加州大学旧金山分校,以确定新的生物标记物并优化在研药物的临床开发。03推出开创性心磁图仪设备,提高诊断准确性与全面性近年来,心血管疾病持续威胁全球人类健康,据世界卫生组织统计,每年约有1800万人死于心血管疾病,占全球总死亡人数的30%以上。在心血管疾病诊断领域,传统心电图(EKG)已沿用150余年,作为监测心脏电活动的重要工具,在诊断心脏节律异常方面发挥着关键作用。但其检测的电脉冲信号容易受到身体组织干扰,影响诊断准确性。仅在美国,每年就有800万急诊患者因胸痛就诊,而通过心电图等标准工具,成功确诊的患者不足5%。针对这一难题,SandboxAQ推出了CardiAQ这一开创性的心磁图仪 (MCG) 设备。该设备通过使用高性能传感器检测心脏磁信号,并利用大型定量模型(LQM)有效消除电磁干扰,避免了传统EKG的局限性,能够更有效地捕捉心脏电磁活动的细节,帮助检测异常模式和潜在的心脏疾病状态,为医生提供更全面的诊断依据。在实际运用中,CardiAQ具有便捷性和高效性,无需专用空间,也无需冷却或屏蔽,只需几分钟就能完成测量,非常适合在病床旁即时检测。这种非接触式的创新诊断方式,凭借高性能传感器与先进 AI 技术的深度融合,改变了心脏疾病的诊断模式,不仅能为患者带来更及时、更准确的诊疗服务,更有望在全球范围内提升心脏疾病的防治水平,拯救患者生命。目前,为了测试CardiAQ的功能,两家知名研究医院使用该设备进行临床研究,同时,SandboxAQ还与Mayo Clinic达成技术合作,将共同探索人工智能驱动的磁描记技术(MCG)在心脏病诊断领域的创新应用,致力于改善心脏病诊断水平。04写在最后近年来,AI制药市场呈现出蓬勃发展的态势,规模不断扩大,增长势头强劲。据Research And Markets数据显示,2022年全球AI制药市场规模达到10.4亿美元,预计到2026年,这一数字将近30亿美元,年平均复合增长率高达30%。到2032年,全球AI药物研发市场规模预计更是会突破200亿美元,未来前景十分广阔。越来越多的企业开始涉足AI制药领域,既有晶泰科技、英矽智能等专注于AI制药的初创企业,也有恒瑞医药、石药集团等传统药企通过战略合作、股权投资等方式积极拥抱AI技术,加速创新药物研发进程。在此竞争格局下,SandboxAQ凭借对LQMs定量大模型的深度应用,在药物研发领域展现出独特优势与发展潜力。凭借前瞻性的战略思维、前沿的技术手段、前卫的创始团队,SandboxAQ不断获得一众投资者的青睐,走出了属于自己的独特发展道路。更广泛意义上来看,SandboxAQ的崛起恰好契合了量子AI研究在当下全球科技中的火热——中、美多国均积极投入,百度、谷歌、微软等科技巨头也纷纷入局。量子AI的特性,比如叠加态和纠缠态,确实能解决传统AI难以应对的问题。比如,量子AI可以在优化问题中同时处理多种可能性,快速解决传统计算需要耗费数年甚至数十年的复杂任务,潜力场景涉及物流、交通、芯片制造、能源分配、药物研发等。量子AI的潜力是一个技术性的切入点,但意义不止于此。它可能会改变我们对智能、创造力和未来的理解。在更遥远的未来,量子AI能够开创出一个全新的AI制药时代吗?我们拭目以待。如果您想对接文章中提到的项目,或您的项目想被动脉网报道,或者发布融资新闻,请与我们联系;也可加入动脉网行业社群,结交更多志同道合的好友。近期推荐声明:动脉网所刊载内容之知识产权为动脉网及相关权利人专属所有或持有。未经许可,禁止进行转载、摘编、复制及建立镜像等任何使用。动脉网,未来医疗服务平台
高管变更
2025-04-25
一、2025 年 3 月 28 日,诺奖得主、蛋白质设计先驱 David Baker 教授在 Nature Methods 期刊发表了题为:Atomic context-conditioned protein sequence design using LigandMPNN 的研究论文。该研究开发了一种新型深度学习方法——LigandMPNN,该方法明确地对生物分子系统中的所有非蛋白质成分进行了建模,预计 LigandMPNN 将在设计新的结合蛋白、传感器和酶方面得到广泛应用。蛋白质的从头设计,能够创造出具有新功能的新型蛋白质,例如催化作用、与 DNA、小分子和金属的结合以及蛋白质间的相互作用。二、在生物信息学领域,传统纯生信研究依赖大量实验,成本高、周期长,限制了研究进展。而随着人工智能技术飞速发展,机器学习算法展现出强大的数据处理与分析能力,为纯生信研究开辟新路径。将机器学习引入纯生信,有望突破实验瓶颈,高效挖掘生物数据价值,助力科研人员更深入理解生命奥秘,在疾病筛查、药物研发等方面发挥关键作用,这一融合趋势成为科研界关注焦点。shop十大热门专题助你发顶刊01AI蛋白质设计02合成生物与基因电路设计03CADD计算机辅助药物设计04AIDD人工智能药物设计与发现05机器学习代谢组学06机器学习微生物组学07深度学习基因组学08深度学习质谱蛋白组学09CRISPR-Cas9基因编辑技术10AI智慧医疗影像技术shop以下为具体内容介绍01AI蛋白质设计内容可向下滑动第一天蛋白质结构及分子动力学基础a)蛋白质设计概述b)蛋白质结构基础 pdb文件格式详解数据库详解c)分子动力学基础分子-蛋白对接蛋白-蛋白对接可视化软件pymol使用AMBER分子动力学模拟高斯加速分子动力学模拟模拟轨迹分析MM-GB/PBSA方法计算结合自由能分子动力学模拟在酶改造中的应用第二天a)Python、Numpy基础b)经典模型线性及非线性映射:线性回归、逻辑回归聚类:K-近邻聚类核方法:支持向量机树方法:决策树、随机森林神经网络:多层感知机scikit-learn 基本调用实操c)深度学习深度神经网络DNN卷积神经网络CNN循环神经网络RNNd)前沿架构及复现TransformerBERTGPTViT第三天蛋白结构预测及其下游应用a)蛋白结构预测背景介绍b)AlphafoldAF和AF2的差异与创新AF3的差异与创新Alphafold使用详解c)基于Alphafold的下游应用基于AF2的蛋白-蛋白对接/蛋白-多肽对接利用AF2做多构象预测和功能发现基于AF2的环肽设计d)trrosetta幻想设计e)基于ProteinMPNN的蛋白质设计MPNN模型简介设计流程方法比较第四天蛋白质的AI生成模型扩散模型a)RFdiffusion实现蛋白结构设计蛋白Binder生成蛋白骨架设计单体蛋白从头生成多聚体蛋白从头生成RFdiffusion在酶改造中的应用b)Chroma的基本构架与实现蛋白构象空间全空间采样c)ProteinGenerator蛋白质骨架与序列设计与rfdiffusion的异同蛋白语言模型a)ProGEN与ProGEN2模型构架讲解与基于结构方法的比较性能与改进 b)ESM-foldESM-fold的基本架构ESMfold网络讲解与alphafold方法的对比ESM-fold的性能评估c) 基于大语言模型的下游应用孤儿蛋白结构的预测大型蛋白复合物结构预测基于蛋白语言模型的酶设计第五天基于深度学习的蛋白质挖掘与改造a)基于alphafold的酶挖掘b)通过预测蛋白复合物发现生命过程新机制c)基于大语言模型的新酶挖掘d)深度学习从基因组挖掘全新蛋白e)综合:结合多种深度学习与计算工具的蛋白改造流程02合成生物与基因电路设计内容可向下滑动第一天合成生物学基础概念与应用领域剖析1.深度解读合成生物学精准定义,系统阐述其涵盖研究内容,沿着发展历程脉络回溯,展望未来趋势,全方位扫描应用领域。2.通过详实案例分析,将合成生物学在医药、农业、工业等实际场景的应用具象化,让学员感知其变革力量。生物元件功能精讲与标准化设计准则1.聚焦生物元件,如启动子精准调控转录起始、终止子界定转录终点、RBS 驱动核糖体结合开启翻译。2.结合 BioBrick 元件设计范式与应用实例,传授生物元件模块化设计黄金法则,助力学员掌握构建标准化生物模块的精髓。第二天基因线路逻辑架构搭建与实例演练1.开启基因线路逻辑世界大门,详细拆解与、或、非基本逻辑门运作原理及设计技巧。2.引入 iGEM 竞赛中经典逻辑门线路应用案例,手把手指导学员实践操作,使其能独立构思简单基因线路并洞悉其功能。复合元件整合与电路深度设计以前两天所学生物元件、基因线路知识为基石,引导学员梳理合成生物学核心玩法。亲手设计、搭建、分析经典电路,实现知识融会贯通,掌握复杂电路中各模块协同运作奥秘。第三天代谢途径精细构建与优化策略1.深入讲解代谢途径搭建步骤,剖析定向进化、代谢工程等优化策略。2.以青蒿素生物合成途径优化为典型案例,传授从理论到实践的代谢途径设计方法,助力学员提升生物合成效率。基因组合成与编辑技术前沿1.系统讲解基因组合成底层原理、多元方法,深度剖析 CRISPR/Cas9 等基因组编辑技术原理。2.结合海量案例,呈现基因组合成与编辑技术在定制生物、精准医疗等前沿领域的实际应用。第四天底盘生物特性解析与应用1.引入底盘生物概念,剖析常见底盘生物(如大肠杆菌、酵母菌等)特性。2.讲解其在基因线路搭载、基因表达中的关键作用,结合实例探讨如何依据项目需求选择适配底盘生物,实现高效基因操作。合成生物系统搭建与优化实践1.全方位讲解合成生物系统定义、分类,通过案例剖析系统构建流程,传授优化策略。2.组织实践操作,让学员亲身体验合成生物系统的构建与优化全过程,学会依据反馈调控系统参数。第五天合成生物学数学建模与性能分析1.开启生物系统数学建模之旅,介绍常微分方程、逻辑模型等常用建模方法。2.结合基因线路动力学模拟案例,指导学员运用数学模型精准分析生物系统性能,为系统设计与优化提供量化支撑。合成生物学全景总结与未来瞻望1.回溯课程所学知识,通过多领域案例分析,展现合成生物学应用广度与深度。2.组织学员热烈讨论未来发展趋势、潜在影响,着重介绍伦理、生物安全与生物安保等关键议题,培养学员全面视角。03CADD计算机辅助药物设计内容可向下滑动第一天1.CADD药物设计概论1.1计算机辅助药物设计的发源和基本概念1.2计算机辅助药物设计中常用的计算方法2. 基于结构的药物设计方法1.1 基于结构的药物设计方法概论1.2 蛋白质数据库的介绍:uniprot、PDB、AlphafldDB3. 高质量画图软件pymol 使用详解1.1 蛋白小分子相互作用、蛋白-蛋白相互作用绘制等1..2 蛋白及小分子表面图、静电势表示等第二天1.蛋白质结构处理方法1.1蛋白质结构预测方法理论介绍及使用1.2蛋白质缺失残基处理、残基质子态确定、单点突变等处理方法2.小分子结构处理方法1.1常用的小分子数据库介绍:zinc,chembl、pubchem、神农Alpha等1.2Chemdraw的使用及小分子性质计算第三天1. 蛋白-小分子对接的基本流程与技巧1.1 分子对接的原理1.2 基于autudock和autodock vina的分子对接步骤详解1.3 对接结果的分析与挑选技巧第四天1.特殊的分子对接的基本流程与技巧1.1蛋白-小分子柔性对接步骤详解1.2蛋白-蛋白/DNA/RNA,小分子-小分子对接方法详解2. 基于分子对接的虚拟筛选详解1.1 虚拟筛选的流程与处理1.2 虚拟筛选的结果分析与技巧第五天1.Openbael的使用1.1小分子不同的表示形式、文件格式介绍1.2Openbabel的文件格式转换、分子结构生成介绍2.ADMET介绍和反向找靶介绍3.QSAR模型介绍及操作1.1 QSAR模型的基本原理1.2 3DQSAR--COMFA和COMSIA模型的构建与结果分析第六天1.Linux系统操作1.1Linux系统介绍1.2Linux常用命令操作2.分子动力学模拟介绍1.1分子动力学模拟理论介绍1.2常用软件和一般流程介绍第七天1.MD实践1.1溶剂化下蛋白质分子动力学模拟1.2 溶剂化下蛋白质-配体的分子动力学模拟1.3 MD结果分析2. 答疑:1.1 课程知识答疑1.2 学员实际课题思路答疑部分模型案例图片04AIDD人工智能药物设计与发现内容可向下滑动第1天:环境搭建与深度学习基本知识讲解1.AIDD概述:从CADD到AIDD2.软件安装与环境搭建(1)anaconda(2)vscode(3)环境变量的配置(4)切换pip和conda镜像源(5)虚拟环境的创建3.RDKIT工具包的使用(1)基于RDKit的分子读写(2)基于RDKit的分子绘制(3)基于RDKit的分子指纹与分子描述符(4)基于RDKit的化合物相似性与子结构4.药物综合数据库的获取方法(1)基于requests的基本爬虫操作(2)小分子数据库PubChem数据获取(pubchempy / requests)(3)蛋白质数据库PDB、UniProt数据获取5.深度学习辅助药物设计(1)神经网络基本概念与sklearn工具包介绍(2)图神经网络与消息传递机制基本知识(3)Transformer模型基本知识:分词、位置编码、注意力机制、编码器、解码器、预训练-微调框架、huggingface 生态介绍(4)模型的评估与验证:准确率、精确率、召回率、F1分数、ROC曲线、AUC计算,平均绝对误差、均方差、R2分数、可释方差分数,交叉验证等第2天:顶刊复现专题1——分子与生化反应的表示学习与性质预测助力药物发现培训背景:在人工智能辅助药物发现(AIDD)中,分子与生化反应的表示学习与性质预测是整个研究流程的基石。分子的结构决定其功能,如何将复杂的分子结构和生化反应过程有效地表示为计算模型能够理解的形式,是实现高效预测和优化的前提。通过构建合理的分子表示(如图神经网络、SMILES编码、指纹等),我们可以让AI模型捕捉关键的化学特征,进而用于预测分子的物理化学性质、生物活性、毒性等,为后续的虚拟筛选、分子生成与反应设计提供可靠基础。因此,本专题不仅奠定了AIDD中建模与预测能力的核心能力框架,也为整个药物发现过程中的智能决策打下了坚实基础。培训内容1:Nature Machine Intelligence|基于注意力的神经网络在化学反应空间映射中的应用《Mapping the space of chemical reactions using attention-based neural networks》1.数据集1.1.Pistachio数据集:包含260万化学反应,来自专利数据,涵盖792个反应类别。数据经过去重和有效性过滤(使用RDKit)。1.2.USPTO 1k TPL数据集:基于USPTO专利数据,包含44.5万反应,通过原子映射和模板提取生成1,000个反应模板类别。1.3.Schneider 50k数据集:公开数据集,包含5万反应,50个类别,用于与传统指纹方法对比。2.模型。研究对比了两种Transformer架构:2.1.BERT分类器:基于编码器的模型,通过掩码语言建模预训练后,在分类任务上微调,使用[CLS]标记的嵌入作为反应指纹(rxnfp)。2.2.Seq2Seq模型:编码器-解码器结构,将分类任务分解为超类、类别和具体反应的层级预测。两者均采用简化版BERT(隐藏层256维),输入为未标注的SMILES序列,无需反应物-试剂区分或原子映射。3.训练。模型训练分为两步:3.1.预训练:BERT通过掩码SMILES令牌预测任务进行自监督学习,学习反应通用表示。3.2.微调:在分类任务上优化模型,使用交叉熵损失,学习率2×10⁻⁵,序列长度512。评估采用混淆熵(CEN)和马修斯相关系数(MCC)以处理数据不平衡。培训内容2:TOP期刊|基于深度学习的生化反应产量预测《Prediction of chemical reaction yields using deep learning》 1.数据。研究使用了三类数据:1.1.Buchwald-Hartwig HTE数据集:包含3955个Pd催化C-N偶联反应,涵盖15种卤化物、4种配体、3种碱和23种添加剂组合,产率通过统一实验测量,数据质量高。 1.2.Suzuki-Miyaura HTE数据集:包含5760个反应,涉及15对亲电/亲核试剂、12种配体、8种碱和4种溶剂的组合,产率分布均匀。 1.3.USPTO专利数据集:从公开专利中提取,包含不同规模(克级与亚克级)的反应产率,数据噪声大且分布不一致,需通过邻近反应产率平滑处理以提升模型表现。2.模型。核心模型基于预训练的rxnfp(反应指纹)BERT架构,新增回归层构成Yield-BERT。输入为标准化反应SMILES,通过自注意力机制捕捉反应中心及关键试剂的上下文信息。模型无需手工特征(如DFT计算描述符),直接端到端预测产率。实验表明,其性能优于传统方法(如随机森林和分子指纹拼接),尤其在HTE数据上接近化学描述符的预测水平,且参数鲁棒性高(超参数调整影响小)。3.训练。训练分为两步:3.1.预训练:BERT通过掩码语言任务学习SMILES的通用表示。 3.2.微调:采用简单Transformers库和PyTorch框架,以MSE损失优化回归层,学习率(2×10⁻⁵)和dropout率(0.1–0.8)为主要调参对象。HTE数据采用随机/时间划分验证,USPTO数据通过邻近反应产率平滑缓解噪声影响。小样本实验(5%训练数据)显示模型能快速筛选高产反应,指导合成优化。培训内容3:TOP期刊|基于T5Chem模型的生化反应表示学习与性质预测: 《Unified Deep Learning Model for Multitask Reaction Predictions with Explanation》1.数据来源和处理。通过自监督预训练与PubChem分子数据集进行训练,以实现对四种不同类型的化学反应预测任务的优异性能。模型处理包括反应类型分类、正向反应预测、单步逆合成和反应产率预测。2.模型架构和原理。T5Chem模型是基于自然语言处理中的“Text-to-Text Transfer Transformer”(T5)框架开发的统一深度学习模型,该模型通过适应T5框架来处理多种化学反应预测任务。T5Chem模型包含编码器-解码器结构,并根据任务类型引入了任务特定的提示和不同的输出层,如分子生成头、分类头和回归头,以处理序列到序列的任务、反应类型分类和产品产率预测。3.训练过程和细节。3.1.T5Chem模型首先在PubChem的97 million分子上进行自监督预训练,使用BERT类似的“masked language modeling”目标。3.2.在预训练阶段,源序列中的tokens被随机掩蔽,模型的目标是预测被掩蔽的正确的tokens。3.3.预训练完成后,模型在下游的监督任务中进行微调,使用不同的任务特定提示和输出层。3.4.模型在测试阶段通过生成分子token by token的方式进行预测,直到生成“句子结束标记”或达到最大预测长度。通过培训可以掌握的内容:1.分子与化学反应的表示方法。学习如何将分子和化学反应编码为机器可处理的格式,如SMILES(Simplified Molecular-Input Line-Entry System)和反应SMILES。理解分子指纹(如Morgan指纹)和反应指纹(如rxnfp)的构建方式,以及它们在化学信息学中的应用。掌握Transformer架构(如BERT)如何用于化学反应的特征提取,并生成具有化学意义的向量表示。2.深度学习在化学反应预测中的应用。了解如何利用序列到序列(Seq2Seq)模型和BERT进行化学反应分类(如反应类型识别)和产率预测。学习如何通过自注意力机制分析化学反应的关键部分(如反应中心、试剂影响),提高模型的可解释性。掌握如何利用预训练+微调策略,使模型在少量标注数据下仍能取得良好性能。3.数据驱动的化学研究范式。认识不同数据来源(如高通量实验HTE、专利数据USPTO)的特点及其对模型训练的影响。学习如何处理数据噪声,并通过数据平滑、邻近分析等方法优化模型表现。了解小样本学习在化学中的应用,例如如何用5%-10%的训练数据筛选高产率反应,指导实验优化。第3天:顶刊复现专题2——蛋白质的表示学习与性质预测助力药物发现培训背景:在AIDD中,蛋白质是药物作用的主要靶标,其结构与功能的复杂性决定了药物设计的成败。蛋白质的表示学习与性质预测是理解分子-靶点相互作用、发现候选药物的重要环节。蛋白质,尤其是酶,作为药物的主要作用靶点,其功能、结构与动力学性质直接影响药物的设计与效果。本专题通过两篇前沿研究工作展开讲解:*《Enzyme function prediction using contrastive learning》展示了如何利用对比学习从蛋白质序列中提取高质量的功能表征,实现对酶功能的精确预测;《CatPred》*则提出了一个整合性深度学习框架,用于体外酶动力学参数(如Km、kcat等)的预测,这对于建立药效模型与优化先导化合物至关重要。这些方法显著提升了蛋白质建模的准确性与泛化能力,为AI驱动的靶点发现、机制理解及候选药物筛选提供了强有力的支持。培训内容1: Nature Communication|体外酶动力学参数深度学习的综合框架《CatPred: a comprehensive framework for deep learning in vitro enzyme kinetic parameters》CatPred 提出了一种全面的深度学习框架,用于预测体外酶动力学参数(kcat、Km、Ki),以解决实验测定成本高、数据稀疏和泛化能力差的问题。该方法不仅提供了准确的预测,还引入了对预测不确定性的量化,支持对训练集外(out-of-distribution)酶序列的稳健预测。此外,作者还构建了新的标准化数据集(CatPred-DB),并对多种酶表示方法进行了系统比较。1.数据:CatPred 使用的数据集来自 BRENDA 和 SABIO-RK 数据库,作者构建了 CatPred-DB,包括:23197 条 kcat,41174 条 Km和11929 条 Ki 数据,每条记录都包含酶的氨基酸序列、AlphaFold 或 ESMFold 预测的结构、底物的 SMILES 表达式。数据经过清洗和标准化处理,去除缺失值和重复值,并对参数取对数转换以符合正态分布。2.模型:CatPred 采用模块化设计,酶和底物分别通过不同的神经网络模块进行表征学习,并采用 概率回归 输出(高斯分布形式的均值和方差),允许进行 不确定性估计(aleatoric + epistemic)。3.训练3.1.所有模型采用负对数似然损失函数(NLL)训练,以同时预测参数均值和不确定性。3.2.使用训练-验证-测试三分法(80%-10%-10%),并设立“训练集外”的测试子集用于泛化能力评估。3.3.为了评估不确定性,CatPred 使用 10个模型的集成,通过不同初始参数训练,以此量化 epistemic uncertainty。3.4.模型训练时考虑了不同相似性(序列identity<99%、80%、60%、40%)的测试集,体现其鲁棒性。培训内容2:Science|基于对比学习的蛋白质分类属性预测《Enzyme function prediction using contrastive learning》1.数据来源和处理: CLEAN模型的训练基于UniProt数据库中的高质量数据,该数据库收录了约1.9亿个蛋白质序列。CLEAN模型以氨基酸序列作为输入,输出按可能性排序的酶功能列表(以EC编号为例)。为了验证CLEAN的准确性和鲁棒性,作者进行了广泛的in silico实验,并将CLEAN应用于内部收集的未表征的卤酶数据库(共36个)进行EC编号注释,随后通过案例研究进行体外实验验证。2.模型架构和原理: CLEAN模型采用了对比学习框架,目标是学习一个酶的嵌入空间,其中欧几里得距离反映了功能相似性。嵌入是指蛋白质序列的数值表示,它由机器可读,同时保留了酶携带的重要特征和信息。在CLEAN的任务中,具有相同EC编号的氨基酸序列具有较小的欧几里得距离,而具有不同EC编号的序列则具有较大的距离。3.训练过程和细节:3.1.在训练过程中,CLEAN模型使用对比损失函数进行监督训练,通过优先选择与锚点(anchor)嵌入具有小欧几里得距离的负序列,以提高训练效率。3.2.模型使用语言模型ESM1b获得的蛋白质表示作为前馈神经网络的输入,输出层产生细化的、功能感知的输入蛋白质嵌入。3.3.预测时,通过计算查询序列与所有EC编号聚类中心之间的成对距离来预测输入蛋白质的EC编号。3.4.CLEAN还开发了两种方法来从输出排名中预测自信的EC编号:一种是贪婪方法,另一种是基于P值的方法。通过培训可以掌握的内容:1. 蛋白质表示学习的基本方法:理解蛋白质序列、结构信息如何被编码为适用于深度学习模型的向量表示,包括基于序列的预训练模型(如ESM、ProtBERT)和结构感知模型的原理与应用。2. 对比学习在生物功能预测中的应用:深入学习对比学习策略,掌握如何通过正负样本构建来提升蛋白质功能分类模型的判别能力。3. 酶动力学参数预测建模框架:学员将理解如何结合序列、结构及辅助特征,利用深度学习模型预测关键的酶学参数(如Km、kcat),并掌握模型架构设计与性能评估的思路。4. 评估与可解释性分析方法:学习如何分析模型预测结果,评估性能指标,并探索特征重要性等可解释性技术,帮助理解模型的决策依据。5. 应用于真实药物研发场景的思维框架:建立从蛋白质建模到下游任务(如药物筛选、作用机制分析)的系统性理解,增强将AI方法应用于实际生物医药问题的能力。第4天:顶刊复现专题3——基于深度学习的分子生成助力药物发现培训背景:分子生成是化学、生物学和材料科学等领域的关键技术,对于新药开发、新材料设计和化学反应预测具有重要意义。传统的分子生成方法依赖于专家知识和试错实验,耗时且成本高昂。随着人工智能技术的发展,特别是自然语言处理和扩散模型在分子生成中的应用,我们现在能够利用计算模型来加速这一过程。本课程将介绍从NLP到扩散模型的设计模式,这些模型能够理解和生成分子结构,从而提高分子设计的效率和准确性。通过本课程的学习,参与者将能够掌握分子生成的最新技术和方法,以及如何将这些技术应用于实际问题。培训内容1:Nature Communication|基于端到端的图生成框架的分子生成:《Retrosynthesis prediction using an end-to-end graph generative architecture for molecular graph editing》1.数据来源和处理:Graph2Edits模型使用了公开可用的基准数据集USPTO-50k,包含50016个反应,这些反应被正确地原子映射并分类为10种不同的反应类型。数据集被分为40k、5k、5k的反应用于训练、验证和测试集。2.模型架构和原理:Graph2Edits模型是一个端到端的图生成架构,基于图神经网络(GNN)预测产品图的编辑序列,并根据预测的编辑序列顺序生成中间体和最终反应物。该模型将半模板方法的两阶段过程(识别反应中心和完成合成子)合并为一锅学习,提高了在复杂反应中的适用性,并使预测结果更易于解释。模型的核心是图编码器和自回归模型,用于生成编辑序列,并应用这些编辑来推断中间体和反应物。3.训练过程和细节:3.1.Graph2Edits模型使用有向消息传递神经网络(D-MPNN)作为图编码器,以获取原子表示和全局图特征,并预测原子/键编辑和终止符号。3.2.模型训练使用教师强制策略,即使用真实的编辑序列作为模型输入。在每个编辑步骤中,模型会计算所有可能的编辑的概率,并选择最高分的k个编辑,将这些编辑应用于输入图以获得k个中间体。3.3.在生成过程中,如果达到最大步骤数或图表示指示终止,则生成分支将停止。3.4.最终,根据可能性对前k个编辑序列和图进行排名,收集为最终预测结果。培训内容2Nature Computational Science|基于等变扩散模型的分子生成网络《Structure-based drug design with equivariant diffusion models》1.简单介绍。这篇文献提出了一种基于结构的药物设计方法(SBDD),利用SE(3)-等变扩散模型(DiffSBDD)生成与蛋白质结合口条件匹配的新颖小分子配体。该方法通过将SBDD问题建模为三维条件生成任务,能够一次性生成所有原子位置,克服了传统自回归方法因顺序生成而丢失全局上下文的局限性。DiffSBDD不仅支持从头分子设计,还能通过属性优化、负向设计和分子局部修饰(inpainting)等多种任务灵活应用。2.数据总结。该研究使用了CrossDocked和Binding MOAD两个数据集进行训练和评估。2.1.CrossDocked数据集包含40,344个训练蛋白-配体对和130个测试对,验证集规模为246个,确保不同集合中的蛋白质来自不同的酶分类主类以避免过拟合。2.2.Binding MOAD数据集经过筛选后用于测试,分析限于所有方法均能生成样本的78个CrossDocked和119个Binding MOAD目标。此外,数据集处理涉及移除损坏条目,并通过Zenodo公开提供处理后的数据和采样分子,确保研究可重复性。3.模型总结。DiffSBDD是一个SE(3)-等变扩散模型,以蛋白质结合口为条件生成三维分子结构,采用3D图表示(原子坐标和类型),避免了传统方法中从密度图回推分子结构的复杂后处理。模型设计尊重三维空间的旋转和平通过培训可以掌握的内容:1.自然语言处理(NLP)在分子生成中的应用:掌握如何使用NLP技术来理解和生成分子结构。学习如何将自然语言描述转换为分子结构(SMILES字符串)。2.扩散模型在分子生成中的应用:理解扩散模型的基本原理及其在分子生成中的优势。学习如何使用扩散模型来优化分子生成过程。3.数据预处理和特征工程:学习如何处理和准备用于训练分子生成模型的数据集。掌握如何从原始数据中提取有用的特征以提高模型性能。4.模型架构和原理:深入理解MolT5,TGM-DLM和GraphEdits模型的架构和工作原理。学习如何设计和实现这些模型以处理复杂的分子生成任务。5.训练过程和细节:掌握模型训练的全过程,包括预训练和微调。学习如何调整模型参数和训练策略以优化性能。6.评估和验证:学习如何使用各种指标(如BLEU分数、Tanimoto相似性等)来评估生成的分子。掌握如何验证模型生成的分子的有效性和准确性。7.模型解释和可视化:学习如何解释模型的预测结果,以及如何使用可视化工具来理解分子生成过程。8.最新研究进展和技术趋势:了解分子生成领域的最新研究进展和技术趋势。学习如何将最新的研究成果应用于实际工作。第5天:顶刊复现专题4: 结合分子动力学的蛋白质-配体复合物相互作用动态预测培训背景:蛋白质-配体相互作用的预测是现代药物发现和生物工程领域的核心任务之一,其重要性不言而喻。在药物开发过程中,准确预测蛋白质与小分子配体的结合位点、三维结构以及亲和力,不仅能够揭示分子间相互作用的机制,还能显著加速候选药物的筛选与优化,降低研发成本和时间。传统实验方法如X射线晶体学和核磁共振虽然精确,但耗时长、成本高,且难以应对大规模筛选需求。而随着深度学习和人工智能技术的快速发展,计算方法在蛋白质-配体预测中展现出巨大潜力。研究内容1: Nature Communication|交互作用感知的蛋白质-配体对接和亲和力预测模型《Interformer: an interaction-aware model for protein-ligand docking and affinity prediction》1.简要介绍:本研究提出了一种名为Interformer的基于Graph-Transformer架构的统一模型,用于蛋白-配体对接和亲和力预测。针对现有深度学习模型忽略蛋白与配体原子间非共价相互作用建模的不足,Interformer引入了交互感知混合密度网络(MDN)来明确捕捉氢键和疏水相互作用,并结合负采样策略和伪Huber损失函数,通过对比学习优化相互作用分布,提升对接姿势的准确性和亲和力预测的鲁棒性。2.数据集:研究使用了PDBBind时间分割测试集(333个样本)评估对接准确性,Posebusters基准测试验证物理合理性,以及内部真实世界数据集测试泛化能力。训练数据来源于PDBBind晶体结构数据库。3.模型:Interformer基于Graph-Transformer架构,包括:(1) 图表示模块,将原子作为节点、邻近关系作为边;(2) 掩码自注意力(MSA)机制,通过Intra-Blocks和Inter-Blocks分别捕捉配体/蛋白内部及两者间的相互作用;(3) 交互感知MDN,融合四种高斯分布模拟常规力、疏水作用和氢键;(4) 边缘输出层整合节点和边特征预测能量;(5) 姿势评分和亲和力模块基于虚拟节点预测正确姿势和实验亲和力值。4.训练细节:训练分两阶段:首先基于晶体结构训练能量模型生成负样本,随后联合正负样本训练姿势评分和亲和力模型。采用负对数似然损失优化MDN,二元交叉熵损失优化姿势评分,伪Huber损失(σ=4)优化亲和力预测(单位IC50、Kd、KI,经负对数归一化)。蒙特卡洛采样生成候选姿势,研究内容2:Nature Communication|分子动力学驱动的蛋白质-配体复合物结构动态预测《DynamicBind: predicting ligand-specific protein-ligand complex structure with a deep equivariant generative model》1.简单介绍:本研究提出了一种名为DynamicBind的深度学习方法,用于预测配体特异性的蛋白-配体复合物结构。传统分子对接方法通常将蛋白视为刚性或仅部分柔性,难以处理蛋白的大尺度构象变化,而分子动力学模拟虽然能捕捉动态构象,但计算成本高昂。DynamicBind通过等变几何扩散网络构建平滑的能量景观,高效模拟蛋白从无配体(apo)状态到配体结合(holo)状态的构象转变,无需依赖holo结构或大量采样。2.数据集:研究基于PDBbind2020数据库(19,443个蛋白-配体复合物晶体结构),按时间划分:2019年前的数据用于训练和验证,2019年的数据用于测试。额外构建了Major Drug Targets (MDT)测试集(599对),聚焦激酶、GPCR等主要药物靶点,要求AlphaFold预测结构与晶体结构的pocket RMSD>2Å,确保测试难度。训练中通过AlphaFold预测结构与晶体结构插值生成蛋白部分的样本。3.模型:DynamicBind是一个基于图神经网络的等变生成模型,使用粗粒化表示(蛋白以Cα节点和侧链二面角表示,配体以重原子节点表示),输出包括蛋白和配体的平移、旋转、扭转角更新,以及结合亲和力和cLDDT置信度评分。模型通过学习从apo到holo的“morph-like”变换,优化能量景观,包含63.67百万参数。4.训练细节:训练在8块Nvidia A100 80GB GPU上进行5天,输入为添加morph变换的蛋白decoy构象和加高斯噪声的配体构象,目标是去噪操作。损失函数包括八项(配体和蛋白的平移、旋转、扭转等),通过Kabsch算法对齐apo和holo结构,结合扩散噪声调整构象过渡。推理时迭代20次更新初始结构。通过培训可以掌握的内容:1.蛋白质-配体复合物结构预测:学员将学习如何利用深度学习方法(如NeuralPLexer)从蛋白序列和配体分子图预测复合物的三维结构,理解多尺度几何建模和扩散过程在捕捉原子级分辨率结构及构象变化中的作用,并掌握其在盲对接和柔性结合位点恢复中的应用。2.对接姿势生成与优化:掌握基于Graph-Transformer架构和蒙特卡洛采样生成对接姿势的技术,学习如何通过姿势评分和对比学习(如伪Huber损失)优化姿势选择,提升对接准确性(如RMSD<2Å的成功率)。3.亲和力预测的计算方法:学员将了解如何从对接姿势预测实验亲和力值(如IC50、Kd、KI),掌握基于虚拟节点和对比学习的姿势敏感性训练策略,以提高亲和力预测的鲁棒性和实际应用价值。4.模型评估与基准测试:熟悉常用基准数据集(如PDBBind)和评价指标(如RMSD、lDDT-BS、TM-score)的使用,理解如何通过时间分割测试集和物理合理性检查评估模型的泛化能力和性能。5.实际药物设计的应用:通过案例分析(如Interformer筛选出高亲和力小分子),学习如何将这些预测技术应用于酶工程和药物发现,加速候选分子的筛选和优化过程。05机器学习代谢组学 内容可向下滑动第一天A1 代谢物及代谢组学的发展与应用(1) 代谢与生理过程;(2) 代谢与疾病;(3) 非靶向与靶向代谢组学;(4) 空间代谢组学与质谱成像(MSI);(5) 代谢组学与药物和生物标志物;(6) 代谢流与机制研究。A2 代谢通路及代谢数据库(1) 几种经典代谢通路简介;(2) 三大常见代谢物库:HMDB 、METLIN 和 KEGG;(3) 代谢组学原始数据库:Metabolomics Workbench 和 Metabolights. A3 参考资料推荐A4 代谢组学实验流程简介A5 色谱 、质谱硬件与原理解析(1) 色谱分析原理与构造;(2) 色谱仪和色谱柱的选择;(3) 色谱的流动相:梯度洗脱法;(4) 离子源、质量分析器与质量检测器解析;(5) 质谱分析原理及动画演示;(6) 色谱质谱联用技术(LC-MS);第二天B1 代谢物样本处理与抽提(1) 各种组织、血液和体液等样本的提取流程与注意事项;(2) 代谢物抽提流程与注意事项;(3) 样本及代谢物的运输与保存问题;B2 LC-MS 数据质控与搜库(1) LC-MS 实验过程中 QC 和 Blank 样本的设置方法;(2) LC-MS 上机过程的数据质控监测和分析;(3) 代谢组学上游分析原理——基于 Compound Discoverer 与 Xcms 软件;(4) Xcms 软件数据转换、提峰、峰对齐与搜库;B3 R 软件基础(1) R 和 Rstudio 的安装;(2) Rstudio 的界面配置;(3) R 中的基础运算和统计计算;(4) R 中的包:包,函数与参数的使用;(5) R 语言语法,数据类型与数据结构;(6) R 基础画图;B4 ggplot2(1) ggplot2 简介(2) ggplot2 的画图哲学;(3) ggplot2 的配色系统;(4) ggplot2 数据挖掘与作图实战;第三天机器学习C1 有监督式机器学习在代谢组学数据处理中的应用(1) 人工智能、机器学习、深度学习的关系;(2) 回归算法:从线性回归、Logistic 回归与 Cox 回归讲起;(3) PLS-DA 算法:PCA 降维后没有差异的数据还有救吗?(4) VIP score 的意义及选择;(5) 分类算法:决策树,随机森林和贝叶斯网络模型;C2 一组代谢组学数据的分类算法实现的 R 演练(1) 数据解读;(2) 演练与操作;C3 无监督式机器学习在代谢组学数据处理中的应用(1) 大数据处理中的降维;(2) PCA 分析作图;(3) 三种常见的聚类分析:K-means、层次分析与 SOM(4) 热图和 hcluster 图的 R 语言实现;C4 一组代谢组学数据的降维与聚类分析的 R 演练(1) 数据解析;(2) 演练与操作;第四天D1 在线代谢组分析网页 Metaboanalyst 操作(1) 用 R 将数据清洗成网页需要的格式;(2) 独立组、配对组和多组的数据格式问题;(3) Metaboanalyst 中的上游分析(原始数据峰提取、峰对齐与搜库)(4) Metaboanalyst 的 pipeline 以及参数设置和注意事项;(5) Metaboanalyst 的结果查看和导出;(6) Metaboanalyst 的数据编辑;(7) 全流程演练与操作。D2 代谢组学数据清洗与 R 语言进阶(1) 代谢组学中的 t、fold-change 和响应值;(2) 数据清洗流程;(3) R 语言 tidyverse;(4) 数据预处理:数据过滤与数据标准化(样本的 Normalization 和代谢物的 Scaling);(5) 代谢组学数据清洗演练;第五天E1 文献数据分析部分复现(1 篇)(1) 文献深度解读;(2) 实操:从原始数据下载到图片复现;(3) 学员实操。E2 机器学习与代谢组学顶刊解读(3 篇);(1) Signal Transduction and Targeted Therapy 一篇有关饥饿对不同脑区代谢组学影响变化的小鼠脑组织代谢图谱类的文献;(数据库型)(2) Cell 一篇代谢组学孕妇全程血液代谢组学分析得出对孕周和孕产期预测的代谢标志物的文献;(生物标志物型)(3) Nature 一篇对胰腺癌患者肠道菌群的代谢组学分析找到可以提高化疗效果的代谢物的文献。(机制研究型)06机器学习微生物组学 内容可向下滑动第一天1. 微生物学基础知识回顾2. 机器学习基本概念介绍a. 什么是机器学习b. 监督学习、无监督学习c. 常用机器学习模型介绍3. 混淆矩阵4. ROC 曲线第二天R 语言简介与实操1. R 语言概述2. R studio 软件与 R 包安装3. R 语言语法及数据类型4. 条件语句和循环Linux 实操1. Linux 操作系统2. Linux 操作系统的安装与设置3. 网络配置与服务进程管理4. Linux 的远程登录管理5. 常用的 Linux 命令6. 在 Linux 下获取基因数据7. Shell script 与 Vim 编辑器第三天微生物组常用分析方法(实操)1. 微生物丰度分析2. 转录组丰度分析3. 进化树分析4. 降维分析第四天机器学习在微生物组学中的应用案例分享1. 疾病预测应用:利用机器学习基于微生物组学数据预测疾病状态2. 肠道菌群研究:机器学习研究饮食对肠道微生物的影响第五天机器学习模型训练和分析(实操)1. 加载数据及数据归一化2. 构建训练模型(GLM, RF, SVM)3. 模型参数优化4. 模型错误率曲线绘制5. 混淆矩阵计算6. 重要特征筛选7. 模型验证,ROC 曲线绘制利用模型进行预测利用机器学习基于微生物组学数据预测宿主表型1. 加载数据2. 数据归一化3. OUT 特征处理4. 机器学习模型构建(RF, KNN, SVM, Lasso 等多种机器学习方法)5. 绘制 ROC 曲线,比较不同机器学习模型模型性能评估利用机器学习基于临床特征和肠道菌群预测疾病风险1. 加载数据2. 机器学习模型构建(RF, gbm, SVM 等等)3. 交叉验证4. 模型性能评估07深度学习基因组学内容可向下滑动第一天理论部分深度学习算法介绍1.有监督学习的神经网络算法1.1 全连接深度神经网络 DNN 在基因组学中的应用举例1.2 卷积神经网络 CNN 在基因组学中的应用举例1.3 循环神经网络 RNN 在基因组学中的应用举例1.4 图卷积神经网络 GCN 在基因组学中的应用举例2.无监督的神经网络算法2.1 自动编码器 AE 在基因组学中的应用举例2.2 生成对抗网络 GAN 在基因组学中的应用举例基因组常用深度学习框架1. 介绍深度学习工具包 tensorflow, keras,pytorch2. 在工具包中识别深度学习模型要素2.1.数据表示2.2.张量运算2.3.神经网络中的“层”2.4.由层构成的模型2.5.损失函数与优化器2.6.数据集分割2.7.过拟合与欠拟合基因组学基础1. 基因组数据库2. 表观基因组3. 转录基因组4. 蛋白质组5. 功能基因组实操内容1.Linux 操作系统1.1 常用的 Linux 命令1.2 Vim 编辑器1.3 基因组数据文件管理, 修改文件权限1.4 查看探索基因组区域2.Python 语言基础2.1.Python 包安装和环境搭建2.2.常见的数据结构和数据类型3. 安装深度学习工具包 tensorflow, keras,pytorch,在工具包中识别深度学习模型要素第二天理论部分1. 介绍 keras_dna 平台,搭建基因组学常用深度学习应用案例2. 深度学习模型 DeepG4 从 Chip-Seq 及 DnaseSeq 中识别基序特征 G4实操内容1.基因组数据处理搭建深度学习模型1.1 安装并使用 keras_dna 处理各种基因序列数据如 BED、 GFF、GTF、BIGWIG、BEDGRAPH、WIG 等1.2 使用 keras_dna 设计深度学习模型1.3 使用 keras_dna 分割训练集、测试集1.4 使用 keras_dna 选取特定染色体的基因序列等2.使用 keras_dna 平台复现 DeepG4 模型,从 Chip-Seq 中识别 G4 特征第三天理论部分深度学习在基因调控预测中的应用1. selene_sdk 预测 DNA 甲基化及转录调控因子等 DeepSEA2. 循环神经网络 RNN 从 RNA 序列中预测 pre-miRNA,dnnMiRPre实操内容复现卷积神经网络 CNN 识别基序特征 DeepG4、基因调控因子 DeepSEA,1. 安装 selene_sdk,复现 DeepSEA 预测 DNA 甲基化,非编码基因变异等基因调控因子2. 复现循环神经网络 RNN 工具 dnnMiRPre,从 RNA-Seq 中预测 pre-miRNA第四天理论部分深度学习在预测疾病表型及生物标志物上的应用1. 从高维基因表达数据中识别乳腺癌分型的自动编码机深度学习工具DeepType2. 深度学习在识别拷贝数变异 DeepCNV 模型实操内容1. 复现 DeepType,从 METABRIC 乳腺癌数据中区分乳腺癌亚型2. 解析 DeepType 中新的乳腺癌亚型的标志基因3. 复现 DeepCNV 利用 SNP 微阵列联合图像分析识别拷贝数变异第五天理论部分深度学习在预测药物反应机制上的应用1. 联合肿瘤基因标记及药物分子结构预测药物反应机制的深度学习工具 SWnet实操内容1. 预处理药物分子结构信息2. 计算药物相似性3. 在不同数据集上构建 self-attention SWnet4. 评估 self-attention SWnet5. 构建多任务的 SWnet6. 构建单层 SWnet7. 构建带权值层的 SWnet08深度学习质谱蛋白组学内容可向下滑动第一天蛋白质组学测序技术及数据库理论讲解1.蛋白质组学测序质谱技术2.介绍蛋白质组学数据库3.深度学习解析蛋白质组学模型介绍GPU服务器上机实操1.Linux操作系统1.1常用的Linux命令1.2 Vim编辑器1.3基因组数据文件管理, 修改文件权限1.4查看探索基因组区域2.Python语言基础2.1.Python包安装和环境搭建2.2.常见的数据结构和数据类型第二天深度学习识别质谱测序中蛋白质肽的理化性质上午 理论讲解1.深度学习模型预测色谱法保留时间及碎片离子浓度Prosit2.深度学习预测质谱测序中截面碰撞CCS工具DeepCollisionalCrossSection3.深度学习预测单细胞蛋白组学覆盖率DeepSCP模型下午 深度学习模型Python代码解析及GPU服务器上机实操1.复现深度学习模型预测色谱法保留时间及碎片离子浓度Prosit模型2.复现深度学习预测质谱测序中截面碰撞工具DeepCollisionalCrossSection3.复现深度学习预测单细胞蛋白组学覆盖率DeepSCP模型第三天深度学习识别肽及肽组装上午 理论讲解1.深度学习从宏蛋白组学中识别肽 DeepFilter模型2.深度学习从蛋白质数据库中识别肽DeepDIA模型3.深度学习实现肽组装DeepNovo 及DeepNovo-DIA模型下午 深度学习模型Python代码解析及GPU服务器上机实操1.复现深度学习从宏蛋白组学中识别肽 DeepFilter模型2.复现深度学习从蛋白质数据库识别肽DeepDIA模型复现深度学习实现肽组装DeepNovo及DeepNovo-DIA模型第四天深度学习识别翻译后修饰结合位点识别疾病及药物靶点上午 理论讲解 1.胶囊网络深度学习模型预测翻译后修饰结合位点模型CapsNet_PTM2.注意力机制深度学习预测MHC I 结合位点ACME模型 3.深度学习模型PUFFIN量化Peptide-MHC结合不确定性提升药物设计中高亲和力肽筛选4.深度学习模型预测癌症抗原ACP-MHCNN 模型下午 深度学习模型Python代码解析及GPU服务器上机实操1.复现胶囊网络深度学习模型预测翻译后修饰结合位点模型CapsNet_PTM2.复现注意力机制深度学习预测pan-specific MHC I 结合位点ACME模型 3.复现深度学习模型PUFFIN量化Peptide-MHC结合不确定性提升药物设计中高亲和力肽筛选4.复现深度学习模型预测癌症抗原ACP-MHCNN模型第五天深度学习识别蛋白质功能上午 理论讲解1.深度学习模型3D卷积网络预测蛋白质-蛋白质相互作用DeepRank2.深度学习模型量化蛋白质表达DLNetworkForProteinAbundance3.基于自然语言注意力机制深度学习模型预测蛋白质功能SPROF-GO4.深度学习模型PCfun 预测蛋白质复合物Gene Ontology功能下午 深度学习模型Python代码解析及GPU服务器上机实操1.复现深度学习模型3D卷积网络预测蛋白质-蛋白质相互作用DeepRank2.复现深度学习模型量化蛋白质表达DLNetworkForProteinAbundance3.复现基于自然语言注意力机制深度学习模型预测蛋白质功能SPROF-GO4.复现深度学习模型PCfun 预测蛋白质复合物Gene Ontology功能09CRISPR-Cas9基因编辑技术内容可向下滑动第一天绪论1.课程简介与学习目标2.基因编辑技术概述2.1 基因编辑的定义、核心原理与技术分类2.2 基因编辑与合成生物学的交叉3.技术应用领域全景图3.1基础研究3.2农业育种3.3疾病治疗3.4生物制造4.伦理与安全问题初探4.1 脱靶效应与基因驱动4.2 人类胚胎编辑的伦理边界第二天基因编辑技术发展简史1.1 ZFN1.2 TALENs 1.3 局限性CRISPR技术的革命性突破2.1 原核生物免疫机制的发现历程 2.2 Cas9系统在真核细胞的应用验证 2.3里程碑事件与诺贝尔奖解读2.4中国科学家在基因编辑领域的突出贡献第三天CRISPR常用工具与实操CRISPR-Cas系统1.1 CRISPR系统的起源与机制1.2 主要工具酶的特征与选择(实操)1.3 sgRNA的设计与优化(实操)1.4 CRISPR 筛选(CRISPR Screnning)1.5 CRISPR-Cas系统在模式生物中的应用1.6 CRISPR-Cas系统与CAR-T细胞治疗碱基编辑器(Base Editing)2.1 腺嘌呤碱基编辑器(ABE)2.2 脱氨酶的活性优化及对脱靶效应的控制2.3 胞嘧啶碱基编辑器(CBE)2.4 C-to-T编辑的特异性与效率平衡2.5 利用碱基编辑器构建动物模型2.6 单核苷酸突变矫正先导编辑(Prime Editing)3.1 逆转录酶的特点与选择3.2 pegRNA的设计与优化(实操)3.3 双pegRNA编辑系统3.4 PM359治疗慢性肉芽肿病CRISPR激活与抑制系统(CRISPRa/i)4.1 dCas94.2 dCas9与转录激活因子、转录抑制因子的融合4.3 CRISPRa在干细胞重编程中的应用4.4 CRISPRa/i研究癌症相关基因的功能网络其他基因编辑工具5.1 大片段DNA精准操纵工具5.2 Cre-loxP系统在模式动物中的应用第四天递送系统CRISPR递送系统概述1.1 CRISPR技术的基本原理和发展历程1.2 CRISPR递送系统的重要性和挑战病毒载体递送系统2.1 病毒载体的类型和特点(如腺相关病毒AAV)2.3病毒载体的构建和优化2.3 病毒载体在CRISPR递送中的应用和案例分析非病毒递送系统3.1 纳米颗粒(如脂质纳米颗粒LNP)的设计与应用3.2 电穿孔技术的原理和应用3.3 非病毒递送系统的优缺点分析植物病毒递送系统4.1 植物-弹状病毒在CRISPR递送中的应用4.2 植物病毒递送系统的优化和挑战第五天CRISPR应用CRISPR在基础研究中的应用1.1 CRISPR在基因功能研究中的应用1.2 CRISPR在疾病模型创建中的应用1.3 CRISPR在基因调控研究中的应用CRISPR在遗传病治疗中的应用2.1 遗传病数据库的建立和应用2.2 CRISPR治疗遗传病的案例分析2.3FDA批准的CRISPR疗法介绍(镰状细胞贫血、杜氏肌肉营养不良、癌症的免疫疗法)小结与展望基本内容小结+当前技术瓶颈分析1.1 递送效率与组织靶向性难题1.2复杂性状的多基因协同编辑新兴技术发展方向2.1 DNA 聚合酶编辑器2.2 CRISPR 引导的重组酶和转座子2.3 表观基因组编辑2.4 RNA编辑2.5 AI与基因编辑新型CRISPR工具临床转化路线图3.1 体内编辑与体外编辑的产业化路径3.2 基因编辑疗法监管体系的国际比较基因编辑的道德考量和安全性10AI智慧医疗影像技术内容可向下滑动第一天智慧医疗中的图像处理与分析概论1、阐述智慧医疗定义,回溯其发展历程,剖析当下智慧医疗多元应用场景,如远程诊断、智能辅助决策等,展望未来在精准医疗、个性化治疗方面的潜力。2、深入讲解医疗图像在智慧医疗体系里的重要性,从辅助诊断、治疗规划到疗效评估全流程解析,明确课程知识、技能双目标,梳理学习计划,强调结合实际案例的实践导向。医疗图像的基本类型和获取方法1、详细科普医疗图像主要类型,简要介绍X射线成像原理、CT断层扫描机制、MRI磁共振成像特色、超声成像的声波利用,每种类型搭配实际应用场景,如X射线在骨折检测,MRI在脑部病变诊断的应用。第二天图像处理和分析的基本技术和算法1、实操演示图像预处理技术,展示去噪算法应用、增强视觉效果手段、对比度调整技巧的实际效果,如使用OpenCV进行高斯模糊去噪、中值滤波去除椒盐噪声,直方图均衡化增强对比度,每种方法结合具体医学图像案例,如去除X光图像中的噪声,增强CT图像的对比度,展示实际的OpenCV代码示例。2、简要讲解边缘检测原理,对比常用边缘检测算子如Sobel、Canny算子的效果,解读形态学操作如腐蚀、膨胀在图像处理中的功效,展示如何用OpenCV实现这些操作。3、拆解图像分割技术,简要介绍阈值分割法、区域分割策略、边缘分割原理,结合医学图像案例,如用阈值分割法分离MRI图像中的脑组织,用区域生长算法标记肿瘤区域,用边缘分割勾勒器官轮廓,展示如何用Python和OpenCV实现这些分割方法,提供相关的代码示例和实际的图像处理结果。深度学习在医疗图像分析中的应用1、从基础概念入手,介绍深度学习框架,简要讲解CNN卷积神经网络架构,说明卷积层、池化层、全连接层的作用,结合医学图像识别中的应用案例,讲解如何用深度学习模型实现图像分类、目标检测和分割。2、结合实际案例,剖析深度学习在图像分类、目标检测、分割中的应用流程,实操演示常用深度学习模型YOLOv5在医疗图像里的调用与优化,提供实际的模型定义和训练代码示例,如用YOLOv5检测X光图像中的肺结节,展示如何配置模型参数、加载预训练模型、训练和优化模型,实现医学图像的目标检测任务。第三天Transformer 技术在医疗图像分析中的应用1、讲解Transformer基本原理,简要介绍架构里自注意力机制运作逻辑,说明自注意力机制如何捕捉图像中不同区域之间的长距离依赖关系,提升模型对上下文的理解。2、分享Transformer在医疗图像分析中的前沿应用案例,如基于Transformer的模型在医学图像分割、分类和目标检测中的应用,讲解如何利用Transformer架构的全局特征提取能力提升诊断准确性,以脑肿瘤分割为例,演示Transformer如何更准确地识别肿瘤边界。3、指导学员动手实践,使用Transformer模型进行医疗图像分析任务,提供相关的代码示例和开源项目链接,如用Vision Transformer(ViT)实现医学图像分类,展示如何将医学图像分割成patches并输入到Transformer中,实现端到端的分类任务。多模态数据融合在医疗图像分析中的应用1、阐述多模态数据概念,列举常见类型,如影像、文本报告、生理数据融合,讲解多模态数据在医学诊断中的互补优势,如影像数据提供组织结构信息,文本报告包含症状和病史,生理数据反映身体机能状态。 2、介绍多模态数据融合方法,如特征级融合、决策级融合技术,结合实际案例,如融合CT图像和临床指标对疾病分期,演示如何用Python实现特征级融合,将不同模态的数据拼接成多维特征向量,输入到机器学习模型中进行分类或回归任务。 3、分析多模态融合在提升医疗图像分析准确性上的作用,展示实际的多模态数据融合代码示例,如用Keras或PyTorch实现多个神经网络分支分别处理不同模态的数据,最后在决策层进行融合。第四天医疗图像分析在疾病诊断和治疗中的实际案例1、以肺结节筛查诊断为切入点,展示图像分析辅助医生精准定位、良恶性判断流程,通过超声、CT、MRI 等检查手段对病情进行综合评估和诊断,提供实际的肺结节检测代码示例和数据集,讲解如何用深度学习模型在胸部CT图像中自动检测肺结节,并根据结节特征判断良恶性。2、延伸至肿瘤识别与分割,讲解如何利用图像处理技术勾勒肿瘤边界,探讨心血管疾病诊断评估,通过血管影像分析病情,展示实际的心血管疾病诊断代码示例和数据集,如用U-Net模型分割脑部MRI图像中的肿瘤区域,用血管成像技术分析冠状动脉狭窄程度。图像处理工具与实践与深度学习模型的训练1、介绍常用图像处理工具OpenCV、MATLAB 功能模块,实操演示图像处理算法实现与优化技巧,提供实际的图像处理代码示例和优化方法,如用OpenCV实现医学图像的量化、特征分析等功能,用MATLAB进行快速傅里叶变换和小波变换。2、指导学员使用OpenCV完成图像预处理和分析任务,上手深度学习框架训练医疗图像分类模型,掌握模型调参方法,提供实际的模型训练代码示例和调参技巧,如在ResNet模型的基础上进行微调,优化学习率、批量大小、正则化参数等。第五天医疗图像分析项目设计与实施1、引导学员进行项目需求分析,规划项目整体架构,涵盖数据收集、处理、模型选择到结果评估全流程,强调项目的临床实用性和快速部署,鼓励学员结合实际工作中的问题设计项目主题。2、提供结构化的课程学习计划,围绕主题进行拓展学习和实践,推荐相关书籍、学术论文、在线课程和实操项目资源,辅助学习者深化理解课程知识,提升专业技能。学习目标AI蛋白质设计课程旨在为学生提供深度学习与蛋白质设计领域的全面知识。通过讲授深度学习的基本概念和前沿技术,学生将理解深度学习在生物信息学特别是蛋白质设计中的具体应用。学生将了解如何使用主流深度学习框架PyTorch进行模型构建与优化,并通过实践操作掌握蛋白质结构预测、蛋白质功能预测和分子对接等关键技术。课程将介绍AlphaFold等先进模型,并探讨其在药物发现中的重要性。同时通过多肽设计、逆向中心法则等专题,学生将全面了解从功能推导结构和从零开始设计蛋白质的策略。合成生物与基因电路设计通过理论与实践结合,掌握合成生物学基础、基因电路设计、代谢途径优化、基因编辑技术及数学建模,培养学员在合成生物领域的创新能力和系统思维,为未来研究与应用奠定基础。CADD计算机辅助药物设计掌握包括PDB数据库、靶点蛋白、蛋白质-配体、蛋白-配体小分子、蛋白-配体结构、notepad的介绍和使用、分子对接、蛋白-配体对接、虚拟筛选、蛋白-蛋白对接、蛋白-多糖分子对接、蛋白-水合对接、Linux安装、gromacs分 子动力学全程实操、溶剂化分子动力学模拟AIDD人工智能药物设计与发现本课程让学员了解药物发现的前沿背景,学习人工智能领域的各类常见算法,熟悉工具包的安装与使用,掌握一定的算法编程能力,能够运用计算机方法研究药物相关问题。通过大量的案例讲解和实践操作,具备一定的AIDD模型构建和数据分析能力机器学习代谢组学熟悉代谢组学和机器学习相关硬件和软件;熟悉代谢组学从样本处理到数据分析的全流程;能复现至少1篇CNS或子刊级别的代谢组学文章图片。机器学习微生物组学课程将涵盖机器学习技术在微生物数据分析中的应用,包括基因组序列分析、基因调控网络构建和多组学数据整合等,并带领学员们深度使用R语言,Python语言实地操作演示。深度学习基因组学课程深入学习与了解深度学习基本框架与逻辑,同时掌握基本的生物信息学软件(Linux、R、python等)的使用,让学员能更好的应对基因组数据,挖掘出超越已有知识的新知识。而构建好的深度学习模型去探求新的研究思路和寻找新的潜在生物学机制,更好的服务于自身的科学研究和探索的过程中。深度学习质谱蛋白组学课程通过对这些深度学习在蛋白组学中的应用案例进行深度讲解和实操,让学员能够掌握深度学习分析蛋白组学数据流程,系统学习深度学习及蛋白组学理论知识及熟悉软件代码实操,熟练掌握这些前沿的分析工具的使用以及研究创新深度学习算法解决生物学及临床疾病问题与需求。CRISPR-Cas9基因编辑技术课程从全局出发,由浅入深,课程通过基础入门+应用案例实操演练的方式,从最初的原理讲解到最后的应用实战,学完本课程你将掌握基因编辑技术的相关原理及其应用,此外可以学到基因编辑系统的优化策略,可以学到如何操作常用的生物学软件。能够快速运用到自己的科研项目和课题上。AI智慧医疗影像技术课程掌握智慧医疗图像处理与分析技术,包括图像预处理、深度学习应用、多模态数据融合及项目实践,培养学员在医疗影像分析中的技术应用能力和问题解决能力讲师介绍AI蛋白设计授课老师来自北大人工智能学院,作为核心成员参与过清华某初创公司大语言模型项目,主导过北大长沙研究院的核心项目智能体的开发,此外和包括百度,字节等多个公司和北大的合作中作为主要负责人,累计参与项目金额超过千万。目前发过多篇ACL等计算机顶会和sci论文。合成生物与基因电路设计两位授课老师均来自清华大学,干湿结合分别引领本课程的实验设计和建模分析,研究方向涉及植物生物学、合成生物学与生物信息学。在对应领域中科院一区有多篇产出,同时曾作为队长和评委多次参加过合成生物学(iGEM)顶级赛事,曾获得全球十佳项目(TOP10)和多个单项奖及提名。CADD计算机辅助药物设计杨教授在计算机/人工智能辅助药物设计(CADD/AIDD)领域具有多年的研究经验,熟悉分子对接和虚拟筛选,分子动力学模拟、先导物优化、人工智能等计算工具和方法,在J. Med. Chem, J. Chem. Inf. Model,J. Phys. Chem. B, Frontiers in Pharmacology 等国际主流期刊发表学术论文20余篇,主持或参与国家自然科学基金、河南省重点研发、企业横向项目十余项。AIDD人工智能药物设计与发现AIDD授课老师曹老师,有十余年的计算机算法研究和程序设计经验。研究方向涉及生物信息学,深度学习,药物合成路径设计,药物不良反应等。发明专利5项,参与国家重点科研项目4项,发表SCI高水平论文10篇,包括BMC Bioinformatics, Journal of Biomedical Informatics, International Journal of Molecular Sciences等知名期刊。机器学习代谢组学讲老师来自985高校神经科学博士,主要利用代谢组学、转录组学和分子生物学等技术研究神经内科慢性病的发病机制和生物标志物。擅长高效液相色谱-质谱联用(LC-MS)技术进行非靶向和靶向代谢组学从样本制备到数据分析的全流程研究,以及多组学大数据的生物信息学整合分析。5年内在J Clin Invest, EBioMedicine, Cell Death Dis, Cell Death Discov, Nanotoxicology等杂志发表SCI论文10篇。机器学习微生物组学主讲老师来自清华大学,研究方向包括生物信息学、机器学习与微生物基因组学,大模型与蛋白质定向进化等。同时他在图神经网络和疾病药物靶向等知识图谱技术方面有丰富的经验,带领并指导多次团队在国际基因工程竞赛(iGEM)中获得国际金牌,并一作发表了多篇一区高水平SCI论文。深度学习基因组学主讲老师刘老师,生物信息学PI,有十余年的测序数据分析经验。研究领域涉及人工智能、自然语言处理、功能基因组学、转录组学、miRNA及靶基因网络分析,单细胞测序数据分析,基因调控网络时序分析,蛋白质互作网络分析,多组学联合分析等。主持省自然科学基金等项目4项,发表SCI论文23篇,论著一部深度学习质谱蛋白组学主讲老师刘老师,生物信息学博士,从事生物信息及医学人工智能研究 15 年,开发过数个生物信息学工具,发表 SCI 论文 20 余篇,其中人工智能算法文章近 10 篇,编著医学数据分析实用教材一部,研究致力于医学人工智能在复杂疾病诊疗中的应用。CRISPR-Cas9基因编辑技术主讲老师均来自清华大学、浙江大学、西湖大学等国内顶尖高校,他们在基因编辑及相关领域拥有深厚的学术背景和丰富的研究经验。在博士期间深入研究基因编辑技术,发表了多篇高水平论文(包括子刊和多篇一区文章)并有各类系统扎实的设计实操经验,助力学员们在基因编辑领域取得更大的进步和发展。AI智慧医疗影像技术授课老师来自清华大学, 拥有生物医学信息学与信息工程处理等丰富的经验。在对应领域中科院一区有多篇产出,也有过多篇CCF-A类同领域,如ICLR的产出和审稿经验,熟悉计算机领域算法和生物医学图像的加工和处理方式。授课时间01AI蛋白质设计2025.06.14全天(9点-11点半-下午1点半-5点)2025.06.15全天(9点-11点半-下午1点半-5点)2025.06.16晚上授课(19:00-22:00)2025.06.18晚上授课(19:00-22:00)2025.06.19晚上授课(19:00-22:00)2025.06.20晚上授课(19:00-22:00)2025.06.21全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放02合成生物与基因电路设计2025.06.14全天(9点-11点半-下午1点半-5点)2025.06.15全天(9点-11点半-下午1点半-5点)2025.06.17晚上授课(晚19:00—晚22:00)2025.06.18晚上授课(晚19:00—晚22:00)2025.06.19晚上授课(晚19:00—晚22:00)2025.06.20晚上授课(晚19:00—晚22:00)2025.06.21全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放03CADD计算机辅助药物设计2025.06.10晚上授课(19:00-22:00)2025.06.11晚上授课(19:00-22:00)2025.06.12晚上授课(19:00-22:00)2025.06.13晚上授课(19:00-22:00)2025.06.15全天(9点-11点半-下午1点半-5点)2005.06.16晚上授课(19:00-22:00)2005.06.17晚上授课(19:00-22:00)2005.06.18晚上授课(19:00-22:00)2005.06.19晚上授课(19:00-22:00)2025.06.21全天(9点-11点半-下午1点半-5点)2025.06.22全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放04AIDD人工智能药物设计与发现2025.06.14全天(9点-11点半-下午1点半-5点)2025.06.15全天(9点-11点半-下午1点半-5点)2025.06.17晚上授课(晚19:00—晚22:00)2025.06.18晚上授课(晚19:00—晚22:00)2025.06.19晚上授课(晚19:00—晚22:00)2025.06.20晚上授课(晚19:00—晚22:00)2025.06.22全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放05机器学习代谢组学2025.06.21全天(9点-11点半-下午1点半-5点)2025.06.22全天(9点-11点半-下午1点半-5点)2025.06.23晚上授课(晚19:00—晚22:00)2025.06.24晚上授课(晚19:00—晚22:00)2025.06.25晚上授课(晚19:00—晚22:00)2025.06.26晚上授课(晚19:00—晚22:00)2025.06.28全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放06机器学习微生物组学2025.06.22全天(9点-11点半-下午1点半-5点)2025.06.23晚上授课(晚19:00—晚22:00)2025.06.24晚上授课(晚19:00—晚22:00)2025.06.25晚上授课(晚19:00—晚22:00)2025.06.26晚上授课(晚19:00—晚22:00)2025.06.28全天(9点-11点半-下午1点半-5点)2025.06.29全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放07深度学习基因组学2025.06.14全天(9点-11点半-下午1点半-5点)2025.06.15全天(9点-11点半-下午1点半-5点)2025.06.17晚上授课(晚19:00—晚22:00)2025.06.18晚上授课(晚19:00—晚22:00)2025.06.19晚上授课(晚19:00—晚22:00)2025.06.20晚上授课(晚19:00—晚22:00)2025.06.21全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放08深度学习质谱蛋白组学2025.06.22全天(9点-11点半-下午1点半-5点)2025.06.23晚上授课(晚19:00—晚22:00)2025.06.24晚上授课(晚19:00—晚22:00)2025.06.25晚上授课(晚19:00—晚22:00)2025.06.26晚上授课(晚19:00—晚22:00)2025.06.28全天(9点-11点半-下午1点半-5点)2025.06.29全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放09CRISPR-Cas9基因编辑技术2025.06.22全天(9点-11点半-下午1点半-5点)2025.06.23晚上授课(晚19:00—晚22:00)2025.06.24晚上授课(晚19:00—晚22:00)2025.06.25晚上授课(晚19:00—晚22:00)2025.06.26晚上授课(晚19:00—晚22:00)2025.06.28全天(9点-11点半-下午1点半-5点)2025.06.29全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放10AI智慧医疗影像技术2025.06.14全天(9点-11点半-下午1点半-5点)2025.06.15全天(9点-11点半-下午1点半-5点)2025.06.17晚上授课(晚19:00—晚22:00)2025.06.18晚上授课(晚19:00—晚22:00)2025.06.19晚上授课(晚19:00—晚22:00)2025.06.20晚上授课(晚19:00—晚22:00)2025.06.21全天(9点-11点半-下午1点半-5点)腾讯会议直播上课课后提供直播回放报名费用及福利1.AI蛋白质设计 公费价:每人每班¥6380 元 自费价:每人每班¥5880元2.CADD计算机辅助药物设计3.AIDD人工智能药物发现4.机器学习代谢组学5.深度学习在基因组学中的应用6.合成生物与基因电路设计7.深度学习在质谱蛋白组学中的应用8.机器学习微生物组学9.CRISPR-Cas9基因编辑技术公费价:每人每班¥5880自费价:每人每班¥548010.AI智慧医疗影像技术公费价:每人每班¥4680自费价:每人每班¥4380优惠一报二赠一:10880(赠送课程任选)优惠二报三赠一:13880(赠送课程任选)报四赠二:18880元(赠送课程任选)特惠三全报:25880两年内可免费参加本公司举办的任何课程(不限次数及课程,包括之后的新开课)限时福利:报名成功后转发朋友圈或转发50人以上群聊即可获得300元现金红包(只限前15名)特惠福利:报一送一可额外送的回放(包含全套课程回放和课件资料ppt)机器学习生物医学单细胞测序与空间转录组深度学习宏基因组学蛋白晶体结构解析报名费用可开具正规报销发票及提供相关缴费证明、邀请函,可提前开具报销发票、文件用于报销 。报名缴费后即可获得全套预习资料供大家课前准备证书:参加培训并通过考试的学员,可以申请获得工业和信息化部工业文化发展中心颁发的“工业强国建设素质素养提升尚工行动”岗位能力适应评测证书。该证书可在中心官网查询,可作为能力评价,考核和任职的重要依据。评测证书查询网址:www.miit-icdc.org(自愿申请,须另行缴纳考试费500元/人)SIMPLICITY官方联系人联系人:苏老师,报名咨询电话:13783571273(同V)往期学员好评截图:
基因疗法
100 项与 Jiangsu Baidu Medical Technology Co., Ltd. 相关的药物交易
登录后查看更多信息
100 项与 Jiangsu Baidu Medical Technology Co., Ltd. 相关的转化医学
登录后查看更多信息
组织架构
使用我们的机构树数据加速您的研究。
登录
或

管线布局
2026年02月08日管线快照
无数据报导
登录后保持更新
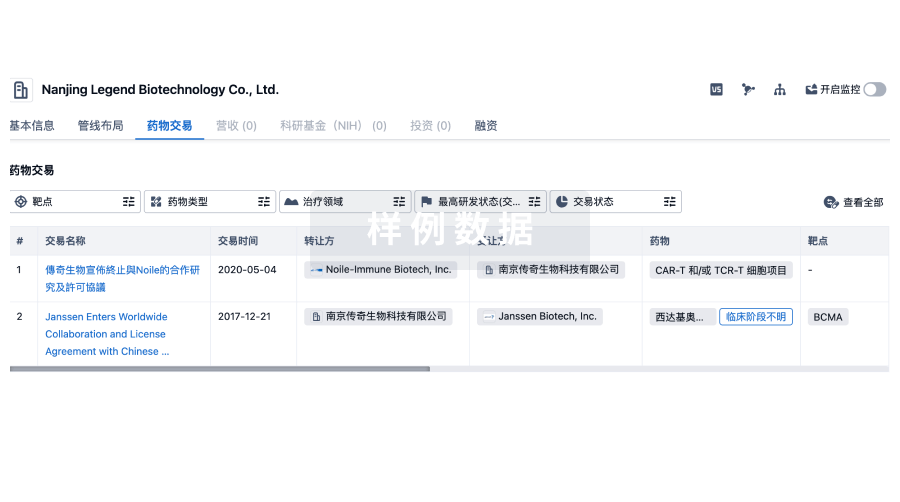
药物交易
使用我们的药物交易数据加速您的研究。
登录
或
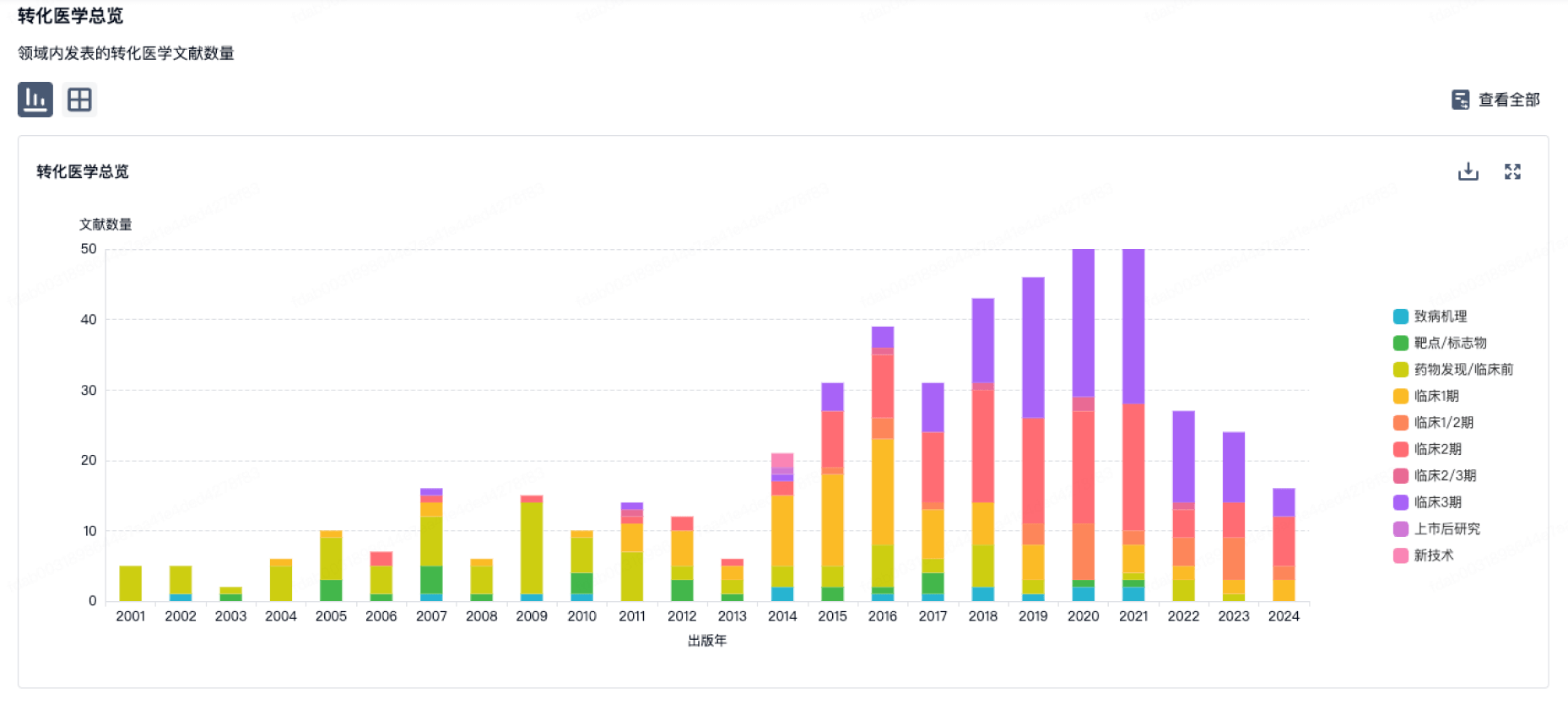
转化医学
使用我们的转化医学数据加速您的研究。
登录
或
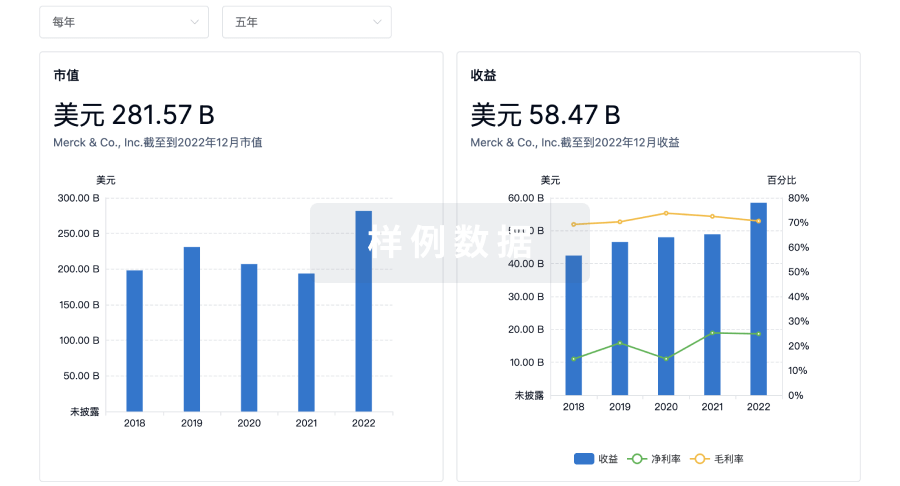
营收
使用 Synapse 探索超过 36 万个组织的财务状况。
登录
或
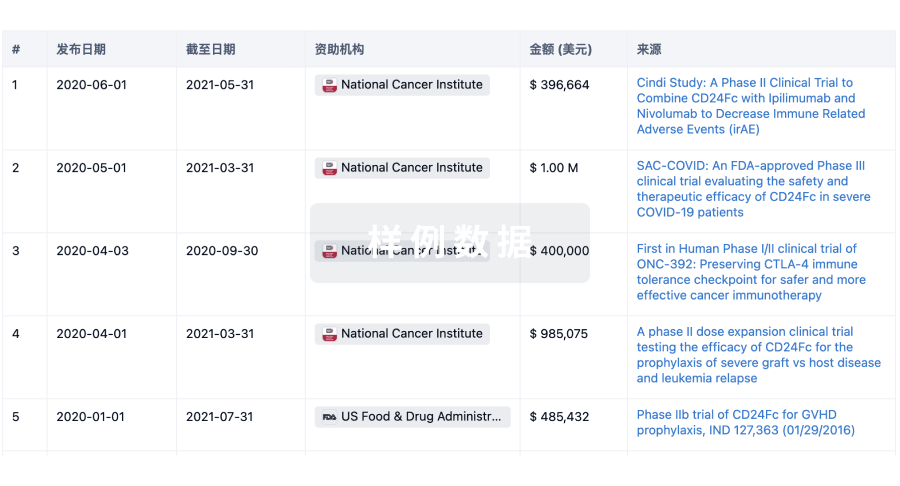
科研基金(NIH)
访问超过 200 万项资助和基金信息,以提升您的研究之旅。
登录
或
投资
深入了解从初创企业到成熟企业的最新公司投资动态。
登录
或
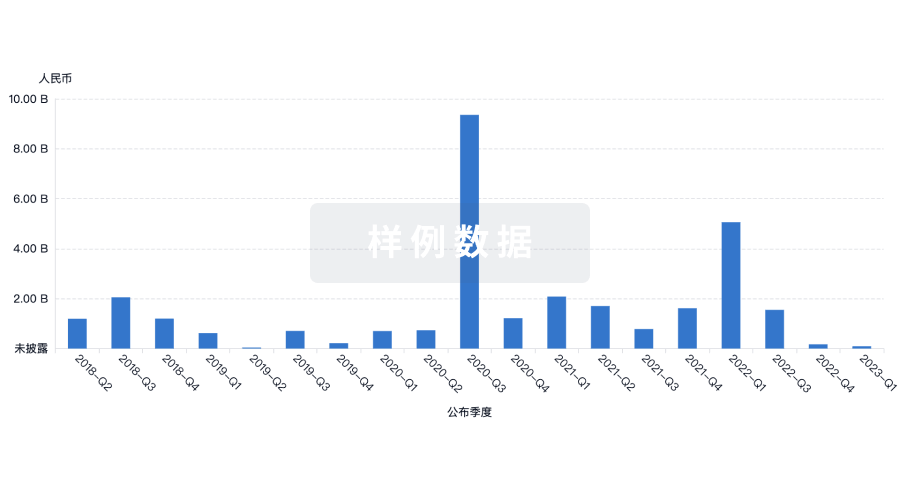
融资
发掘融资趋势以验证和推进您的投资机会。
登录
或
生物医药百科问答
全新生物医药AI Agent 覆盖科研全链路,让突破性发现快人一步
立即开始免费试用!
智慧芽新药情报库是智慧芽专为生命科学人士构建的基于AI的创新药情报平台,助您全方位提升您的研发与决策效率。
立即开始数据试用!
智慧芽新药库数据也通过智慧芽数据服务平台,以API或者数据包形式对外开放,助您更加充分利用智慧芽新药情报信息。
生物序列数据库
生物药研发创新
免费使用
化学结构数据库
小分子化药研发创新
免费使用