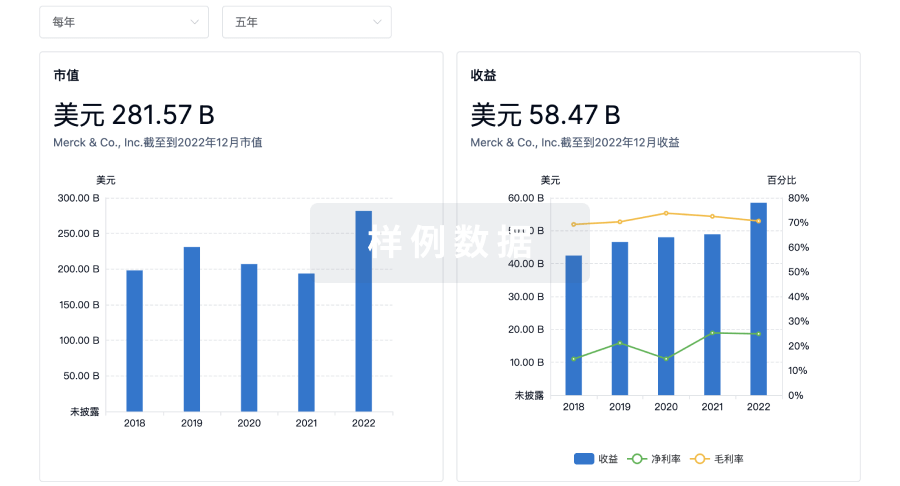
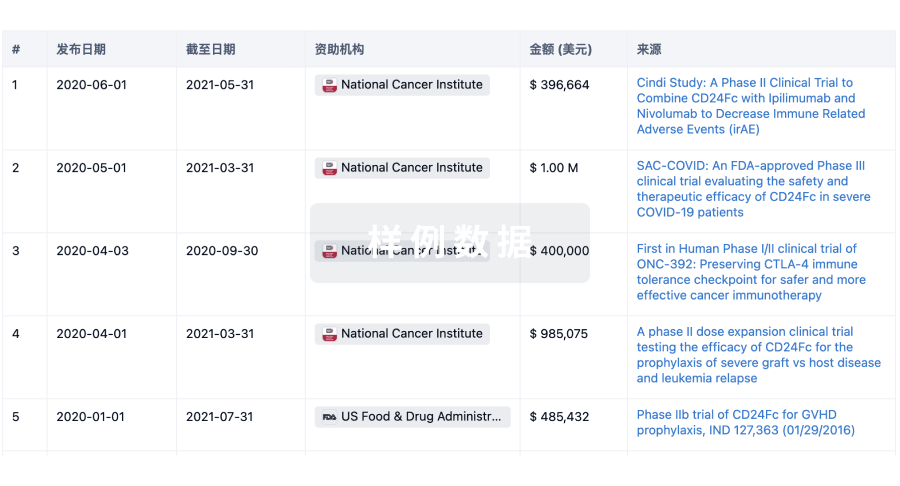
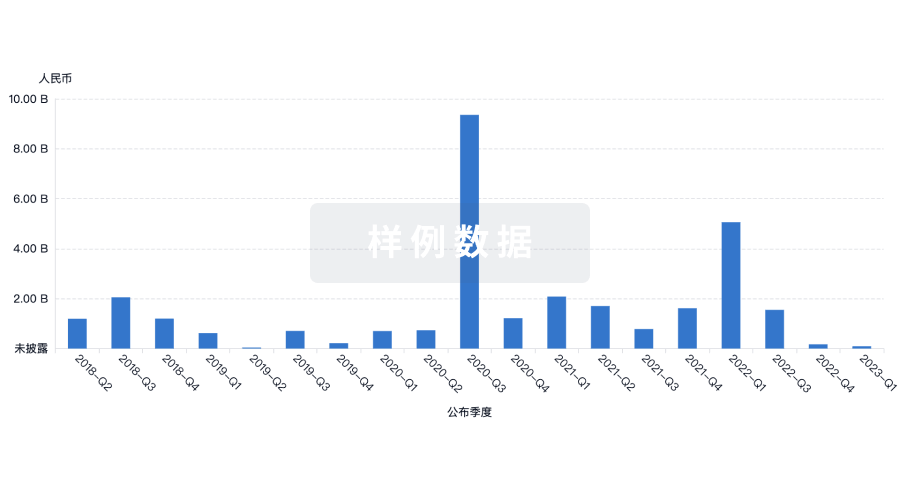
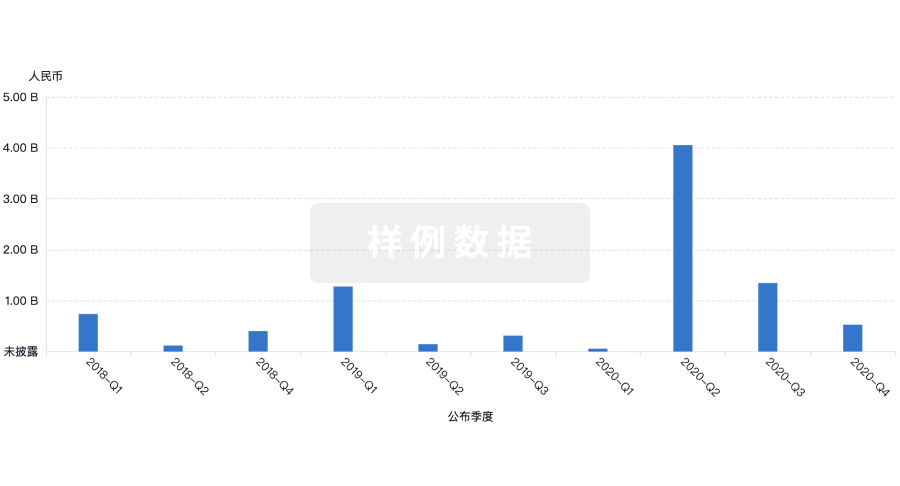
100 项与 Beijing Mida Biomedical Technology Co., Ltd. 相关的临床结果
0 项与 Beijing Mida Biomedical Technology Co., Ltd. 相关的专利(医药)
引言人类小脑包含大量神经元,表现出与大脑不同的发育模式。2024年2月26日,复旦大学周文浩、Xia Mingyang、清华大学米达及许执恒共同通讯在Cell Discovery(IF 34)在线发表题为“Single-cell multi-omics analysis of lineage development and spatial organization in the human fetal cerebellum”的研究论文,该研究对孕周(GW) 13至18周的胎儿样本进行了scRNA-seq、scATAC-seq和空间转录组学分析,以探索发育中的人类小脑细胞多样性和发育程序的出现。该研究鉴定了跨物种保守的瞬时颗粒细胞祖细胞。颗粒细胞和浦肯野细胞的特殊模式被多维解剖。还对小脑叶的物种特异性基因表达模式进行了表征,发现PARM1在人和小鼠颗粒细胞中的分布不一致。在菱形唇上发现了一组新的潜在神经上皮细胞。还分析了浦肯野细胞和单极刷细胞的不同亚型,并揭示了控制它们多样化的基因调控网络。因此,该研究为人类胎儿小脑的多组学研究提供了一个有价值的视角,并促进了人们对人类小脑发育和空间组织的理解。小脑中复杂的神经元类型占人脑神经元总数的80%以上。小脑不同区域的综合神经网络协调各种功能,包括运动、认知、情感和语言。小脑回路的损伤会导致思维或运动的不对称。在自闭症谱系症状或注意力障碍中也观察到小脑皮层灰质减少。小脑皮层的结构在整个进化过程中相对保守。几乎所有脊椎动物的成熟小脑都有三层,即分子层(ML)、浦肯野细胞层(PCL)和颗粒细胞层(GCL)。在白质上方,GCL包含密集颗粒细胞(GCs),被一层浦肯野细胞(PKCs)覆盖。ML由PKCs的树突树和GCs的平行纤维组成。各种中间神经元在不同的层内相互交织,支配着小脑的信号转导。在发育过程中,小脑中的大部分神经元来源于两个生发中心,即心室区(VZ)和上菱形唇(URL)。GABA能谱系包括PKCs和各种中间神经元是由VZ和伯格曼胶质细胞产生的。首先生成PKCs,然后径向迁移并组织到PCL中。兴奋性谱系包括小脑深部核神经元,GCs单极刷细胞(UBCs)来源于URL。当UBC祖细胞定居在URL时,增殖性GC祖细胞切向迁移覆盖小脑表面,形成外颗粒层(EGL)作为次生生发区。由于有丝分裂后的GC在向内迁移后形成了内EGL (iEGL),因此发生了外EGL (oEGL)和GC祖细胞的分离。GCs将继续迁移并穿过PKCs形成内部颗粒层(IGL),最终发展成GCL。UBC祖细胞和UBC亚系的GRN(Credit: Cell Discovery)该研究采用单细胞多组学方法,包括单细胞RNA测序(scRNA-seq)、转座酶可及染色质单细胞测序(scATAC-seq)和空间转录组学,在高时空分辨率下描绘了人类小脑的发育图景。该研究生成了发育中的人类胎儿小脑的大规模多组学图谱。这个全面的图谱揭示了早期人类小脑发育和进化的分子机制。在这项研究中确定的关键调节因子将在体外利用,以产生所需的小脑细胞,用于未来的临床应用。该数据集对于增强人们对神经发育障碍中分子变异和细胞类型之间联系的理解也很重要。原文链接https://www.nature.com/articles/s41421-024-00656-1责编|探索君排版|探索君文章来源|“iNature”End往期精选围观一文读透细胞死亡(Cell Death) | 24年Cell重磅综述(长文收藏版)热文Nature | 破除传统:为何我们需要重新思考肿瘤的命名方式热文Nature | 2024年值得关注的七项技术热文Nature | 自身免疫性疾病能被治愈吗?科学家们终于看到了希望热文Nature | 癌症与神经系统的致命舞蹈
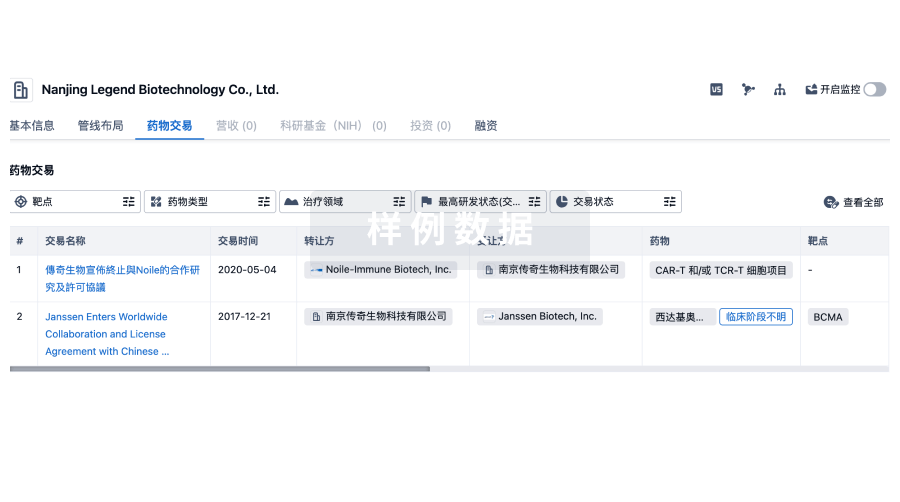
100 项与 Beijing Mida Biomedical Technology Co., Ltd. 相关的药物交易
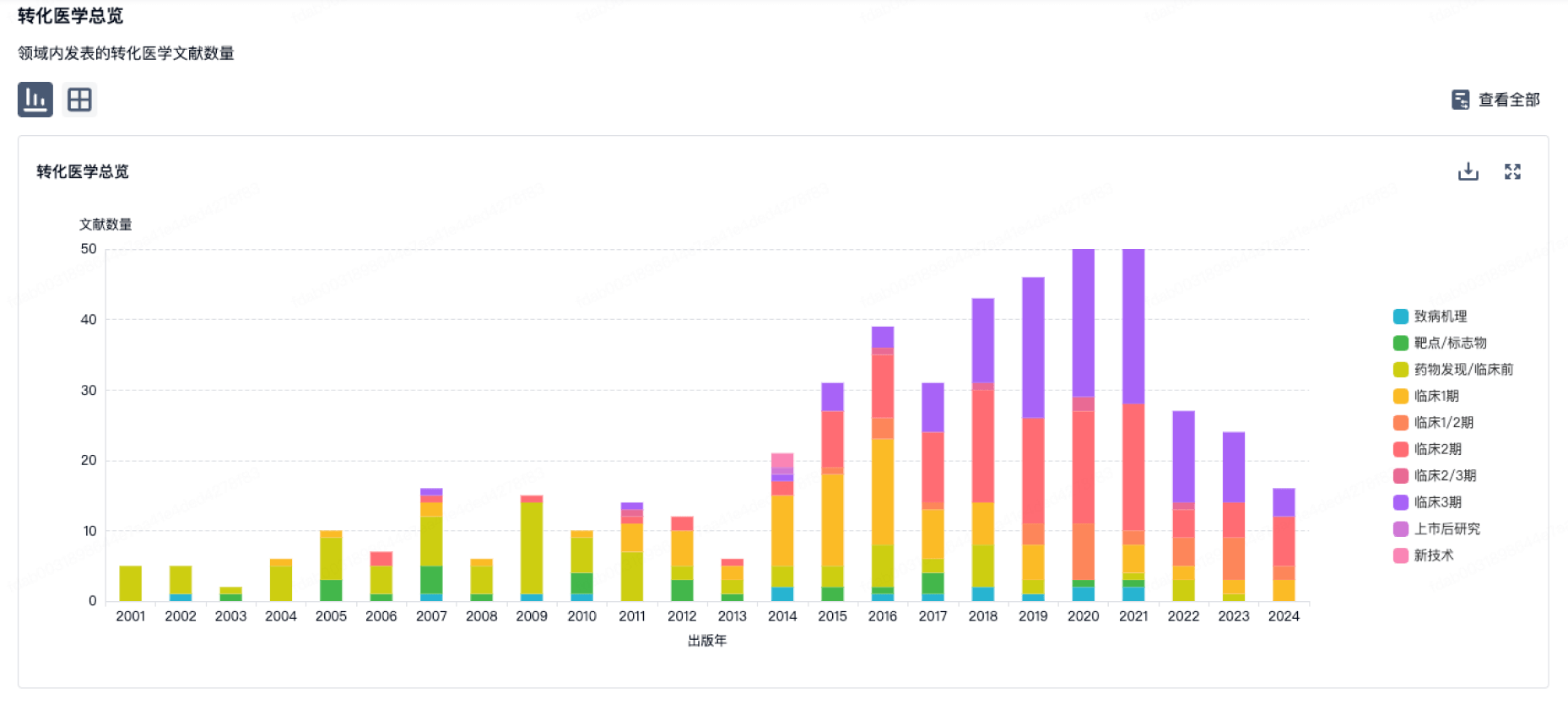
100 项与 Beijing Mida Biomedical Technology Co., Ltd. 相关的转化医学