预约演示
更新于:2026-01-31

Mozhuo Biotechnology (Zhejiang) Co., Ltd.
更新于:2026-01-31
概览
关联
100 项与 墨卓生物科技(浙江)有限公司 相关的临床结果
登录后查看更多信息
0 项与 墨卓生物科技(浙江)有限公司 相关的专利(医药)
登录后查看更多信息
16
项与 墨卓生物科技(浙江)有限公司 相关的新闻(医药)2025-12-30
长文警告!全文12600字,可点赞收藏后细看。
一、什么是单细胞时空多组学?
单细胞时空多组学,指的是从时间与空间两个维度,对细胞内的RNA、蛋白质、代谢物等生物分子进行定性、定位、定量研究的一系列技术总称。
它包括:• 单细胞测序• 空间转录组• 空间代谢组• 空间蛋白组
目前广泛应用于肿瘤学、发育生物学、神经生物学等领域,是生命科学前沿研究方向之一。
二、单细胞时空多组学技术解析
✅ 1.1 单细胞测序定义:高通量单细胞转录组测序,可一次性检测上万个细胞的基因表达谱。应用:揭示细胞异质性、疾病相关亚群、发育轨迹等。常用平台:• 进口:10x Genomics(V4/GEM-X)、BD Rhapsody• 国产:墨卓、华大C4、寻因、新格元等选择建议:• 珍贵样本 → GEM-X• 性价比优先 → 国产优质平台
单细胞测序,更准确的说,是指高通量的单细胞转录组测序,能够对一份组织或血液样本解离制备成单细胞(核)悬液后,利用微流控或微孔板油包水技术,一次性检测上万个细胞的基因表达谱,且每个细胞的基因检出中位数在1千多到5千多不等。经过近10年的发展和应用,目前早已是“飞入寻常百姓家”。由于能够很好的解决bulk RNA-seq无法解决的问题,比如组织样本中的细胞异质性、肿瘤组织中的耐药或敏感的亚克隆、可能发现跟疾病或发育轨迹密切相关的核心细胞亚群,因此在大部分课题组都会涉及到。
目前常用的平台有进口的经典微流控方法的10xGenomics、微孔板原理的BD Rhapsody,以及百花齐放百家争鸣的国产品牌,比如墨卓Mobinova、华大C4、寻因、新格元、M20等等。从文章数量来说,10xGenomics是遥遥领先。从数据质量(捕获细胞数、中位基因值、饱和度等)来说,10x的GEM-X平台也就是V4版本,也属于遥遥领先其他任何平台的地位。对珍贵样本或者重要项目来说,选择GEM-X一定是更加稳妥的方案;对于样本较多、追求性价比而言,国产品牌也是不错的选择,从目前看到的数据来说,墨卓和C4表现还不错。
当然,要注意的一点,GEM-X也有纯原装版本和fake版本,各服务商价格不一,要擦亮眼睛。小红书有个贴子“真假美猴王:单细胞GEM-X实测数据对比”讲得比较清晰。总体来说,可对比看一下服务平台是否多样、是否具备多项授权认证、已发表的项目文章的数量(文章数量跟项目经验一定是成正比的)、价格(价格跟数据一般也是成正比的)、数据指标承诺、售后服务承诺等。
✅ 1.2 空间转录组测序原理:对组织切片中RNA进行高通量检测,实现基因在空间上的定位与表达分析。常用平台:• 10x Genomics(Visium、Xenium)• 华大 Stereo-seq特点:空间分辨率高,可映射细胞类型至空间位置。
空间转录组,主要是将冰冻组织切片或FFPE石蜡切片中的RNA跟玻片上的探针或polydT引物结合后,经过建库测序,实现对组织切片中的基因进行高通量的定性、定量、定位,从而获得切片中不同位置和不同区域的基因表达谱,以解析比如癌和癌旁、脑组织中海马区和皮层区的基因表达差异、找到疾病相关的核心基因。如果通过反卷积等分析,还可以将具体细胞类型映射到空间分布上,得到疾病相关的基因A位于肿瘤B区的细胞C中。常用于肿瘤学、发育生物学、神经生物学等领域的研究。
目前最常用的平台有2个,一个是进口的10xGenomics,包括Visium(polyA法、V1、spot大小55um)、Visium Cytassist(探针法、V2、spot大小55um)和HD(探针法、V3、2um大小)等具体产品。目前最新的版本HD也就是V3能做到2um的分辨率,但一般是bin到8um(也就是近似一个细胞的大小)来做分析。10x还有一个接近单细胞分辨率的大芯片——Xenium 5k原位成像技术,可以覆盖5000个基因,芯片面积较大,可以拼多个样本,虽然单张芯片价格高,但拼样后算下来其实性价比蛮高的。优点是分辨率很高、加上细胞分割后能精确到单细胞水平、还可以拼片,缺点是覆盖基因仅5000个、目前只适合人和小鼠。
另一个是国产的华大Stereo-seq,包括冰冻样本的v1.3版本和针对石蜡样本的v1.1版本,spot的大小是200nm也就是0.2um,分辨率是比较高的。目前这两个品牌算是应用最多的。最初stereo-seq的数据质量确实不咋地,不过经过几轮的更新迭代后,目前实测数据还是不错的,某些指标不亚于老大哥10x了。
这里顺便多提一句华大。华大其实是一个比较大的集团,先不管他是不是褒贬不一。华大分做临床基因检测和NIPT的临检公司华大医学、有做科技服务的华大科技、有专门搞新技术合作发文章的华大研究院、有生产测序仪和stereo-seq空转的华大智造,不管您是要骂还是要夸,可能得先分清具体的对象。
其他平台比如CosMx 6k、Slide-seq、寻因、百迈克,也有一些适用场景。因为了解不多所以在此不做过多评价。
✅ 1.3 空间代谢组又称:质谱成像检测对象:小分子代谢物(70-1000 Da)常用技术:• DESI(电喷雾电离)• MALDI(基质辅助激光解吸电离)• SIMS(二次离子质谱)应用领域:肿瘤代谢、神经代谢、发育代谢等。
空间代谢组也叫做质谱成像(Imaging Mass Spectrometry),跟空间转录组有类似的地方:都是对冰冻组织切片进行检测;但不同点在于,空间代谢组是逐点扫描的方式去高通量的检测70-1000Da的小分子代谢物并对其进行定性、定量和定位。
目前文章中常见的技术有DESI(电喷雾电离技术Desorption ElectroSpray Ionization)、MALDI(基质辅助激光解吸电离Matrix Assisted Laser Desorption Ionization)和SIMS(二次离子质谱Secondary Ion Mass Spectrometry)等3个平台。实际上前两个平台商用较多,两种方法各有特色。一般而言,由于MALDI主要检测质量范围是300Da以上的,而DESI是100Da以上,因此DESI的检测范围更广,如果各类物质都关注的话,可优先选择DESI平台,如果更关注更大分子量的比如脂质的话,可优先选择MALDI平台。
对于研究神经系统疾病、肿瘤代谢重编程、代谢性疾病、生长发育等领域的研究人员而言,空间代谢组能够提供对脑、肾、肝、肿瘤等多组织器官的空间层面的小分子代谢物的分布和重编程,如果再结合单细胞和空间转录组,能够更清晰的知道哪些细胞类型A的哪些代谢物B在什么空间位置C上有何变化,能够更好的揭示疾病发生发展和药物治疗的作用机制。
✅ 1.4 空间蛋白组方法分类:• 基于抗体(如CODEX)• 基于质谱(如LCM-MS)特点:分辨率高(可达0.2 μm),目前主要用于靶向验证,非全蛋白发现。
空间蛋白组目前有基于激光捕获显微切割+ 高灵敏度4D-DIA质谱(LCM-MS)和基于抗体的两大类方法,基于抗体的主要是AKOYA的CODEX方法。
前者是首先通过显微切割获取目的区域,再对切割下来的特定区域样本进行蛋白质谱DIA检测,优点是依赖质谱的蛋白检测方式无偏依可检测数千种蛋白,缺点是非全片扫描,无法形成比较完整的对整张组织切片的全景覆盖。
后者是以CODEX为例的抗体法,是将特定靶标抗体与DNA条形码进行偶联后,与组织切片进行抗体孵育、染色、成像并洗脱,继续下一轮的孵育成像洗脱,实现循环成像。可在单张切片上实现20-60种蛋白的同时检测(最高100多种蛋白),优点是分辨率很高,0.2um,亚细胞级,芯片较大,可以拼多个样本,缺点是panel已定制好,只能从抗体库中选已有的抗体,要添加更多靶标的话会较麻烦且有额外成本,另外AKOYA被Quanterix收购后,整个货期和技术支持不太稳定。类似原理的有COMET平台,可检测28-40种蛋白。其他如IMC质谱成像是通过金属标记的抗体来原位检测,成本较高,定制较困难。
总体而言,空间蛋白组学目前还没有既全片覆盖、又全蛋白覆盖的大而全的技术。要么需要显微切割特定区域、要么需要选择特定的目标蛋白。因此这个技术更多是用于辅助或validate而非discovery。
✅ 1.5 小结
单细胞测序能解决组织中的细胞异质性、疾病相关细胞亚群及关键基因、细胞状态拟时序变化轨迹、细胞间通讯等问题,空间组学能解决组织切片上的RNA、蛋白质、代谢物等分子的空间定位和空间相互作用等问题,单细胞时空多组学联合应用,能从时间、空间、多组学角度更高维度更加全面的揭示疾病和发育等背后的生物学机制。
✅ 1.6 彩蛋:时空新热点-空间组学三维重建
目前在胚胎发育、肿瘤研究中,也陆续开始有空间组学三维重建技术的顶刊文章(4篇Cell如下)发表。简单的说,就是对胚胎、肿瘤、脑组织等样本,进行连续切片后做空间转录组或空间代谢组,最后通过3D建模把连续切片的数据重建为三维模型。3D重构能提供组织显微镜级别的“全息影像”,覆盖切片中心及边缘全部组织、细胞和分子维度,实现多维的时空表达的全景式展现。
空间组学三维重建,可以为相关领域研究者构建一个真正的三维全景空间图谱的基础性数据。但费用也相对比较高。是否有必要开展,需要根据自身研究目的和经费情况评估。仁者见仁智者见智。
三、单细胞时空多组学在高分文章中的应用案例
发表高分文章需要满足众多因素,比如:研究的创新性是属于“技术驱动型”还是“概念突破型”、机制研究的深度如何、样本量与统计效力是否足够、数据整合与分析复杂度是否达到顶级标准、领域竞争是否激烈是否已有同类研究发表、图表与可视化是否达到顶级期刊美学标准、语言与叙事逻辑是否完善。尤其是顶刊更看重的是这项研究是否有突破性的或创新性的新发现。
作为相对前沿和“高大上”的技术,单细胞时空多组学也确实通常能为我们的研究锦上添花。下面我们看几篇单细胞时空多组学技术在高分文章中是如何应用的。
��案例一 | Cell(2025)• 标题:肿瘤产生的氨被调节性T细胞代谢以进一步阻碍抗肿瘤免疫• 技术:空间转录组 + 空间代谢组• 亮点:揭示Tregs代谢氨促进免疫耐药,靶向GLUD1可克服PD-1耐药
2025年12月24日,徐州医科大学附属医院吕凌教授与南京医科大学第一附属医院的古鉴教授研究团队相关研究成果以“Tumor-produced ammonia is metabolized by regulatory T cells to further impede anti-tumor immunity”为题,被国际顶级期刊Cell接收发表。作者通过空间多组学系统揭示Tregs细胞的代谢特征,研究了肿瘤来源氨被Tregs细胞代谢利用,从而进一步抑制抗肿瘤免疫并促进免疫治疗耐药的关键机制。该研究使用了欧易生物的空间转录组和空间代谢组等单细胞时空多组学技术。
该研究首先通过对6例人肝细胞癌(HCC)患者的肿瘤及癌旁组织进行 10xGenomics Visium CytAssist(V2)空间转录组测序 和 AFADESI-MSI空间代谢组学检测以及,将空转数据(spots)与空代数据(pixels)进行点对点共定位联合分析,发现调节性T细胞Tregs在肿瘤内部显著富集。通过KEGG富集分析发现Tregs富集的肿瘤亚区域具有独特的 “高谷氨酰胺分解、低尿素循环活性” 的代谢特征,这就导致谷氨酰胺分解的主要副产物“氨”在局部积累。结合鼠源原位肝癌模型、人结肠癌和肺癌等多种肿瘤模型发现氨水平与Tregs丰度呈正相关。提示肿瘤微环境中的氨积累是驱动Tregs空间富集的关键代谢因素。
面对肿瘤内部这种极端高氨环境,Tregs是如何感知和解氨毒、是如何生存和适应、会起到什么样的作用呢?
于是作者设计并通过一系列严密且详实的实验(如单细胞测序、ChIP、CUT&Tag、代谢组、代谢流追踪、化学蛋白质组学、SPR和ITC等蛋白质互作、X射线晶体/结构生物学),发现Tregs一方面是通过 “SRC3/STAT3-ASL”尿素循环轴解毒氨以求生存,同时另一方面又通过 “FOXP3/SMS/精胺/PPARγ”代谢轴利用氨以增强OXPHOS和免疫抑制功能,从而形成了完整的适应环路。
那么,上述机制在肿瘤免疫治疗中又有何临床意义?能否找到新靶点?如何更好的用于临床治疗?
作者在接受抗PD-1新辅助治疗后手术的肝癌HCC患者样本中发现,治疗后的肿瘤组织氨水平升高且Tregs浸润增多。结合多种HCC肝癌模型实验,最后发现:抗PD-1治疗通过诱导肿瘤细胞死亡→增加氨产生→强化Treg功能→导致耐药,而靶向氨生成通路(特别是GLUD1)是克服免疫检查点抑制剂耐药的有效策略。这对PD-1治疗耐药机制和未来联合用药思路提供了很好的方向。
更多详细解读,可参考:https://mp.weixin.qq.com/s/g6zE_8y37sv8C_7ak1pSgg
��案例二 | Nature(2025)• 标题:溶瘤病毒VG161在难治性肝细胞癌中的应用• 技术:单细胞转录组 + 单细胞免疫组库 + 空间转录组• 亮点:首次人体试验,构建疗效预测模型,揭示免疫重塑机制
2025年3月19日,浙江大学梁廷波团队在Nature(IF=50.5)上发表题为“Oncolytic virus VG161 in refractory hepatocellular carcinoma”的研究论文。作者发现,溶瘤病毒VG161在难治性肝细胞癌患者中表现出良好的安全性和显著的抗肿瘤活性,有望成为三线治疗难治性肝细胞癌的有效选择,并为溶瘤病毒免疫疗法的发展提供了新的方向。该研究使用了欧易生物的单细胞转录组、单细胞免疫组库及空间转录组等单细胞时空多组学技术。
研究亮点:
u机制协同:VG161是全球首个在同一病毒载体中整合多种免疫调节因子(IL-12/15/15Rα + PD-1/PD-L1阻断)的溶瘤病毒,旨在协同激活固有和适应性免疫,同时解除免疫抑制。
u靶向难治人群:研究针对的是标准治疗(包括靶向和免疫治疗)失败后的晚期肝癌患者,满足了迫切的临床需求。
u首次人体试验:这是VG161在肝癌领域的首次临床试验报告,证明了其在难治性HCC患者中的安全性、初步疗效和独特的免疫激活能力。
u疗效预测模型:利用转录组数据构建了首个针对溶瘤病毒治疗肝癌的疗效预测模型,为精准治疗和患者筛选提供了潜在工具。
u深入机制解析:综合运用了单细胞测序、单细胞免疫组库、空间转录组等多组学技术,深入揭示了VG161重塑肿瘤免疫微环境、诱导远隔效应的分子和细胞机制。
更多详细解读,可参考:https://mp.weixin.qq.com/s/zCLQSSo8uSPhjZjmhUmpTQ
��案例三 | Advanced Materials(2025)• 标题:蒿舒酯纳米平台靶向CAF逆转三阴性乳腺癌光热抵抗• 技术:单细胞测序 + 空间转录组 + 空间代谢组• 亮点:揭示CAF介导的丝氨酸代谢-MAPK轴驱动耐药机制
2025年6月17日,复旦大学附属肿瘤医院周世崇/常才/郑东东、芬兰埃博学术大学张宏博团队在Advanced Materials(IF=27.4)上发表了题为“Artesunate Nanoplatform Targets the Serine-MAPK Axis in Cancer-Associated Fibroblasts to Reverse Photothermal Resistance in Triple-Negative Breast Cancer”的研究论文,深入探究了肿瘤相关成纤维亚群ECM CAFs通过丝氨酸代谢-MAPK轴介导光热疗法耐药的作用机制,为突破吲哚菁绿(ICG)介导PTT耐药,促进三阴性乳腺癌的临床治疗提供了新的策略。该研究使用了欧易生物的单细胞测序、空间转录组、空间代谢组等单细胞时空多组学技术以及转录组/蛋白组/代谢组等传统多组学。
更多详细解读,可参考:https://mp.weixin.qq.com/s/XxWJYmGm6txvFTg_vwgDxQ
��案例四 | Nature Genetics(2024)• 标题:透明细胞肾细胞癌的多组学分析• 技术:单细胞多模态 + 空间转录组 + 空间代谢组• 亮点:提出新型分子分型策略,指导临床治疗
2024年2月15日,华中科技大学附属同济医院谌科教授,陶振教授,管维教授和海军军医大学附属长征医院任善成教授合作在Nature Genetics(IF:30.8)在线发表题为“Multi-omic profiling of clear cell renal cell carcinoma identifies metabolic reprogramming associated with disease progression”的研究论文。该研究基于ccRCC肾癌大队列转录组、蛋白组、代谢组、空间转录组、空间代谢组等多组学数据以及公共数据,揭示了ccRCC的进展机制,并提出一种新型分子分型诊疗策略,可以用来指导ccRCC的临床治疗。该研究使用了欧易生物的单细胞多模态、空间转录组、空间代谢组等单细胞时空多组学技术。
更多详细解读,可参考:https://mp.weixin.qq.com/s/CAbpR51GrdDXTFbyuVzg2Q
��案例五 | Nature Communications(2023)• 标题:胃癌细胞特异性代谢重塑与互作• 技术:空间代谢组 + 空间脂质组 + 空间转录组• 亮点:国内首篇空间代谢+空间转录联合分析文章
2023年5月,中国医学科学院药物研究所贺玖明与齐鲁工业大学(山东省科学院)孙成龙、北京大学肿瘤医院季加孚/步召德、上海市生物医药技术研究院戴文韬等多个课题组密切合作,在Nature Communications(IF=17.694)发表题为“Spatially resolved multi-omics highlights cell-specific metabolic remodeling and interactions in gastric cancer”。该工作在空间分辨多组学新技术研发及胃癌代谢重编程及交互作用研究方面取得重要进展,并得到了上海欧易生物和上海鹿明生物的有力支持。本文提出了一种整合空间分辨多组学(空间代谢组+空间脂质组+空间转录组)方法,以探索胃癌微环境中的细胞特异性代谢重塑和相互作用。
这是国内第一篇联合应用空间转录组和空间代谢组的空间多组学文章。
更多详细解读,可参考:https://mp.weixin.qq.com/s/JHdHwdCLi0hV6GHcrn6wYg
��案例六 | Cell Reports Medicine(2023)• 标题:人脑损伤中的代谢异质性• 技术:单细胞测序 + 空间转录组 + 空间代谢组• 亮点:国内首篇单细胞+空转+空代三技术联用文章
2023年5月31日,上海市浦东新区人民医院神经外科郑平教授在Cell Reports Medicine期刊(IF:16.988)发表题为“Integrated spatial transcriptome and metabolism study reveals metabolic heterogeneity in human injured brain”的研究成果。文章对创伤性脑损伤患者的大脑样本进行了空间多组学分析,发现了一个受伤神经元的脂质过氧化区域,以及不平衡的肌醇和肌醇磷酸酯以及相关的空间标记。结果突出了受伤大脑中复杂的转录组调节和代谢改变,并将直接促成设计试剂,以针对人脑中的特定基因进行功能分析。该研究使用了欧易生物的单细胞测序、空间转录组、空间代谢组等单细胞时空多组学技术。
这是国内第一篇联合应用单细胞、空间转录组和空间代谢组三个技术的文章。
更多详细解读,可参考:https://mp.weixin.qq.com/s/LpTY6Gcm9GO3jFEO384O9Q
��案例七 | Advanced Science(2024)• 标题:口腔鳞癌的肿瘤微环境空间图谱• 技术:空间转录组 + 空间代谢组• 亮点:揭示多胺代谢在免疫逃逸中的作用
2024年1月16日,中南大学湘雅医学院附属肿瘤医院曾朝阳、熊炜及湘雅二医院口腔医学中心龚朝建团队课题组在Advanced Science(IF:15.1)上发表“Spatial Transcriptomic and Metabolomic Landscapes of Oral Submucous Fibrosis-Derived Oral Squamous Cell Carcinoma and its Tumor Microenvironment”的研究论文。该研究通过先进的空间转录组和空间代谢组技术平台揭示了口腔鳞状细胞癌患者通过原位癌-上皮间充质化-成纤维化途径促进肿瘤恶性进展。另外,发现异常的多胺代谢,在促进肿瘤发生和免疫逃避中发挥关键作用。本研究的空间多组学数据为OSCC的机制提供了新的思路,也为预防和治疗OSCC提供了宝贵的线索。该研究使用了欧易生物的空间转录组、空间代谢组等单细胞时空多组学技术。
更多详细解读,可参考:https://mp.weixin.qq.com/s/OQbKr7mMDSAHOwRFTkvB6w
��案例八 | Advanced Science(2025)• 标题:中性粒细胞胞外陷阱促进肺癌脑转移• 技术:单细胞测序 + 空间转录组 + 空间代谢组• 亮点:发现LOX+恶性细胞与NETs共定位形成“转移龛”
这篇中南大学湘雅医院团队于2025年10月发表在Advanced Science的“Neutrophil Extracellular Trap Reprograms Cancer Metabolism to Form a Metastatic Niche Promoting Non-Small Cell Lung Cancer Brain Metastasis”文章,探讨了非小细胞肺癌(NSCLC)脑转移(BMs)的机制,发现了一种特殊的恶性细胞亚群——LOX+ Malig-5,这类细胞作为转移起始细胞(MICs),与特定释放中性粒细胞胞外陷阱(NETs)的中性粒细胞亚群(Neutro-0 和 Neutro-3)共定位并相互作用,共同形成了一个促转移微环境(转移龛)。该研究使用了欧易生物的单细胞测序、空间转录组、空间代谢组等单细胞时空多组学技术。
一、该研究揭示了以下机制轴:
uLOX+ Malig-5 通过 CXCL8-CXCR2 招募 Neutro-0/Neutro-3;
uNeutro-0/Neutro-3 释放 NETs;
uNETs 通过与肿瘤细胞表面的 KRT10 结合,促进上皮-间质转化(EMT);
u该微环境中代谢重编程显著,棕榈酸(palmitic acid)水平显著升高,进一步驱动转移;
u靶向棕榈酸合成的 FASN 抑制剂 TVB-2640 在小鼠模型中能抑制转移。
二、研究亮点(Research Highlights)
u识别了 NSCLC 脑转移的关键起始细胞:LOX+ Malig-5 作为 MICs,具有高干性、EMT 状态与转移潜能。
u揭示了 NETs 与肿瘤细胞的直接作用机制:NET-KRT10 信号轴介导了中性粒细胞与肿瘤细胞的相互作用,促进 EMT 与转移。
u构建了“转移龛”的空间代谢图谱:首次通过空间多组学技术揭示了转移龛中棕榈酸代谢显著上调。
u提出了新的治疗靶点与药物:FASN 抑制剂 TVB-2640 通过抑制棕榈酸合成,在小鼠模型中显著抑制 NSCLC 脑转移。
u跨队列验证与AI辅助药物筛选:结合湘雅医院与玛丽医院队列数据,使用机器学习算法识别关键代谢物,并进行虚拟药物筛选。三、技术路线:步骤 1:多组学数据获取与细胞图谱构建
技术:单细胞转录组测序(scRNA-seq)、空间转录组、空间代谢组、蛋白质组、TCGA 批量 RNA-seq。
样本:湘雅医院 9 例 NSCLC 患者(4 例原发灶 + 5 例脑转移灶);玛丽医院 25 例患者血液代谢数据。
结论:
构建了 NSCLC 原发灶与脑转移灶的单细胞图谱,识别出 11 种主要细胞类型(Fig. 1b–d)。
恶性细胞通过 CNV 分析被区分出来(Fig. 1e–f)。
免疫细胞组成在转移与非转移样本中存在差异(Fig. 1g–h)。步骤 2:识别转移起始细胞(MICs)
技术:CytoTRACE、Monocle2、RNA velocity、扩散图、PyVIA 等伪时间分析。
结论:
Malig-5 细胞具有最高干性与起始分化能力(Fig. 2c–e)。
被标记为LOX+ Malig-5,其 LOX 表达与 EMT 状态正相关(Fig. 2k, Fig. S5)。
在 TCGA 数据中,LOX+ Malig-5 高表达与患者预后不良显著相关(Fig. 2i–j)。步骤 3:识别共定位的中性粒细胞亚群
技术:空间共定位分析(misty)、Cell2location 空间解卷积。
结论:
LOX+ Malig-5 与 Neutro-0 和 Neutro-3 高度共定位(Fig. 3b–c, e)。
这两个中性粒细胞亚群具有高 NET 释放能力(Fig. 3j)。
在转移患者中比例更高,可预测转移(Fig. 3f–g)。步骤 4:揭示 NET-KRT10 相互作用机制
技术:Pull-down 实验 + 质谱、细胞共培养、脑类器官模型、Transwell 实验。
结论:
KRT10 是 NETs 在肿瘤细胞上的主要结合蛋白(Fig. 4a–b)。
NET-KRT10 信号轴促进肿瘤细胞 EMT 与迁移(Fig. 4h–k)。
LOX+ Malig-5 通过 CXCL8-CXCR2 招募中性粒细胞(Fig. S11)。步骤 5:构建“转移龛”并分析其代谢特征
技术:空间代谢组、代谢通路富集分析(MetaboAnalyst)、基因-代谢物互作网络。
结论:
LOX+ Malig-5 + Neutro-0/Neutro-3 形成转移龛(Fig. 5a–b)。
转移龛中棕榈酸代谢显著上调(Fig. 5c–g)。
棕榈酸水平与 EMT/NET 评分正相关(Fig. 5i–j)。步骤 6:识别关键代谢物与治疗药物
技术:机器学习(Boruta、Random Forest、XGBoost)、虚拟药物筛选、分子对接、小鼠尾静脉注射模型。
结论:
棕榈酸是转移龛的关键代谢驱动物(Fig. 6a–c)。
TVB-2640 是抑制 FASN-ERD 的潜在药物,在小鼠模型中抑制转移(Fig. 6k–m, Fig. S12, S14)。
��案例九 | Molecular Therapy(2025)• 标题:SPP1+成纤维细胞驱动结直肠癌肝转移• 技术:单细胞测序 + 空间转录组 + 空间代谢组• 亮点:揭示CAF代谢异质性及其促转移机制
这篇浙江大学医学院团队于2025年5月发表在Molecular Therapy(IF=12)的“Spatial multi-omics reveals the potential involvement of SPP1+ fibroblasts in determining metabolic heterogeneity and promoting metastatic growth of colorectal cancer liver metastasis”文章,该研究利用前沿的空间多组学技术,深入解析了结直肠癌肝转移CRLM这一致命转移过程的肿瘤微环境空间结构和代谢特征,该研究揭示了CRLM中一个由特定成纤维细胞亚群(SPP1+ CAFs)驱动的、具有免疫抑制和代谢重编程特征的促转移微环境。靶向 SPP1-CD44 相互作用或利用其特异性代谢物(辛二酸、四甘醇)作为诊断标志物或治疗靶点,有望为CRLM的治疗提供新策略。该研究使用了欧易生物的单细胞测序、空间转录组、空间代谢组等单细胞时空多组学技术。该研究亮点总结:
u创新性技术整合:首次将空间转录组学、空间代谢组学和单细胞RNA测序 技术联合应用于CRLM研究,实现了在保留空间位置信息的前提下,对基因表达、代谢活动和细胞异质性进行全方位解析。
u揭示空间功能微域:不仅识别了CRLM中的不同细胞区域,更精准定位了一个富含CAFs的微结构域,并证实该区域与患者不良预后密切相关,是肿瘤进展和免疫抑制的关键空间单位。
u锁定关键细胞亚群:在复杂的CAFs中,利用单细胞分辨率鉴定出在肝转移中特异富集的 SPP1+ 成纤维细胞亚群,并明确了其与肿瘤细胞的直接通信桥梁。
u阐明核心分子机制:首次在空间上证实了 SPP1-CD44轴 在CRLM中作为成纤维细胞与肿瘤细胞间通信的关键通路,为开发阻断性疗法提供了直接靶点。
u发现代谢标志物:结合空间代谢组学,发现了该促转移微环境特有的两种小分子代谢物——辛二酸和四甘醇,并验证了其在独立队列中对肝转移的预测价值,为无创诊断提供了新思路。
��案例十 | Theranostics(2025)• 标题:鼻咽癌代谢重编程与PD-1表达关联• 技术:空间转录组 + 空间代谢组• 亮点:构建6基因预测模型,AUC达0.964
武汉大学中南医院团队于2025年2月发表于《Theranostics》的题为“Spatial Metabolomics and Transcriptomics Reveal Metabolic Reprogramming and Cellular Interactions in Nasopharyngeal Carcinoma with High PD-1 Expression and Therapeutic Response”的文章,通过空间多组学技术,揭示了NPC治疗反应中6个关键分子的核心作用,为其作为预后生物标志物和治疗靶点的潜在应用提供了有力证据,有助于制定旨在改善患者预后的精准肿瘤学策略。该研究使用了欧易生物的空间转录组和空间代谢组等单细胞时空多组学技术。
鼻咽癌(NPC)是一种异质性强的癌症,患者治疗反应差异显著,因此亟需深入理解影响其疗效的分子机制。本研究旨在探索具有不同治疗反应和PD-1表达水平的NPC患者的空间代谢与基因表达变化。
方法: 本研究采用空间代谢组学(SM)和空间转录组学(ST)技术,探究对治疗敏感或具有高程序性死亡受体1(PD-1)表达的NPC患者中显著的代谢通路和代谢物改变。同时,研究了肿瘤微环境中不同细胞类型的空间分布及其复杂的相互作用。利用TCGA等公共数据集对识别出的预后靶点进行了验证,并通过体外功能分析进一步证实。
结果: SM分析揭示了脂质代谢、支链氨基酸(BCAA)代谢和谷氨酰胺代谢的重编程,这些代谢变化与治疗反应和PD-1表达密切相关。ST分析强调了前体T细胞与恶性上皮细胞之间的相互作用在调控NPC治疗反应中的关键作用。值得注意的是,本研究识别出参与BCAA代谢的六个关键基因(IL4I1, OXCT1, BCAT2, DLD, ALDH1B1, HADH),可用于区分治疗敏感患者和耐药患者。对DLD和IL4I1的功能验证表明,基因沉默能显著抑制NPC细胞的增殖、克隆形成、伤口愈合和侵袭能力,并诱导细胞周期阻滞。免疫组化分析进一步证实,这六个基因的高表达与NPC患者的不良预后显著相关,这一趋势也在TCGA头颈癌队列中得到证实。
该研究亮点总结:
u多组学整合与空间解析:首次在NPC中整合空间代谢组学(SM)与空间转录组学(ST),实现了代谢物与基因在组织原位的高分辨率空间关联分析。
u关联临床表型:将空间多组学数据与治疗反应(敏感/耐药) 和 PD-1表达水平(高/低) 直接关联,为理解疗效差异的机制提供了新视角。
u识别关键通路与基因:系统揭示了BCAA代谢、谷氨酰胺代谢和脂质代谢在NPC治疗反应中的核心作用,并筛选出6个关键的BCAA代谢相关基因作为潜在生物标志物。
u解析肿瘤微环境互作:利用ST数据深入分析了恶性上皮细胞、前体T细胞、巨噬细胞、成纤维细胞等关键细胞类型的空间分布、相互作用及与疗效的关系。
u构建高精度预测模型:基于6个关键基因构建的预测模型,在区分治疗敏感与耐药样本方面展现出极高的AUC值(0.964),临床转化潜力显著。
u多层次验证:结合公共数据库(GEO, TCGA)、独立样本验证(IHC、TMA)、以及体外功能实验(基因敲降、细胞功能学实验),形成了完整的证据链。
��案例十一 | Journal of Advanced Research(2025)• 标题:宫颈癌糖酵解抑制剂靶向递送系统• 技术:空间转录组 + 空间代谢组• 亮点:基于ASCT2靶点的纳米递送系统逆转化疗耐药
2025年11月发表于《Journal of Advanced Research》的题为“Spatial multi-omics guides ASCT2-targeted delivery of glycolysis inhibitor for cervical cancer suppression and chemoresistance reversal”文章,提出了一种基于空间多组学指导的靶向糖酵解抑制剂递送策略,能够显著抑制宫颈癌并逆转其化疗耐药性。该研究使用了欧易生物的空间转录组和空间代谢组等单细胞时空多组学技术。
宫颈癌死亡率居高不下,化疗耐药是主要原因。靶向糖酵解重编程是一个有前景的策略,因为宫颈癌细胞依赖增强的糖酵解进行增殖和耐药。然而,临床转化面临两大障碍:缺乏对宫颈癌标本中糖酵解脆弱性的空间解析验证,以及缺乏针对洛尼达明(Londamine, LND)的宫颈癌特异性递送系统。
该研究采用空间代谢组学与空间转录组学分析临床宫颈癌样本,验证糖酵解活性并识别特异性高表达的表面标志物。研究发现,宫颈癌恶性区域中糖酵解活性上调,并识别出谷氨酰胺转运蛋白ASCT2作为优于CD44的宫颈癌特异性靶点。基于此,设计了一种谷氨酰胺功能化的脂质体系统,用于靶向递送LND。该纳米系统能有效抑制宫颈癌生长,并通过抑制MRP2介导的顺铂外排和阻断核糖-5-磷酸介导的DNA修复,逆转顺铂耐药。
��案例十二 | Developmental Cell(2025)• 标题:胶质瘤CHI3L1/SPP1正反馈环路驱动免疫抑制• 技术:单细胞测序 + 空间转录组• 亮点:发现潮霉素B可阻断STAT3通路抑制肿瘤
2025年发表在《Developmental Cell》的这篇题为 “STAT3-controlled CHI3L1/SPP1 positive feedback loop demonstrates the spatial heterogeneity and immune characteristics of glioblastoma” 的文章,该研究聚焦于胶质母细胞瘤(GBM)中 CHI3L1(壳三糖酶-3样蛋白1) 与 SPP1(骨桥蛋白,即OPN) 通过 STAT3 调控形成的正反馈环路,揭示了GBM的空间异质性和免疫微环境特征。主要发现包括:
lCHI3L1 通过直接结合STAT3的卷曲螺旋结构域(CCD),促进STAT3的磷酸化、核转位和转录活性。
lSPP1 通过其受体 ITGB1 激活 NF-κB 和 STAT3 通路,进一步上调CHI3L1表达,形成一个正反馈环路,促进胶质瘤从原神经型向间充质型(PMT) 的恶性转变。
l巨噬细胞与间充质型(MES)细胞在空间上紧密相关,共同高表达CHI3L1和SPP1,形成肿瘤-免疫细胞通讯网络。
l筛选出一种STAT3-CCD结构域抑制剂——潮霉素B(Hygromycin B, HB),可阻断CHI3L1-STAT3相互作用,在体内外模型中显著抑制肿瘤生长和巨噬细胞重编程。
该研究使用了欧易生物的单细胞测序、空间转录组等单细胞时空多组学技术。
研究亮点总结:
l揭示了CHI3L1-STAT3-SPP1正反馈环路在GBM恶性进展中的核心作用。
l阐明了巨噬细胞与肿瘤细胞在空间上的互作模式,解释了GBM免疫抑制微环境的形成机制。
l首次发现CHI3L1直接结合STAT3的CCD结构域,而非传统的SH2结构域,为靶向治疗提供了新思路。
l鉴定出潮霉素B作为STAT3-CCD抑制剂,具有双重抗肿瘤效应:抑制肿瘤自身恶性进展 + 阻止巨噬细胞M2型极化。
l融合单细胞测序、空间转录组学与多组学分析,系统解析了GBM的细胞异质性与细胞间通讯网络。
��案例十三 | Cardiovascular Diabetology(2024)• 标题:糖尿病大血管病变平滑肌表型转化图谱• 技术:单细胞测序 + 空间转录组 + 空间代谢组• 亮点:构建首个糖尿病血管空间多组学图谱
2024年发表在《Cardiovascular Diabetology》题为 “Spatial multiomics atlas reveals smooth muscle phenotypic transformation and metabolic reprogramming in diabetic macroangiopathy” 的文章,该研究针对糖尿病大血管病变,通过单细胞转录组、空间转录组、空间代谢组多组学技术,系统揭示了胫前动脉在糖尿病状态下的细胞组成、空间异质性、代谢重编程及平滑肌细胞表型转化的分子机制。该研究使用了欧易生物的单细胞测序、空间转录组、空间代谢组等单细胞时空多组学技术。
一、主要发现包括:
l构建了首个糖尿病血管空间多组学图谱,涵盖基因与代谢物的空间分布。
l平滑肌细胞(SMCs) 具有三种表型转化趋势:炎症趋化型、巨噬细胞样/泡沫样型、成纤维细胞/软骨细胞样型。
l转录因子网络(如KDM5B、DDIT3等)驱动SMCs表型转化。
l代谢激活网络显示糖尿病血管中多种代谢通路被激活,如花生四烯酸代谢、不饱和脂肪酸合成等。
l代谢重编程关键基因:CYP27A1与HNMT在糖尿病血管中表达更广泛。
l钙化区域富集异环化合物,并激活叶酸合成通路。
二、研究亮点
l首次构建糖尿病血管“基因-代谢-空间”三位一体多组学图谱。
l系统揭示SMCs表型转化的三种趋势及其转录调控网络。
l整合空间代谢组揭示糖尿病血管代谢激活与重编程。
l发现钙化周边区域存在核富集区,伴随炎症与代谢通路激活。
l提出CYP27A1与HNMT作为糖尿病血管代谢重编程的潜在标志物。
四、全文小结
单细胞时空多组学技术通过整合时间、空间与多维度数据,为疾病机制、发育过程、免疫微环境等研究提供了前所未有的系统性视角。随着技术迭代与成本优化,其在高水平研究中的应用将更加广泛。
五、参考文献(案例1-13):
1.Lyu, L. & Gu, J. et al. Tumor-produced ammonia is metabolized by regulatory T cells to further impede anti-tumor immunity. Cell (2025).
2.Shen, Y., Bai, X., Zhang, Q. et al. Oncolytic virus VG161 in refractory hepatocellular carcinoma. Nature (2025). https://doi.org/10.1038/s41586-025-08717-5
3.Zhou, S., Zhang, H., Chang, C. et al. Artesunate nanoplatform targets the serine-MAPK axis in cancer-associated fibroblasts to reverse photothermal resistance in triple-negative breast cancer. Adv. Mater. (2025).
4.Chen, K., Tao, Z., Guan, W. et al. Multi-omic profiling of clear cell renal cell carcinoma identifies metabolic reprogramming associated with disease progression. Nat. Genet. 56, 308–320 (2024). https://doi.org/10.1038/s41588-023-01651-0
5.He, J., Sun, C., Ji, J. et al. Spatially resolved multi-omics highlights cell-specific metabolic remodeling and interactions in gastric cancer. Nat. Commun. 14, 2699 (2023). https://doi.org/10.1038/s41467-023-38397-6
6.Zheng, P. et al. Integrated spatial transcriptome and metabolism study reveals metabolic heterogeneity in human injured brain. Cell Rep. Med. 4, 101208 (2023). https://doi.org/10.1016/j.xcrm.2023.101208
7.Zeng, C., Xiong, W., Gong, C. et al. Spatial transcriptomic and metabolomic landscapes of oral submucous fibrosis-derived oral squamous cell carcinoma and its tumor microenvironment. Adv. Sci. 11, e2305780 (2024). https://doi.org/10.1002/advs.202305780
8.Li, M., Xie, W. et al. Neutrophil extracellular trap reprograms cancer metabolism to form a metastatic niche promoting non-small cell lung cancer brain metastasis. Adv. Sci., e2502100 (2025).
9.Wang, Z., Liang, T. et al. Spatial multi-omics reveals the potential involvement of SPP1+ fibroblasts in determining metabolic heterogeneity and promoting metastatic growth of colorectal cancer liver metastasis. Mol. Ther. 33, 1234-1248 (2025).
10.Chen, Y., Yuan, Y. et al. Spatial metabolomics and transcriptomics reveal metabolic reprogramming and cellular interactions in nasopharyngeal carcinoma with high PD-1 expression and therapeutic response. Theranostics 15(3), 1123-1141 (2025).
11.Liu, X., Zhang, Y. et al. Spatial multi-omics guides ASCT2-targeted delivery of glycolysis inhibitor for cervical cancer suppression and chemoresistance reversal. J. Adv. Res., In press (2025).
12.Xu, H., Wang, J. et al. STAT3-controlled CHI3L1/SPP1 positive feedback loop demonstrates the spatial heterogeneity and immune characteristics of glioblastoma. Dev. Cell 60(5), 567-580 (2025).
13.Zhang, L., Liu, Q. et al. Spatial multiomics atlas reveals smooth muscle phenotypic transformation and metabolic reprogramming in diabetic macroangiopathy. Cardiovasc. Diabetol. 23, 115 (2024).
如需技术交流或者需要全文,可联系作者如下二维码(请备注单位名字)。
声明:本文仅代表作者个人观点,作者水平有限,如有不科学之处,请在下方留言指正!部分内容来自网络,侵删。
2025-12-15
·今日头条
2025年12月,首批“浙江省科技新小龙”企业名单正式公布,共96家新锐科技企业入选。作为省科技厅、新华社浙江分社等多部门指导的“科技新小龙探访和护航行动”核心成果,此次评选旨在发掘培育具有核心技术突破能力、成长性强的科技企业,为全省创新驱动发展注入新动能。

1. 评选的核心逻辑:新锐、高成长、强研发
“科技新小龙”的评选以“硬指标”筛选企业创新与潜力:企业需注册3年以上、原则上不超过10年(若有原创性、颠覆性技术可适当放宽);近3年营收或估值年增长率不低于30%,体现高成长性;近3年研发投入占比不低于10%或年度研发投入超1000万元,彰显研发强度。三项标准共同构成对企业创新成色的系统评估。
2. 区域与产业:前沿赛道的集群化突破
从区域看,余杭以18家入选居全省第一,覆盖新一代信息技术、脑科学与类脑智能等六大领域;绍兴6家、嘉兴7家、金华5家、宁波12家紧随其后,形成“核心区引领、多地协同”格局。从产业看,企业集中在战略性新兴领域:

- 新一代信息技术领域:福瑞泰克(智能驾驶)、赛思电子(时频同步芯片)等;
- 新能源领域:钠创新能源(钠离子电池材料)、致德新能源(新能源材料)等;
- 生物医药领域:新码生物(抗肿瘤ADC药物)、墨卓生物(单细胞测序)等;
- 新材料领域:航引新材料(碳化硅陶瓷基复合材料)、奥氏芯材(分离膜材料)等。
3. 企业样本:从技术突破到产业落地
多家企业已实现技术突破与产业落地的衔接:
- 嘉兴福瑞泰克:智能驾驶解决方案2024年占国内L2级市场21.3%份额,合作51家主机厂,量产项目超290个,核心产品搭载200多万台汽车;

- 桐乡墨卓生物:自主研发单细胞测序系统,打破“卡脖子”技术,稳居国内科研市场国产厂商前列;
- 绍兴新码生物:首个抗肿瘤ADC药物完成临床Ⅲ期,计划2025年申报上市;双抗ADC药物对叶酸受体低表达卵巢癌疗效显著,预计2026年启动临床试验;
- 义乌创豪半导体:专注集成电路高阶封装基板,攻克精细线路制作等技术,项目投产后有望打破国外垄断。
4. 护航机制:从“新小龙”到“巨龙”的培育链路
入选企业将获得“全周期护航”:对接省实验室、双一流高校等高能级科创平台的“技术直通车”;“一对一”专家团辅导创新战略;联动金融机构提供专项金融支持;建立“创新成长档案”跟踪研发、专利等指标,3年后汇编《从新小龙到独角兽》案例集。这些支持将助力企业从“技术突破”走向“产业引领”。
抗体药物偶联物上市批准申请上市
2025-11-09
2025年11月8日,作为本届世界互联网大会的系列活动之一,由乌镇镇人民政府、墨卓生物科技(浙江)有限公司、上海欧易生物医学科技有限公司主办,中国生物物理学会微流控系统分会、乌镇大数据高新技术产业园区联合主办;转化医学网、乌镇院士之家、祥符实验室协办,以“从数字世界到生命健康:多技术融合构建生命科学未来”为主题的研讨会,在大会分会场成功召开。乌镇镇党委副书记、镇长张雪峰出席会议。
当下,数字科技作为时代发展的新引擎,正在全面重构生命科学的发展方式。在这一过程中,人工智能、强大的算力与海量数据相互融合,不断激发出新的生命健康技术和商业发展模式。随着数字技术的全方位渗透和跨界融合,我们正在见证生命科学未来发展的新质生产力在不断地融合出现。为此,墨卓生物作为本次活动的发起者与来自全国生命科学和互联网科技两大领域的专家学者、产业领袖荟聚一堂,共同展望生命科学技术的未来。
研讨会由乌镇镇党委副书记、镇长张雪峰开场致辞拉开帷幕,张雪峰镇长为与会的嘉宾展示了乌镇作为世界互联网大会永久举办地的卓越成就,通过多维度发展成功打造数字经济新高地、文旅融合新标杆、民生福祉新典范及国际科创小镇新风貌,彰显了乌镇镇政府在推动经济社会高质量发展方面的显著成效与广阔前景。
随后,由墨卓生物创始人兼CEO裴颢博士致欢迎辞,感谢各界嘉宾远道而来,齐聚乌镇,共同寻找生命科学发展的新脉络,分享最新研究成果。同时,墨卓生物将继续深耕生命科学,推动数字科技与AI融合,期待与各方携手,在融合中发展,在发展中创新,共谱行业新篇章。
在学术分享环节:中国微流控芯片学术泰斗、中国科学院大连化学物理研究所的林炳承教授带来了主题为《尽快推进 AI 技术融入微流控芯片的研究和开发》的报告,展示了结构设计、实时操控、数据处理三种AI与微流控结合的方式,并指出微流控与AI技术融合,能通过图像识别, 深度学习和自主决策,实现对微小尺度下液滴、细胞、微粒的实时感知与反馈并进行操控,同时具有高精度与高可靠性,从自动化发展到智能化。
接着,上海交通大学、微生物代谢国家重点室长聘教授李志勇带来了题为《多组学视角的海绵共生微生物》的报告,介绍了海绵这一珊瑚礁主要底栖生物在环境生态中的重要功能,并通过多组学技术进行深度研究。
来自浙江清华长三角研究院的刘晓军研究员为大家带来了《基于多维组学和分子对接联合筛选的 SCOL 多肽抗心衰特效新药开发》的主题报告,展示了如何利用多维组学和分子对接联合的方法从珍珠中筛选出SCOL 多肽这一极具应用潜力的抗心衰特效新药。
来自浙江大学的蒋超研究员带来了题为《技术驱动的精准暴露组—健康轴研究探索》主题报告,强调了环境化合物和微生物暴露对人类健康的重要影响,并展示了团队近年开发的一系列用于暴露组研究的方法和设备。
最后一位报告嘉宾是来自香港大学的黄适教授,他的报告《Decoding Human Oral-Gut Microbiome Associations with Chronic Diseases via Advanced Microbiome Methods》为大家解析了口腔与肠道微生物组的协同机制及其与慢性疾病的关联。
在新闻发布环节,由墨卓生物和欧易生物牵头起草的行业首个单细胞测序服务类团标(T/STIC130029-2024)正式揭牌。该标准由墨卓生物与欧易生物牵头,联合上海质量体系审核中心、上海市检验检测认证协会等近20家高校、企事业单位共同起草完成,凝聚了行业众多专家与企业的智慧与心血。作为本领域首个服务类标准,它首次对微生物单细胞测序服务提出了规范性要求,为行业的规范化建设和健康发展奠定了基础。
揭牌仪式之后,墨卓生物市场总监於佳乐博士和欧易生物技术支持部经理王惠惠女士分别介绍了各自公司的最新产品和应用进展。
在产业研讨中,来自真迈生物董事长兼CEO颜钦先生、解码生物创始人兼CEO潘加奎先生、韦翰斯生物CEO高鹏飞先生、欧易生物副董事长肖云平先生、霆科生物董事长叶嘉明先生、墨卓生物创始人兼CEO裴颢先生围绕“变革·融合:迈向人工智能+生物技术新时代”展开讨论。嘉宾们从技术创新、产业转化、学术交流、学科融合等不同角度,分享他们的前瞻性洞见,并展望了数智化生命科学的发展趋势,为与会者提供了宝贵的经验和思考路径。
凝心聚力强根基,融合赋能促发展。本次研讨会的成功召开,既是乌镇作为世界互联网大会永久会址,打造数字经济新高地的绝佳展示窗口,也是一次多学科融合和产业交叉赋能的深度交流良机。相信在与会专家和学者的共同努力下,一定能够在百年未遇之大变局的背景下,实现生命科学领域的自立自强,树立全球高端科学仪器研发、生产和应用的新标杆。
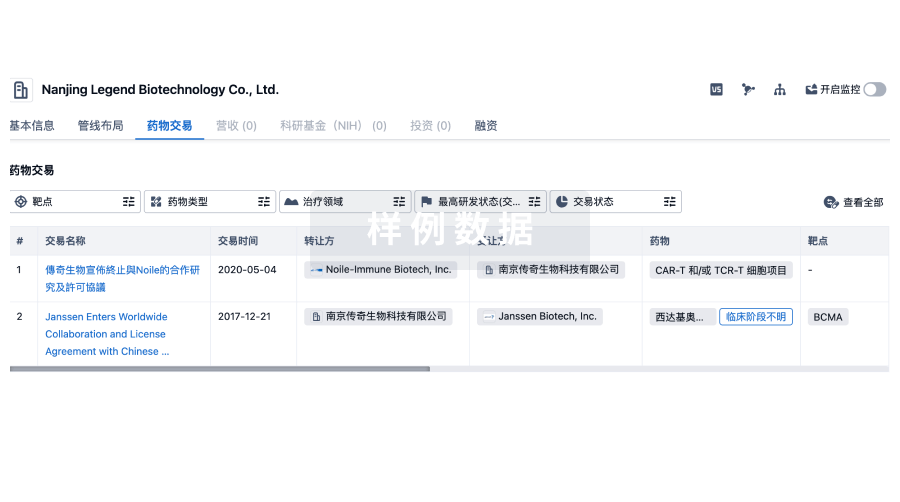
100 项与 墨卓生物科技(浙江)有限公司 相关的药物交易
登录后查看更多信息
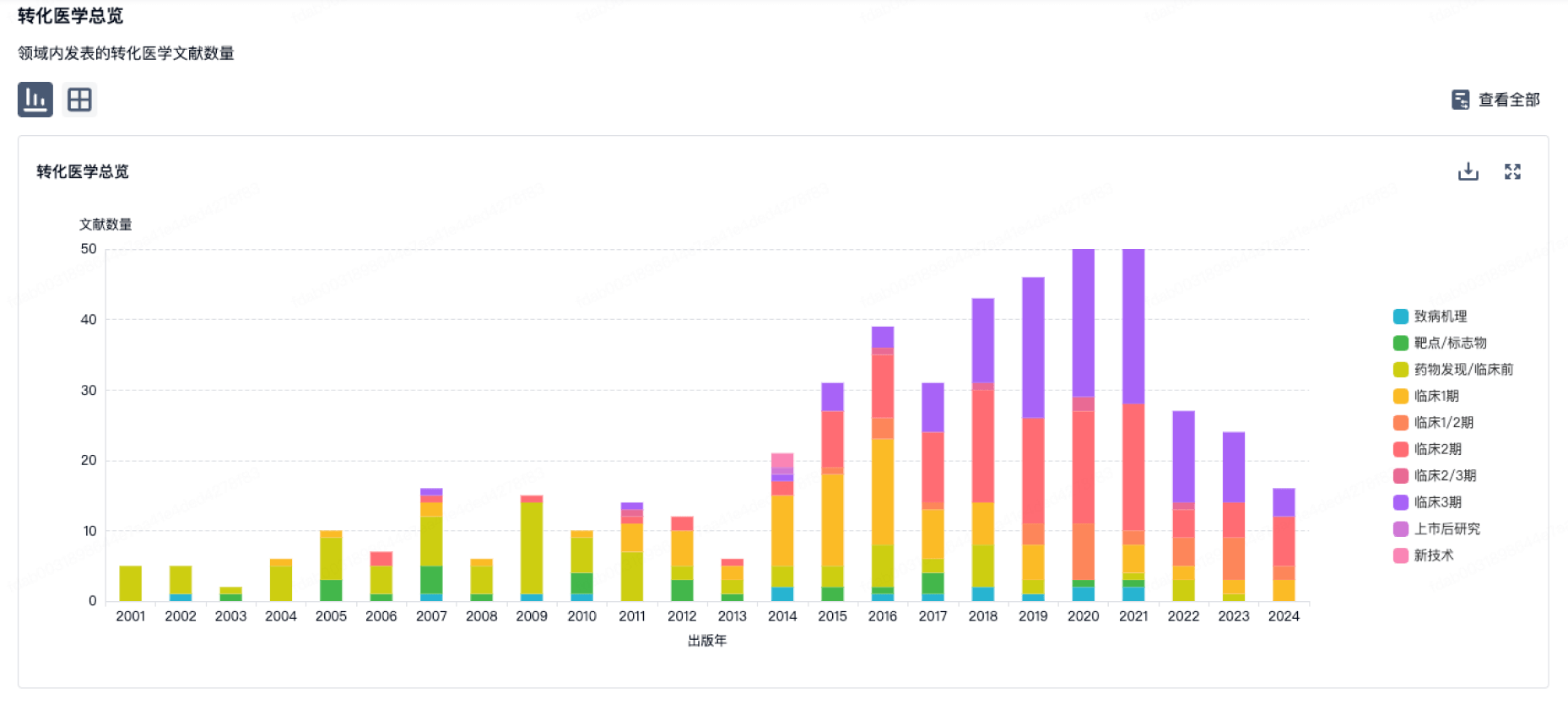
100 项与 墨卓生物科技(浙江)有限公司 相关的转化医学
登录后查看更多信息
组织架构
使用我们的机构树数据加速您的研究。
登录
或

管线布局
2026年02月08日管线快照
无数据报导
登录后保持更新
药物交易
使用我们的药物交易数据加速您的研究。
登录
或
转化医学
使用我们的转化医学数据加速您的研究。
登录
或
营收
使用 Synapse 探索超过 36 万个组织的财务状况。
登录
或
科研基金(NIH)
访问超过 200 万项资助和基金信息,以提升您的研究之旅。
登录
或
投资
深入了解从初创企业到成熟企业的最新公司投资动态。
登录
或
融资
发掘融资趋势以验证和推进您的投资机会。
登录
或
生物医药百科问答
全新生物医药AI Agent 覆盖科研全链路,让突破性发现快人一步
立即开始免费试用!
智慧芽新药情报库是智慧芽专为生命科学人士构建的基于AI的创新药情报平台,助您全方位提升您的研发与决策效率。
立即开始数据试用!
智慧芽新药库数据也通过智慧芽数据服务平台,以API或者数据包形式对外开放,助您更加充分利用智慧芽新药情报信息。
生物序列数据库
生物药研发创新
免费使用
化学结构数据库
小分子化药研发创新
免费使用