预约演示
更新于:2025-05-07
MPG
更新于:2025-05-07
基本信息
别名 3-alkyladenine DNA glycosylase、3-methyladenine DNA glycosidase、AAG + [8] |
简介 Hydrolysis of the deoxyribose N-glycosidic bond to excise 3-methyladenine, and 7-methylguanine from the damaged DNA polymer formed by alkylation lesions. |
关联
100 项与 MPG 相关的临床结果
登录后查看更多信息
100 项与 MPG 相关的转化医学
登录后查看更多信息
0 项与 MPG 相关的专利(医药)
登录后查看更多信息
1,309
项与 MPG 相关的文献(医药)2025-12-31·Annals of Medicine
Identification of ALDH7A1 as a DNA-methylation-driven gene in lung squamous cell carcinoma
Article
作者: He, Jinxian ; Zhang, Liang ; Yu, Kaizhong ; Shen, Weiyu ; Chen, Tian ; Liang, Gaofeng
2025-12-01·Cancer Chemotherapy and Pharmacology
Pharmacokinetics, metabolism, and excretion of [14C]-valemetostat in healthy male participants, and in vitro plasma protein binding
Article
作者: Shimizu, Takako ; Tachibana, Masaya ; Abutarif, Malaz A ; Kazui, Miho ; Inaba, Shinichi ; Craveiro, Thuy Vu ; Ikeda, Tomoko ; Siebers, Nicholas
2025-05-01·Diabetes & Metabolism
Screening for autoimmune atrophic gastritis by serum gastrin measurement in subjects with type 1 diabetes
Article
作者: Larger, Etienne ; Pacheco, Aude ; Mallone, Roberto ; Goulvestre, Claire ; Alexandre-Heymann, Laure ; Dubois-Laforgue, Danièle ; Diedisheim, Marc
3
项与 MPG 相关的新闻(医药)2023-05-22
·生物谷
该研究创造性地设计了一种不依赖任何脱氨酶的新型DNA碱基编辑器,通过蛋白质工程优化开发出了基于糖基化酶的鸟嘌呤碱基编辑器。这一工作不仅填补了目前碱基编辑器不能直接编辑G的空白,而且提出了基于糖基化酶的
截止目前,所有的碱基编辑器(包括线粒体碱基编辑器,DdCBE)都需要以A或C的脱氨基反应作为起始步骤,才能引发后续的DNA修复过程,进而实现目标碱基的编辑。虽然非编辑链上的G或T在编辑链上的C或A被编辑的同时也会相应发生转变,但受限于PAM及编辑窗口等因素的影响,很多情况下仍需要对G或T进行直接的编辑。现有的脱氨酶不能将基因组DNA中的鸟苷(G)和胸苷(T)催化为其他核苷,因此对鸟苷(G)和胸苷(T)的进行直接编辑在基因编辑领域存在技术空白。
2023年5月16日,辉大(上海)生物科技有限公司杨辉团队(以下简称“辉大基因”)在 National Science Review 期刊发表了题为:Programmable deaminase-free base editors for G-to-Y conversion by engineered glycosylase 的研究论文。
图2:gGBEv6.3在內源位点上的性能评估及相关应用
2023-01-11
·生物谷
研究团队通过引入DNA损伤修复蛋白Polη(一种在跨损伤DNA复制过程中倾向于在AP中间体对侧添加A的DNA聚合酶),大大提高了A-to-T的编辑效率和纯度
碱基编辑器(Base editor,BE)是一类可以在不产生DNA双链断裂的情况下实现单碱基水平碱基替换的工具,其在基础研究和基因治疗领域显示出了极大的潜力。在导致人类产生遗传疾病的单点突变中,约四分之一的致病突变的修复需要实现腺嘌呤碱基的颠换(A-to-T和A-to-C;或互补链的T-to-A和T-to-G)。然而,还没有一种碱基编辑器能够实现A-to-T和/或A-to-C这类碱基之间的颠换(即嘌呤变嘧啶)。开发能够实现腺嘌呤碱基颠换的新型DNA碱基编辑器对基因治疗领域有着重大的潜在价值。
2023年1月9日,杨辉团队与临港实验室/上海脑科学与类脑研究中心胥春龙团队合作,在 Nature Biotechnology 期刊发表了题为:Programmable A-to-Y base editing by fusing an adenine base editor with an N-methylpurine DNA glycosylase 的研究论文。该研究开发出了一种新型DNA碱基编辑器,首次实现了高效的腺嘌呤碱基颠换编辑。
该研究综合利用蛋白质工程、流式细胞术、深度测序等技术手段,对ABE进行一系列的重新设计、工程化改造、蛋白质进化、突变筛选及验证,开发出了新型DNA碱基编辑器——腺嘌呤碱基颠换编辑器(AYBE,Y = C or T)。该研究对基础研究领域疾病模型的建立及基因治疗领域等都有着非常重要的意义。
这是辉大基因继获得中国首个自主研发CRISPR-Cas13基因编辑工具(Cas13X/Y)并拥有底层专利、开发出具有高效靶向编辑活性但极低脱靶编辑活性的高保真xCas12i变体(hfCas12Max)之后,在新一代基因编辑工具开发领域取得的新突破,。
研究团队发现N-甲基嘌呤DNA糖基化酶(MPG;也称烷基腺嘌呤DNA糖基化酶,AAG)有一定的切除Hx的活性,于是猜想可以通过工程化改造MPG逐步优化,开发出高效的AYBE。研究团队首先将MPG融合在ABE8e的不同位置构建了AYBE的三种原型版本。为了方便且灵敏地评估腺嘌呤碱基颠换的发生以及颠换编辑的效率,研究团队设计了一套基于内含子剪接可激活的荧光报告系统。
在该荧光报告系统中,只有发生了腺嘌呤碱基的颠换,才能纠正内含子剪接信号使剪接过程正确发生,进而激活绿色荧光蛋白(EGFP)的正常表达,以便结合流式细胞术检测绿色荧光阳性细胞的数目及荧光强度。研究团队利用A-to-T报告系统及A-to-C报告系统都检测到了腺嘌呤碱基的有效颠换编辑,并发现与另外两种融合原型相比,将野生型MPG融合在ABE8e的C端(TCM,称为AYBEv0.1)获得了最高比例的阳性编辑。
图1:AYBE的工程化设计、蛋白质进化、突变筛选及颠换编辑活性检测
为了提升AYBE的腺嘌呤颠换编辑活性,研究团队利用上述报告系统在哺乳动物细胞中对AYBE中的MPG组分开展了逐步的优化。研究人员设计了多种工程化改造方案,构建了一系列突变体库,经过多轮的突变筛选获得了腺嘌呤颠换编辑效率较高的突变体(图1)。利用报告系统评估,AYBEv3版本的腺嘌呤颠换编辑效率达到了初代版本AYBEv0.1的4.78倍,获得了非常显著的优化升级。各个突变体的颠换编辑效率在基因组内源位点上也得到了验证(A-to-T的编辑从 5.88%提高到了 15.49%,A-to-C的编辑从 14.42%提高到了 30.98%),而且AYBEv3有着与AYBEv0.1相当的低indels频率。

基因疗法细胞疗法免疫疗法
2023-01-10
碱基编辑器(Base editor,BE)是一类可以在不产生DNA双链断裂的情况下实现单碱基水平碱基替换的工具,其在基础研究和基因治疗领域显示出了极大的潜力。在导致人类产生遗传疾病的单点突变中,约四分之一的致病突变的修复需要实现腺嘌呤碱基的颠换(A-to-T和A-to-C;或互补链的T-to-A和T-to-G)。然而,还没有一种碱基编辑器能够实现A-to-T和/或A-to-C这类碱基之间的颠换(即嘌呤变嘧啶)。开发能够实现腺嘌呤碱基颠换的新型DNA碱基编辑器对基因治疗领域有着重大的潜在价值。
辉大(上海)生物科技有限公司(以下简称“辉大基因”)继获得中国首个自主研发CRISPR-Cas13基因编辑工具(Cas13X/Y)并拥有底层专利点击阅读、开发出具有高效靶向编辑活性但极低脱靶编辑活性的高保真xCas12i变体(又称为hfCas12Max)点击阅读之后,在新一代基因编辑工具开发领域又获新的突破,又一次展现出了辉大基因开发原创型成果的研发实力。辉大基因创新研究院研发团队主导与辉大基因首席科学顾问杨辉研究员带领的课题组合作开发出新型DNA碱基编辑器,首次实现了高效的腺嘌呤碱基颠换编辑。研究成果“Programmable A-to-Y base editing by fusing an adenine base editor with an N-methylpurine DNA glycosylase”于2023年1月9日在国际顶级学术期刊《Nature Biotechnology》(IF: 68.164)上在线发表。
辉大基因系该论文的第一作者单位,辉大基因创新研究院童华威博士为第一作者,刘纳纳博士为该论文的共同第一作者。辉大基因李芸、罗佳敏、吴丹妮等积极参与课题并做出了重要贡献。辉大基因创始人&首席科学顾问杨辉博士、辉大基因创新研究院童华威博士为该论文的通讯作者。
该研究综合利用蛋白质工程、流式细胞术、深度测序等技术手段,对ABE进行一系列的重新设计、工程化改造、蛋白质进化、突变筛选及验证,开发出了新型DNA碱基编辑器——腺嘌呤碱基颠换编辑器(AYBE,Y = C or T)。本研究对基础研究领域疾病模型的建立及基因治疗领域等都有着非常重要的意义。
腺嘌呤碱基颠换编辑器的开发是碱基编辑器开发领域中仍需解决的难题
现在广泛使用的ABE(Adenine base editor,腺嘌呤碱基编辑器)和CBE(Cytosine base editor,胞嘧啶碱基编辑器)可以实现A-to-G和C-to-T此类碱基之间的转换(transition;即嘌呤变嘌呤,嘧啶变嘧啶)【1-2】。在CBE基础上开发出的CGBE或GBE(Glycosylase base editor,糖基化酶碱基编辑器)能实现C-to-G和C-to-A此类碱基之间的颠换(transversion;即嘧啶变嘌呤)。CGBE或GBE的开发得益于对CBE编辑副产物(包括 C-to-G 和 C-to-A的颠换)的深入研究,拓展了人们对DNA损伤修复机制的研究并拓宽了碱基编辑器的应用【3-7】。细胞中存在可以高效切除胞嘧啶脱氨产生的尿嘧啶(U)的酶(UNG,尿嘧啶DNA 糖基化酶),引入UNG或剔除CBE融合蛋白中的尿嘧啶糖基化酶抑制剂(UGI)组分都能通过生成无碱基(AP,apurinic/apyrimidinic site)中间体介导高效的C-to-G 和/或 C-to-A的碱基颠换。ABE的编辑副产物极少,是由于细胞中尚未发现能高效切除腺嘌呤脱氨形成的脱氧肌苷(I)中次黄嘌呤碱基(Hx)的酶,A脱氨形成的I直接被当作G读取,进而获得A-to-G的高纯度编辑【1】。这也为开发AYBE增加了很大的困难。
研究人员发现N-甲基嘌呤DNA糖基化酶(MPG;也称烷基腺嘌呤DNA糖基化酶,AAG)有一定的切除Hx的活性【8-9】,于是猜想可以通过工程化改造MPG逐步优化,开发出高效的AYBE。研究人员首先将MPG融合在ABE8e的不同位置构建了AYBE的三种原型版本。为了方便且灵敏地评估腺嘌呤碱基颠换的发生以及颠换编辑的效率,研究人员设计了一套基于内含子剪接可激活的荧光报告系统。在该荧光报告系统中,只有发生了腺嘌呤碱基的颠换,才能纠正内含子剪接信号使剪接过程正确发生,进而激活绿色荧光蛋白(EGFP)的正常表达,以便结合流式细胞术检测绿色荧光阳性细胞的数目及荧光强度。研究人员利用A-to-T报告系统及A-to-C报告系统都检测到了腺嘌呤碱基的有效颠换编辑,并发现与另外两种融合原型相比,将野生型MPG融合在ABE8e的C端(TCM,称为AYBEv0.1)获得了最高比例的阳性编辑。
为了提升AYBE的腺嘌呤颠换编辑活性,研究人员利用上述报告系统在哺乳动物细胞中对AYBE中的MPG组分开展了逐步的优化。研究人员设计了多种工程化改造方案,构建了一系列突变体库,经过多轮的突变筛选获得了腺嘌呤颠换编辑效率较高的突变体(图1)。利用报告系统评估,AYBEv3版本的腺嘌呤颠换编辑效率达到了初代版本AYBEv0.1的4.78倍,获得了非常显著的优化升级。各个突变体的颠换编辑效率在基因组内源位点上也得到了验证(A-to-T的编辑从 5.88%提高到了 15.49%,A-to-C的编辑从 14.42%提高到了 30.98%),而且AYBEv3有着与AYBEv0.1相当的低indels频率。
AYBE在基因治疗领域有极大的应用潜力
接着,研究人员在基因组内的35个靶点对AYBEv3作了综合评估,发现其可实现高达72%的颠换编辑效率(其中个别位点A-to-C的颠换效率可达53%,纯度可达70%,图2)。AYBEv3在A7、A8这两个位置及CAA和CAG这两种基序有着最高的颠换编辑效率。另外,AYBEv3在多种哺乳动物细胞类型中都能有效的实现腺嘌呤的颠换编辑。通过对gRNA依赖的及gRNA非依赖的脱靶分析,研究人员发现与ABE8e相比,AYBEv3的DNA脱靶水平更低。本研究也进一步通过建立稳转细胞系探究了AYBEv3在一些疾病相关突变位点上的应用潜力。研究人员发现基因DMD和SLC26A4中提前终止密码子的突变及基因ATM和TTN中内含子剪接位点处的突变可以使用AYBEv3得到有效的纠正。
该研究中,研究人员通过引入DNA损伤修复蛋白Polη(一种在跨损伤DNA复制过程中倾向于在AP中间体对侧添加A的DNA聚合酶)【10】,大大提高了A-to-T的编辑效率和纯度,为进一步开发更精准的ATBE或ACBE提供了新的策略(图2)。虽然AYBEv3的编辑效率及纯度仍需进一步提高,腺嘌呤颠换碱基编辑器的开发填补了目前碱基编辑器不能高效实现A-to-C或A-to-T的空白,对相关疾病模型的建立及基因治疗领域等都有着非常重要的意义。
https://www.nature.com/articles/s41587-022-01595-6
1. Gaudelli, N.M. et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature 551, 464-471 (2017).
2. Komor, A.C., Kim, Y.B., Packer, M.S., Zuris, J.A. & Liu, D.R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420-424 (2016).
3. Kurt, I.C. et al. CRISPR C-to-G base editors for inducing targeted DNA transversions in human cells. Nat Biotechnol 39, 41-46 (2021).
4. Zhao, D. et al. Glycosylase base editors enable C-to-A and C-to-G base changes. Nat Biotechnol 39, 35-40 (2021).
5. Koblan, L.W. et al. Efficient C*G-to-G*C base editors developed using CRISPRi screens, target-library analysis, and machine learning. Nat Biotechnol 39, 1414-1425 (2021).
6. Chen, L. et al. Programmable C:G to G:C genome editing with CRISPR-Cas9-directed base excision repair proteins. Nat Commun 12, 1384 (2021).
7.Yuan, T. et al. Optimization of C-to-G base editors with sequence context preference predictable by machine learning methods. Nat Commun 12, 4902 (2021).
8. Saparbaev, M. & Laval, J. Excision of hypoxanthine from DNA containing dIMP residues by the Escherichia coli, yeast, rat, and human alkylpurine DNA glycosylases. Proc Natl Acad Sci U S A 91, 5873-5877 (1994).
9.Lau, A.Y., Scharer, O.D., Samson, L., Verdine, G.L. & Ellenberger, T. Crystal structure of a human alkylbase-DNA repair enzyme complexed to DNA: mechanisms for nucleotide flipping and base excision. Cell 95, 249-258 (1998).
10.Choi, J.Y., Lim, S., Kim, E.J., Jo, A. & Guengerich, F.P. Translesion synthesis across abasic lesions by human B-family and Y-family DNA polymerases alpha, delta, eta, iota, kappa, and REV1. J Mol Biol 404, 34-44 (2010).
上一条 重磅|辉大团队开发出特异性更高,安全性更好的高保真Cas13蛋白变体,研究成果登《Nature Biotechnology》 下一条 辉大基因科学家团队合作发现治疗杜氏肌营养不良症的潜在新方法
2023-05-25 辉大基因宣布任命陆英明博士为公司首席执行官 2023-09-06 重磅|全球首例!辉大基因原研CRISPR/Cas13 RNA编辑疗法HG202完成首例nAMD患者给药 2023-08-07 重磅|辉大基因首款眼科基因治疗药物HG004获美国FDA儿科罕见病资格认定 2023-08-04 辉大基因与恺佧生物战略合作, 推进新一代基因编辑酶hfCas12Max®的商业化应用
基因疗法细胞疗法
分析
对领域进行一次全面的分析。
登录
或
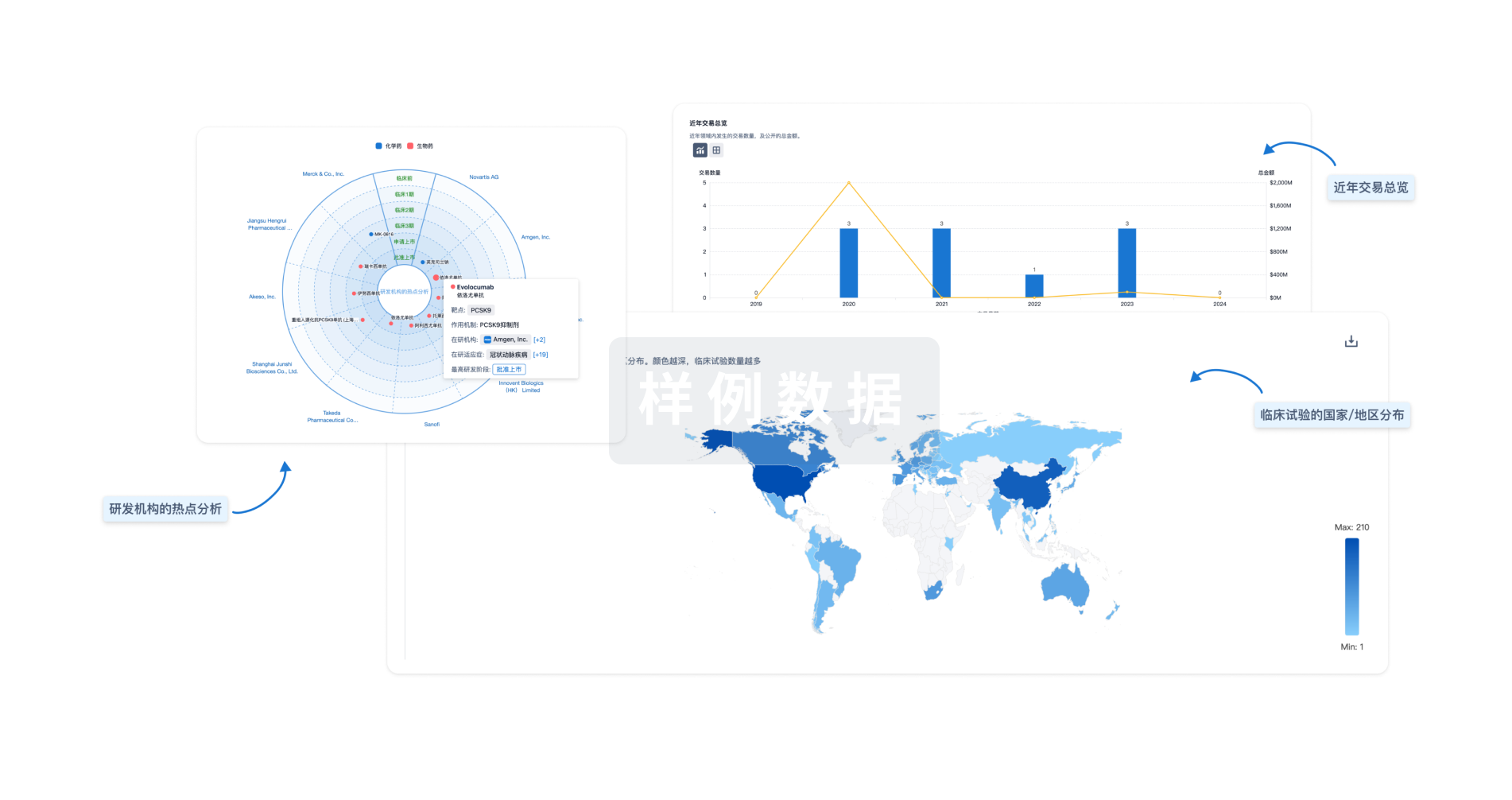
生物医药百科问答
全新生物医药AI Agent 覆盖科研全链路,让突破性发现快人一步
立即开始免费试用!
智慧芽新药情报库是智慧芽专为生命科学人士构建的基于AI的创新药情报平台,助您全方位提升您的研发与决策效率。
立即开始数据试用!
智慧芽新药库数据也通过智慧芽数据服务平台,以API或者数据包形式对外开放,助您更加充分利用智慧芽新药情报信息。
生物序列数据库
生物药研发创新
免费使用
化学结构数据库
小分子化药研发创新
免费使用