预约演示
更新于:2026-02-07
CGX-1007
更新于:2026-02-07
概要
基本信息
原研机构 |
在研机构- |
权益机构- |
最高研发阶段终止临床1期 |
首次获批日期- |
最高研发阶段(中国)- |
特殊审评- |
登录后查看时间轴
结构/序列
分子式C88H138N26O44 |
InChIKeyHTBKFGWATIYCSF-QGXIKSNHSA-N |
CAS号93438-65-4 |
Sequence Code 13443596

关联
2
项与 CGX-1007 相关的临床试验ChiCTR2400088130
Study on the therapeutic effect of traditional Chinese medicine fumigation therapy based on the theory of "XIN ZHU SHEN MING" on the prevention and treatment of ICU delirium caused by excitement
开始日期2022-11-01 |
申办/合作机构 |
ChiCTR-IOR-16008394
Study on the neuroprotective mechanism of compound Cong-Rongjing to GDNF and observation on the effect of early-stage Parkinson's disease
开始日期2016-06-01 |
申办/合作机构 |
100 项与 CGX-1007 相关的临床结果
登录后查看更多信息
100 项与 CGX-1007 相关的转化医学
登录后查看更多信息
100 项与 CGX-1007 相关的专利(医药)
登录后查看更多信息
112
项与 CGX-1007 相关的文献(医药)2024-09-01·Journal of telemedicine and telecare
Using a telemedicine-assisted airway model to improve the communication and teamwork of tracheal intubation during the coronavirus disease 2019 pandemic
Article
作者: Wu, Cheng-Yi ; Tseng, Wen-Pin ; Tien, Yu-Tzu ; Wu, Shih-Ni ; Huang, Chien-Hua ; Tsai, Min-Shan ; Lin, Chien-Hao ; Lin, Hao-Yang ; Chen, Wei-Ting
Introduction:
Isolated spaces impair communication and teamwork during tracheal intubation (TI) in suspected coronavirus disease 2019 patients. We thus aimed to evaluate the telemedicine-assisted airway model (TAM) to improve communication and teamwork during the pandemic.
Methods:
This two-stage prospective study included adult patients intubated in the emergency department of the National Taiwan University Hospital between 1 August 2020 and 31 July 2021. First, we randomised patients receiving TI in the standard setting into the conventional group (Con-G) and the isolation area into the isolation group (Iso-G). We evaluated the obstacles to communication and teamwork in an isolation scenario. Second, we developed the TAM to facilitate communication and teamwork between staff in separate spaces during TI and assigned patients to the TAM group (TAM-G). Communication and teamwork were evaluated using the Team Emergency Assessment Measure (TEAM). Subjective evaluations were conducted using a questionnaire administered to medical staff.
Results:
Eighty-nine patients were enrolled: 17, 34, and 38 in the Con-G, Iso-G, and TAM-G, respectively. The communication frequency (CF) of the Con-G and Iso-G was the highest and lowest, respectively. The CF of the TAM-G increased and approached that of the Con-G. The overall TEAM score was the highest in the Con-G and the lowest in the Iso-G, while the overall score in the TAM-G was comparable to that of the Con-G.
Discussion:
The TAM may improve communication and teamwork for TIs without compromising efficacy during the pandemic. This study was registered at ClinicalTrials.gov; registration numbers: NCT04479332 and NCT04591873.
2023-08-01·Omics : a journal of integrative biology
Correction to:
Multi-Omics and Artificial Intelligence-Guided Drug Repositioning: Prospects, Challenges, and Lessons Learned from COVID-19, by Cong and Endo.
OMICS
2022;26(7):361–371; doi: 10.1089/omi.2022.0068
2023-03-23·Scientific reports
The effect of rope jumping training on the dynamic balance ability and hitting stability among adolescent tennis players.
Article
作者: Lin, Bing ; Xuan, Shu ; Chen, Long ; Xu, Binglong ; Shi, Zuozheng ; Deng, Yue ; Zhang, Xinru
To discuss the effects of 12 weeks of jump rope training on dynamic balance and stroke stability in junior tennis players. Sixteen junior tennis players at CTN7 level were used as subjects and randomly divided equally into ExG and ConG for 12 weeks of training, 3 times a week. The ExG performed Jump Rope training and Special Preparation Activity (JR + SPA), the ConG performed only Special Preparation Activity (SPA) to ensure that the training intensity was basically the same. The Biodex Balance System (BBS) was applied to examine the participants' dynamic balance ability, including the Limits of Stability (LOS) and the Single Leg Stability Test (SLST), the International Tennis Number (ITN) tests ball striking stability. Experimental data were statistically analyzed using independent samples t test, paired samples t test, and repeated measures ANOVA test. We observed significant (P < 0.05) differences in limits of stability (LOS) before and after the test in subjects in the ExG and significant (P < 0.05) differences in ODC, BDC, RDC, FLDC, BLDC, and BRDC metrics in the ExG, and a 2 (group: ExG, ConG) × 2 (time: 0w, 12w) repeated measures ANOVA was performed and found significant (P < 0.05) differences in LDC, RDC, FLDC, BLDC, BRDC, and LOST (S) indexes interacted significantly (P < 0.01) in single-leg stability test (SLST) pre- and post-measures were found in subjects in ExG and in MLSI (L) in ExG (P < 0.05), and 2 (group: ExG, ConG) × 2 (time: 0w, 12w) repeated measures ANOVAs were performed and found that OSI (L), MLSI (L), OSI (R), MLSI (R) interaction on indicators was significant (P < 0.05). The difference in hitting stability between subjects in ExG was significant (P < 0.01) at the pre and post test, while the difference in ConG was significant (P < 0.01) at the pre and post test only on the hitting stability test (depth). 2 (Group: ExG, ConG) × 2 (Time: 0w, 12w) repeated measures ANOVA was performed and found a significant interaction between the two (P < 0.01). We suggest to incorporate Forward double-legged alternating jump rope, Reverse double-legged alternating jump rope, Double-legged alternating cross jump rope, Squatting alternating single-legged jump rope into the regular tennis training course with special techniques, which will help the junior tennis players' dynamic balance ability and hitting stability, and can effectively improve the competitive level of junior tennis players.
88
项与 CGX-1007 相关的新闻(医药)2026-02-05
·微信
1. 生物医药行业长期受到一个问题的困扰:创新周期往往比回报周期更长。
2. 在几乎所有治疗领域里,礼来的整体研发速度,平均仍然比下一家同规模的制药公司快大约40%。
3. CEO的工作职责是创造条件,让伟大的想法能够系统性地不断涌现。
4. 在过去10年里,我当初做过的一个最重要的决定,就是在公司还负担不起的时候,依然为英伟达建造了一台当时全球规模最大的超级计算机之一。
5. 我现在真的认为,生物医药行业的“关键时刻”已经到来了。能够从“药物发现”这种有点像在森林里找松露的方式,转向真正的计算机辅助药物设计。
6. 当然,这背后的科学极其复杂,人类生物学本身也极其复杂,可能(完全实现)还需要10年时间。
7. 如果我们能把这件事从一种“手工艺式的发现”,变成一个工程问题,那对人类生命的影响将是巨大的。
8. 我们更关注一个更现实的问题:如果最大寿命上限是100岁,我们怎么让更多人更有机会真正走到那一天?而我们的方式,是通过消除疾病来实现这一点。
9. 如果说在AI领域目前最容易落地,也最可操作的方向,我认为是医疗服务。
在上个月举行的2026年摩根大通医疗健康大会上,英伟达创始人黄仁勋与礼来公司CEO戴文睿(Dave Ricks)在宣布重大合作后进行了一场深度对话,大约1小时。今天刚放出了完整视频。
两家公司将在旧金山湾区共建一个首创性的AI联合创新实验室,并计划在未来五年内共同投入高达10亿美元,将AI技术彻底融入药物发现和生物医学研究流程。
两位掌舵者都无比兴奋。
对于生物医药行业来说,传统新药研发周期长、成本高、成功率低已成为公认的结构性难题。而AI具备高速模拟、海量空间探索和自动化协同的潜力,被视为可能改变这一格局的关键力量。
过去几年,礼来在代谢性疾病领域的突破已无需赘述。
以替尔泊肽(Tirzepatide)为代表的GLP-1药物,不仅在减重和糖尿病治疗中展现出前所未有的疗效,也正在显著改变慢性病管理的边界。
戴文睿在对话中反复强调的是一个更长期的目标:把药物发现从高度经验化的探索,推进为可以不断复制、持续优化的工程体系。
对话还提及,该合作框架未来将向生物医药初创公司开放,通过联邦学习等机制,在不共享原始数据、不损害知识产权的前提下,共同训练模型、放大创新效率。
看起来挺酷的。
值得一提的是,2026年2月4日礼来发布2025年财报,全年营收651.79亿美元,同比增长45%,预计2026年营收规模在800-830亿美元之间。
戴文睿在对话中反复提到的替尔泊肽合计销售额365.07亿美元,其中四季度销售额116.70亿美元。替尔泊肽全年销售额超过司美格鲁肽和Keytruda,正式登顶药王宝座。
在这个利好刺激下,礼来最新股价丝毫没有理会指数的颓废,蹭蹭蹭涨了10.33%,重返1万亿美元市值。
聪明投资者(ID: Capital-nature)抱着学习心态整理精译主要内容分享给大家。嗯,考虑到篇幅,忍痛删除了一些两人逗捧哏的有趣过程。
加速药物研发速度以及如何更好接棒
黄仁勋 说起来,我们两个人的成长背景,几乎不可能更不同了。戴文是鹰级童子军,而我则上过一所“问题少年学校”。先澄清一下,我是被误送进去的,那并不是什么糟糕的学校,其实是一所非常好的学校,而且学费也很便宜。
所以回到正题。2017年,你成为了CEO。我的问题是:当时的礼来是一家什么样的公司?那时它对生命科学未来的愿景是什么?而今天的礼来又处在什么状态?在信念的层面上,你现在对生命科学未来的看法又是什么?
戴文睿 2017年的时候,礼来刚刚超过140年,是一家相当成熟、根基深厚的公司。不过,在那之前,我们的行业经历过一段相当艰难的时期,这一点稍后我也可以再展开讲。
正如在座很多人所知道的,我们这个行业长期受到一个问题的困扰:创新周期往往比回报周期更长。
平均来看,你会不断经历高峰和低谷的循环。
而在低谷期,你要么撑不过去,被并入其他公司,最终只剩下别人公司名字后面的一个“连字符”;要么就学会如何活下来,并做出极其艰难的决策。
我认为,礼来几乎是唯一一家在经历这些周期时,没有通过并购或合并来渡过难关的大型制药公司。
我们已经经历过好几轮这样的周期,因此也逐渐形成了一种在艰难时期具有生存韧劲的组织DNA。
其中有一件事对我触动非常深。当时我已经是执行委员会的一员,我们经历了2010、2011、2012 年那一轮周期。那段经历让我意识到,在发明和创新这件事情上,你必须极端地、持续地保持强度。
因为这个时间周期非常残酷:一旦你遇到专利悬崖,却又没有任何产品可以接续,你就不得不对整个公司进行重塑。
想象一下,每隔10到15年就来一次这样的过程,组织根本不可能积累真正的动能。人会离开,隐性知识会流失等等。
于是我们开始着手应对这个问题。我认为大致有两种路径。
第一种是说“那我们就要有更好的点子” 。但正如你们所知道的,这很难长期维持。即便你一度有很好的想法,外界也觉得你很聪明,现在大家对礼来的看法就是如此,但这种状态是很难持续的。
所以我们当然也在努力提高发明本身,但更重要的一件事,是加快发明的速度。
我们系统性地拆解了整个研发流程,几乎是逐个环节来做,从候选分子的选择到第一次获批,中间有上百个步骤,我们把时间一段一段地压缩下来。
直到今天,在几乎所有治疗领域里,礼来的整体研发速度,平均仍然比下一家同规模的制药公司快大约40%。
这意味着,我们可以在专利周期之内完成真正的发明,然后公司才有可能重新进入增长轨道。
这在2017年还只是一个设想,而现在已经被真正落实了。
与此同时,我们也做了很多事情来推动更多的发明。我们很幸运,在2006年就较早押注了一个叫GLP-1的方向,并推出了全球第一款相关产品。
当时几乎没人听说过这个概念。我们在这个方向上反复摸索、不断尝试,大概折腾了12年。对整个人类来说,确实是一个非常好的想法。
黄仁勋 听起来像个芯片名字,GLP-1。
戴文睿 确实有点像。你是不是想问,会不会有GLP-2、GLP-3?
确实有GLP-2,那是一种真实存在的蛋白,不过作用基本相反。我们并不希望把这两种激素结合在一起,因为市场非常小,我得说,真的非常小。
我并不是医疗专家,但如果我没理解错的话,现在我们其实已经算是在“GLP-2”阶段了,因为替尔泊肽是把两种蛋白融合在一起的,而这是我们开发出来的一项技术。
接下来还有一个三重作用机制的分子,预计在2027年推出,那会是一个相当有意思的阶段。
不过总的来说,我们抓住了这一波浪潮。最初是竞争对手在肥胖领域率先完成了创新,而我们随后追赶并执行得相当不错,最终在去年成为了市场领导者。
所以现在,我们不仅拥有一台更快的创新引擎,而且还处在一个由单一理念带来的巨大成功周期之中。
摆在我们面前的挑战是:如何在这一轮成功周期结束之前,就提前找到下一轮成功,并获得持续向前的速度。
这件事,坦率说,行业里还没有哪家大型公司真正做到过。大家一直只是经历一轮又一轮的周期,而我们的目标,是打破这一点。
(聪投注:这段让人想起3万亿美元的近百年的资管巨头资本集团(Capital Group)。其历史上最杰出的分析师之一——杰森·皮拉拉斯(Jason Pilalas)在职业生涯中,正是通过对单一突破性产品的前瞻判断,捕捉到一批伟大的医药投资机会:因胃溃疡药物泰胃美(Tagamet)押中史克,因善胃得(Zantac)投中葛兰素,又因降压新药依那普利(Vasotec)看准默克的长期潜力。这些成功案例为资本集团带来过丰厚的回报。
但同样重要的是,当他意识到某些公司在核心产品之后缺乏清晰、可验证的后续研发接力时,即便当期业绩仍处高峰,也会选择退出。资本集团内部始终坚持一个判断:医药公司的长期价值,不取决于“这一款药有多成功”,而取决于“成功是否能够被接续”。这恰恰呼应了戴文睿在对话中的核心命题——真正困难的,不是创造一次突破,而是在每一次产品周期之后,为下一次增长提前铺路。)
CEO的根本职责是创造条件
黄仁勋 你刚才提到的一点,其实我经常对我的员工说,CEO的根本职责,并不是亲自去想点子。我们的工作是创造条件,让伟大的想法能够系统性地不断涌现。
你刚才描述的,本质上就是礼来在长期实践中形成的一种智慧:必须创造出让这些重大创新得以发生的条件。
我们以前也聊过英伟达早期进入AI领域的事情。回头看,英伟达做过的最重要的一件事,就是在基础设施上提前下注,去创造条件,让最优秀的AI研究人员愿意加入我们,从而让伟大的发明自然发生。
今天我们对这件事其实讲得不多,但越来越多的人开始意识到,在过去10年里,我当初做过的一个最重要的决定,就是在公司还负担不起的时候,依然为英伟达建造了一台当时全球规模最大的超级计算机之一。
那时,甚至没有多少AI研究人员能真正用得上它。
如果你想吸引世界上最顶尖的粒子物理学家,却没有大型强子对撞机,那几乎是不可能的。
这也把我们的对话自然引向了一个结论:AI已经取得了足够的进展,使我们有可能把它应用到人类最非凡、也最困难的挑战之一——生物学。
于是,我们两个人就开始设想,如果全球最大的计算与计算机科学公司,能够与全球最重要的生命科学公司展开深度合作,会发生什么?
这就好像我们共同创建了一间实验室,在某种意义上把两家公司当成一家公司,把所有最顶尖、最不可思议的能力整合在一起。
戴文睿 非常令人兴奋。今天我们正式宣布了这项合作。这项合作大致分为四个部分。
首先,我们采购了大量芯片,并正在建设一台全球最大的、专门用于生物学研究的本地部署超级计算机,预计就在本周完成。这台计算机将位于印第安纳波利斯,这里本身就是全球生物药物发现的重要中心。
在超级计算这件事之外,我们还会在湾区组建一支研究团队。如果有优秀的科学家或者计算机科学家想加入,这将会是一个礼来—英伟达联合的 AI 实验室。
我们还会着力开发全新的数据。因为任何真正了解这个问题的人都知道,你可以训练模型,但如果数据“只有一英寸厚”,你是走不了多远的。
而生物学在很大程度上仍然是未知的。所以,我们需要为了训练机器而开展大规模的实验,持续生成数据。
接着,我们还要在英伟达已经构建的模型基础之上继续往上“爬梯子”,做出更复杂、更高级的预测。
从“手工艺式的发现”变成一个工程问题
黄仁勋 理解AI的一个重要方式,是把它想象成一块多层蛋糕。
最底层,你需要能源来驱动这些计算机;再往上一层,是计算机里的芯片;再往上,是基础设施层。这一层本质上是在解决这样的问题:如何让计算机学习?如何处理数据?如何设置安全护栏?如何微调?如何教学?
这层有点像AI的人力资源部门,只不过他们的工作效率要高得多。
在基础设施之上,是模型层;再往上,是领域特定的数据和飞轮机制。
真正令人兴奋的一点在于:我们可以训练模型来合成蛋白质或化学分子,把这些结果送进机器人实验室进行实验,收集新的数据,再把这些数据反馈回模型中。
这个飞轮——用AI生成蛋白或分子、进行真实实验、获得现实世界的数据,再把整个科学飞轮加速运转。这样的未来,令人无比兴奋。
所以,我们正在系统性地把药物发现领域中最优秀的专家,与计算机科学领域中最优秀的人才聚集到一起。
在我们的联合创新实验室里,基本上会覆盖这块“蛋糕”的每一层,希望最终能勾勒出一幅关于未来药物发现的蓝图。
大概在40多年前,准确地说是43年前,我属于第一代工程师,那一代人第一次迎来了“计算机辅助设计”这个概念。
在我之前的工程师世代里,他们会设计一款芯片,可能会成为一次成功,但接下来好几年都没有新的突破;再设计一款,又成功一次,然后又停滞几年。
戴文睿 现在这个行业已经完全不是那样了。
黄仁勋 没错。而我们这一代人出现之后,第一次能够用软件来表示晶体管、逻辑门和系统功能。
计算机终于可以准确地表示你的领域,并模拟它的行为,尽可能在硅片里完成设计和验证。
今天我们设计的是极其庞大的系统。大约有1.5万人共同参与一套计算系统的设计。我们刚刚完成了Vera Rubin的流片,投入了数十亿美元的研发费用。
最终完成的计算机重达三吨,包含大约150万个部件、6颗全新芯片,总计220万亿个晶体管。
而最关键的是,它第一次运行,就完全符合我们的预期。每一代系统,都是在上一代基础上构建的。
我现在真的认为,生物医药行业的“关键时刻”已经到来了。
经过百万倍、甚至更高数量级的算力提升之后,我们终于也许能够表示并理解世界上最困难的问题之一——人类生物学。
能够从“药物发现”这种有点像在森林里找松露的方式,转向真正的计算机辅助药物设计。
顺便说一句,礼来当年真的有一个“土壤发现部门”,会派人去热带雨林采集土壤样本,带回印第安纳波利斯,从中提炼出后来变成抗生素的物质。
事实上,医院里最重要、也是最后防线的抗生素之一——万古霉素,就是礼来在婆罗洲的一份土壤样本中发现的。
那他们当初怎么知道要去哪里?其实也不知道,只是因为真菌会产生对抗细菌的天然机制,于是他们就去挖土、去找这些天然防御体系,再把它们转化成药物。
这才是真正意义上的“药物发现”。
你仔细想想,这种工作方式是有生命风险的,甚至可能得疟疾。那是1950年代的事情,距今已经70年了。
但现在不同了。现在,你们的工作方式开始变得像我的工作一样,基本不用离开办公室。
戴文睿 是的,全都在屏幕里完成。
黄仁勋 所以,真正让我感到兴奋的是,我们也许真的能够实现计算机辅助药物设计。当然,这背后的科学极其复杂,人类生物学本身也极其复杂,可能还需要10年时间。
但我真心认为,这次我们之间的合作宣布,可能会成为某种起点,一种蓝图的开始。
戴文睿 我也希望如此。我们确实需要这样的突破。
你在摩根大通的大会上会看到,行业里有着令人难以置信的研发管线,但其中大多数成果,都是一点点凿出来的,是非常艰苦、非常经验主义的过程。
偶尔我们能借助一些系统性突破,比如单克隆抗体,本质上是“劫持”了生物体自身解决问题的方式。
但大多数时候,每一个小分子发现都像一件艺术品,独一无二,稍有偏差就前功尽弃。
如果我们能把这件事从一种“手工艺式的发现”,变成一个工程问题,那对人类生命的影响将是巨大的。
关于GLP-1的机理以及更多价值
黄仁勋 回到GLP-1。你刚才提到,它其实已经进入第三阶段了。那么接下来会走向哪里?还能用来做什么?
戴文睿 事实上,肥胖是成年人慢性疾病中一个非常关键的枢纽变量。
很多当下的疾病,在人类进化史上根本不是问题。我们进化得太慢,而环境变化得太快,于是出现了错配。肥胖正是这种错配的产物。
我们是在一个资源稀缺的环境中进化出来的。在人类进化过程中,其实并不需要为“饥饿”设计一个关闭开关。因为当时的世界并不存在“过剩”这个概念。
现在不一样了,现在的关键词是“丰裕”,是过剩。不论是热量、智能,还是生产力,我们都生活在一个高度丰裕的世界里。至少对我来说,我的生活几乎被各种各样的“过剩”所包围。
但问题在于,我们现在也处在一个热量极度过剩的环境中,而进化并没有提前为我们设计好一个“停止进食”的机制。
从进化角度看,这是完全没有必要的。所以我们才会出现肥胖问题,而肥胖又会引发大量慢性疾病,折磨人类,甚至缩短寿命。
如果我们能拥有一套相当有效、而且还会不断变得更有效的工具去真正帮助人们管理体重,用一种人工合成的方式,帮身体装上一个关闭开关,那么我们就有机会下调、甚至显著缓解200多种慢性疾病。
黄仁勋 真的有这么多?
戴文睿 确实如此。接下来24个月里,会发生两件非常重要的演进。
第一,是选择会显著增多。GLP-1本身只是一种肽,但它属于一个家族。
通过在替尔泊肽中把GIP和GLP结合在一起,我们已经看到药效更好、耐受性也更强的结果。未来你可以预期会看到更多类似的组合。
在这个“超级蛋白家族”中,我们可以整合不同成员的优势,做出更容易耐受、同时效果更强的药物。
不论是在维持阶段、诱导减重阶段,还是针对不同患者特征的个性化需求,都会有更多方案出现。正如我刚才提到的,我们的三重作用分子也在推进中。
而单纯的体重下降,本身就会带来极其广泛的下游好处。这也引出了第二个重要变化:应用场景的拓展。
现在我们已经清楚地看到,使用Zepbound(替尔泊肽减重注射液)后,平均体重可下降约23%。但真正逐步显现的,并不只是减重本身,而是一系列原本可以预期、却远比想象中深远的连锁结果,包括心梗风险下降,整体代谢系统明显改善。
在糖尿病与糖尿病前期人群中,向糖尿病发展的转化概率下降了93%。可以设想,如果在全国范围内,为约7000万名糖尿病前期人群提供 Zepbound,美国的糖尿病患者规模在理论上将大幅收缩。
这是一个极其重大的发现。若与特朗普政府刚刚签署的医保准入协议能够顺利推进,或许真的有机会向这一方向迈出实质性一步。
除此之外,还有一些不那么直观、却同样关键的作用机制,其中最重要的一项是炎症。肥胖所引发的,并非一次性的急性反应,而是一种长期存在、系统性的慢性炎症。这种状态持续侵蚀心血管系统,加重关节负担,也会在更广泛的层面上损害身体机能。
在早期临床试验阶段,我们就开始自发地听到患者的反馈:他们不仅体重下降了,而且有人说,这是十年来第一次能够轻松地从椅子上站起来;膝盖不再像以前那样疼;背部的不适明显缓解。还有一些患有克罗恩病或结肠炎的患者,甚至报告症状几乎消失。
为什么会出现这些变化?关键在于,我们并不是在处理某一个孤立症状,而是在系统层面降低了长期存在的慢性炎症水平。基于这一机制,我们已经启动了一系列针对不同疾病的研究。
上个月公布的首项结果显示,在银屑病关节炎患者中,在原有基础用药之上联合使用Zepbound,整体疗效提升了约50%。
接下来,这些潜在应用将以医学体系能够接受的方式逐步推进——也就是通过清晰、可验证的适应症,让创新真正进入临床实践。
黄仁勋 这和AI的采用路径其实很像,你也需要在具体场景中证明它的价值。
戴文睿 完全一致。我们已经启动了一系列脑健康相关研究,并且专门为神经系统疾病设计了GLP-1 / GIP的特定组合,其中一个重点方向就是成瘾问题。
成瘾,是一个非常严峻的现代社会挑战。有意思的是,这类药物抑制食欲的机制,其实并不只是因为身体需要更少的热量,而是因为它们作用于一些后天学习形成的行为回路。
听起来是不是越来越像AI了?同样的机制,也可能适用于其他具有自我强化特征的有害行为,比如赌博、饮酒,甚至戒烟。
因此我们也在系统性地探索这些方向。当然,我们希望它不会影响那种工作过于拼命的行为。
黄仁勋 对,那可不行。
戴文睿 放心,只会针对不良行为。事实上,这些行为在模式上,与过度进食高度相似。
顺便说一句,这是给在座各位创业公司CEO的一个友情提醒:如果你发现自己的意志力开始下降了,先停一停微剂量试验。
黄仁勋 现场可是有不少创业CEO。
戴文睿 目前大家也在同步研发作用时间更长的方案,以进一步降低用药频率。今年春天,我们还将推出口服GLP-1。这不仅是使用便利性的突破,更重要的是一次全球可及性的突破。
从外表看,我们现在的自动注射装置似乎很简单,但从制造角度来看,这其实是全球最复杂的药物之一,而且面临的是数亿人次级别的需求规模。
我们刚刚在中国、印度以及东南亚推出产品,而这些地区的肥胖问题同样非常严峻。仅依靠注射制剂,根本无法满足需求,尤其是在东南亚,那里糖尿病负担极其沉重。
某种意义上,东南亚人群在肥胖问题上“输掉了遗传彩票”:在更低的体重水平下,哪怕是小幅度的体重增加,也更容易触发一系列疾病。因此,我们必须具备大规模生产能力。
口服制剂在化学层面实现,不依赖蛋白合成,也不需要无菌注射系统——只是一颗药片,就能实现极大规模的扩产。这一产品也将于今年上市。
黄仁勋 也就是说,这一个想法,你们还可以再跑五十年?
戴文睿 我们是这么希望的,也是在向你学习。
GLP-1的发现过程以及之后的重点
黄仁勋 当然,我们面临的一个挑战是:能否持续不断地发明出足够好的新产品,让人们愿意为其付费,从而跨越专利周期?历史上有没有哪一种生命科学技术,曾经如此广泛地造福人类?
我能想到的,可能只有抗生素这一整个药物类别。
戴文睿 没错。
黄仁勋 青霉素的发现,几乎创造了现代医学。如果没有抗生素,就不可能有现代牙科、现代外科。今天我们已经把这一切视为理所当然。
但从弗莱明在实验室里发现那团奇怪的霉菌开始,这开启了一条持续60年、诞生100多种新药的创新浪潮。
我觉得,GLP-1也可能属于这样的技术浪潮。不同之处在于,它针对的不是急性感染,而是现代智人所面临的慢性疾病。
戴文,你有没有意识到,你的工作听起来竟然会这么“轻松”?我太太也问过我这个问题。
为什么我不来你们这儿工作?一次伟大的发现,然后几十年都在享受红利。你们在森林里挖着挖着,就挖到了GLP-1这颗“松露”。
戴文睿 其实历史并不是那样的。
这个故事要追溯到上世纪70年代。当时,一位德国科学家发现了所谓的“促胰岛素效应”(incretin effect)。GLP-1正是这一激素家族中的一员。
这类激素由肠道分泌,用来向全身传递一个信号:你已经进食了。
他观察到,如果将糖分通过口服方式给予人体,与静脉注射相比,胰岛素的反应完全不同。胰岛素本身是一种促进糖吸收的激素,而在口服条件下,这一反应明显更慢。这一现象提示,肠道的参与会改变整个系统的反应路径。
到了80年代,研究人员首先发现了GIP,随后发现了GLP-1,这正是Zepbound的两个组成部分。
此后,人们又陆续识别出更多相关激素。但始终存在一个核心难题:这些靶点极难做成真正可用的药物。天然GLP-1在人体内的半衰期只有大约7分钟。
如果直接使用这种天然肽作为药物,患者就需要全天候持续输注,这在现实中根本不可行。
直到我们逐步学会将作用时间从每天两次延长到每周一次,如今甚至在探索按月给药的可能性,GLP-1才真正具备了成为产品的基础。
而在AI药物发现领域,已经开始显现效果的一点是:我们可以生成海量的组合方案,在计算环境中进行筛选,再从中过滤出真正具有潜力的候选物。
我们已经在规模化地推进这项工作,而通过这次合作,规模还可以再放大一个数量级。
黄仁勋 那我们能不能用AI找到更多生物学靶点?那才是真正的“圣杯”。因为如果把这两件事结合起来,我们就有可能一次性对整个系统进行建模,那会非常、非常令人兴奋。
还有一件事,是戴文一直特别想做的——把这个AI实验室、这个联合创新实验室放在这里。放在硅谷,甚至就在这座“AI之城”的附近。
旧金山确实是很多AI研究者的聚集地,我们在这里也有办公室。所以,这个实验室要么设在南旧金山——那一带是湾区的生物科技中心,要么就设在这里。
戴文睿 没错。我们想去人们真正愿意生活的地方。
黄仁勋 说到GLP-1以及它之后的东西,硅谷现在有不少人在聊长寿、聊延缓衰老。那你跟我讲讲,这背后的技术路径是什么?
戴文睿 这是个很有意思的话题。它当然对很多人都有吸引力,毕竟谁想变老呢?
细胞为什么会“到期”,这里面有很多生物学机制,你可以把它外推到细胞系统,再外推到器官,最终甚至外推到整个个体。但正如我们在台下聊到的,那不是我们现在主要的工作方向。
那条路径的核心问题,是能否把人类在理想条件下的最大寿命上限进一步往后推。
比如说,在理想条件下人可能活到100岁,然后系统出于某些原因就关闭了。坦率说,如果人能永生,那世界会很奇怪。那我们为什么要繁衍?我们怎么进化?这里面有很多问题,甚至是哲学层面的:自然的“原始设计”要怎么自洽?
我们更关注一个更现实的问题:如果最大寿命上限是100岁,我们怎么让更多人更有机会真正走到那一天?而我们的方式,是通过消除疾病来实现这一点。
你如果回看1900年的美国,男性的预期寿命大约只有46岁。最主要的原因,并不是人老得快,而是儿童死亡率极高。大量人群在很小的时候,就因为感染等原因去世。
随着抗生素、疫苗,以及一整套控制儿童疾病的医学工具出现,预期寿命迅速被拉升。
从1960年到今天,我们又大约增加了10年的预期寿命,这一阶段的提升,主要来自慢性病管理能力的持续进步。
癌症治疗取得了巨大突破,在座的很多公司,都在慢性病管理中发挥了重要作用;HIV/AIDS 以及其他新型病原体,也被逐步控制。
今天,美国的预期寿命已经接近80岁。事实上,如果不是阿片类药物滥用和交通事故死亡率异常偏高,这个数字可能更接近84岁。
而这两项,在美国都显著高于其他发达国家。自动驾驶或许能解决其中一个问题;另一个问题,也许GLP-1能发挥作用,因为我们已经开始研究它在阿片成瘾上的潜在影响。
我们正是通过这样一项一项、系统性地推进,不断拉长人类的健康寿命。当然,随着人活得更久,与衰老相关的新问题也会随之出现。
对我来说,在GLP-1之后,我最兴奋、也最关注的下一个前沿,是痴呆症以及与衰老相关的脑部疾病。
当我们的身体可以支撑我们走到八十岁、九十岁,大脑这件事,却还远没有被真正解决——无论是对生活质量,还是对寿命长度来说,这都会变得至关重要。
我们已经在这一领域投入了大量精力,也有一些令人期待的项目正在推进。从药物发现的角度看,如果我们能够模拟脑组织,去寻找新的靶点,并理解那些我们目前尚不清楚的靶点之间的相互作用,也许就能打开新的突破口。
衰老的大脑中,同时存在炎症和蛋白质错误折叠等问题,那里可能正隐藏着下一代治疗的关键。
关于联合创新实验室的构想
黄仁勋 如果我们退一步来看这间联合创新实验室,第一件事,其实是把计算平台搭建好,让研究者能够真正开展工作。
所以我们有算力、有基础设施层、有BioNeMo,也有一整套已经构建完成的模型和模型架构。接下来,就是把科学家聚到一起,在这些基础之上开始协作。
但如果你要给研究团队一个愿望清单,你最希望他们把精力放在哪个方向?
戴文睿 我觉得可以先把问题简化为两个方向。当然在每个方向里,我们都需要进一步聚焦,只有这样才能持续推进,也才能形成可交付的成果。
第一个问题,其实我们这个行业已经有相当的动能了,也就是工程化的药物设计。
今天很多药物,本质上都属于这一类:靶点已经存在、也被充分研究过,真正的挑战在于,如何更精准地定制那把“钥匙”,去匹配那把“锁”。
与此同时,也出现了一些新的治疗范式,比如RNA、基因疗法等。这些技术出现的时间还不够长,还远没有被充分优化。
所以我认为,围绕药物工程和药物优化这一整套问题,本身是相对可解的,而英伟达的科学家与我们的研究团队结合起来,很可能在这一领域较快取得实质性进展。
这类成果,也非常适合通过Tune Lab这样的接口开放出来,与更多生物科技合作伙伴共同使用。我们可以把“更好的药物制造”这件事,通过计算机内的设计与工程化,真正推向规模化。
简单解释一下Tune Lab:这是一个平台,让礼来能够与初创公司或第三方一起合作,共同推动某个具体方向的进展。
它基于联邦学习的技术框架,底层是我们贡献的NVFlare。研究者不需要集中或混合数据,却仍然可以共同训练模型,从而实现协作式创新。
自从我们宣布合作以来,几乎一整天都有初创公司主动来询问如何参与Tune Lab 和联合创新。
Tune Lab的构想非常有前瞻性,它解决了一个长期存在、却极其棘手的问题:如何在保护数据与知识产权的前提下,让不同公司的AI研究者真正协作。
我们不收取费用,但会进行筛选,确保项目是严肃、可信的生物科技研究。合作的成果,也会被整合回Tune Lab,让不仅是礼来的科学家,更多生物科技研究者都能从中受益。
第二个问题,是靶点本身。在这一点上,我认为机器人实验体系必须真正发挥作用。
为什么我们可用的靶点还不够多?为什么我们对已有靶点的理解仍然不够深入?很大程度上,是因为它们的结构和行为,比我们以为的要复杂得多。
药物研发失败的一个重要原因,正是我们以为理解了靶点,但实际上并没有。
这背后需要的是围绕关键靶点,获取海量、系统性的实验数据,把它真正“刻画清楚”。
我们已经有机器人湿实验室在运行,但接下来会建更多,让它们24小时不间断工作,在一个靶点空间内持续实验、持续生成数据,形成完整画像,再与工程化药物设计结合。
这条路更难,也更偏经验主义,但机器天生适合日夜不停地做这件事。一旦这个飞轮转起来,它会转得非常快。
AI领域目前最可操作的可能是医疗服务
黄仁勋 戴文,如果你回头看我们在其他领域取得的一些突破,第一件事往往是:你最终必须让这个飞轮落到现实世界。
你得做真实的实验,收集真实的数据,用它把闭环跑起来。随着循环推进,模型会变得足够好,基础模型会变得足够强,你就可以教它去生成、去做生成式工作。
最终可能会出现这样一种形态:一个模型在不断与另一个模型对抗、验证。而那个“另一个模型”,其实就是“世界模型”或者“靶点模型”。
靶点模型会和你训练中的模型一起,在一个闭环里运行。从这个意义上讲,实验室本身也在变成一个模型。
当然,你最终还是要重新落地,真正去合成蛋白,产生真实数据。
但在这个过程中,我们会进入一个合成数据飞轮:先在模型里生成、再在模型里筛选、再用真实实验去校准。
这个飞轮的效率极高。我认为在我们这个行业里,以往从未有人以这样的规模去尝试,而我相信我们会在这里做到。
真正关键的是,挑选出少量我们高度怀疑与重大疾病密切相关的靶点,然后在机器人湿实验室里对它们做完整、系统的刻画,用实验数据训练数据集,再让机器去填补中间的空白、扩展整个空间,最终做出更好的药物——因为我们真的理解了这些靶点。
这实在太令人兴奋了。我认为这可能是第一次,我们拥有一个在专业深度和规模上都足以吸引顶尖人才的实验室,让他们愿意把一生投入到这个交叉领域。
这场大会有一件特别棒的地方,就是大型公司和小型公司能够聚在一起。你这边看到了哪些公司?哪些方向让你觉得特别有意思?
戴文睿 我看到的方向很多。比如机器人实验室相关的公司、AI 科学家团队,当然也包括代理式医疗(agentic healthcare)相关的公司。像Abridge在做的事情,还有Open Evidence,也都非常、非常酷。
如果说在AI领域目前最容易落地、也最可操作的方向,我认为是医疗服务。
这当然不像我们今天讨论的这些问题那么复杂,而是如何用代理式AI服务去替代或增强人类层级的服务。这是整个经济体系里生产率最低的部分之一,从逻辑上说,采用AI是非常显而易见的选择。
但现实里有很多需要磨合的地方。我们经常讨论怎么合作、怎么推动。
比如我们现在运营着全国规模最大的直达型药房平台之一,是真正意义上的直销。业务才一年,但目前年化已经接近每季度10亿美元,也就是一年约40亿美元的药房销售规模。
于是很多团队会问:礼来能不能用你们的市场体量去推动这些新技术的采用?坦率讲,保险公司并不喜欢改变,医生群体对这些技术也会有顾虑。
但我们确实需要让美国医疗系统变得更高效。只有这样,才能为创新、为前沿药物的采用腾出空间,也才能对患者做更准确、更全面的评估。
与此同时,正如我之前提到的,过去十年里,即便没有把AI完整引入主流程,药物研发在一些新范式上的生产率其实也在加速。这令人兴奋,但仍处在早期阶段。
现在很多公司在把新旧范式组合起来,做成“双功能药物”:一端是靶向部分,另一端是“战斗部”,比如用于杀伤癌细胞。甚至只是更精准地把药物送到正确组织,本身就是一个非常重要、也非常难的工程问题。
今天我也见了几家在做不同路径探索的公司,对我来说都很有意思。这是药物制造这一侧。
我认为AI能在这里继续加速进展,而且已经有很多聪明的人在解决这些问题。
戴文睿 说真的,十年前、也就是你刚才问到的2017年,礼来的外界形象,可能还是一家有点“昏昏欲睡”的中西部公司。但给在座的计算机行业朋友们讲几个有意思的事实。
你可能不知道,第一个获批、由活体生物制造出来的生物技术药物——用最现代方式生产的药物,其实是礼来的产品Humulin,是我们和 Genentech 合作完成的。
黄仁勋 哦,那挺酷的。
戴文睿 然后在12年之后,礼来买下了制药行业里的第一台超级计算机,把它放在了印第安纳波利斯。那是一台Cray超算名字叫 Big Red,顺便一提,我们公司的标志也是红色的。
黄仁勋 Big Red。
戴文睿 对,很巧妙。后来我们正是在那台机器上,设计出了第一款由计算机设计并最终获批的药物——胰岛素赖脯胰岛素(Humalog)。直到今天,它仍然被全球大量一型和二型糖尿病患者使用。
当时我们基本上就是通过不断尝试不同的氨基酸序列,用相对经验的方法把这件事做成的。
黄仁勋 你一直在试图说服我,说礼来其实很酷。
戴文睿 我们确实很酷。
—— / Cong Ming Tou Zi Zhe / ——
排版:唐唐
责编:艾暄
转载开白,请联系小编微信【fanxiaocom】,添加备注“转载+新媒体名+姓名”,按顺序邀请加入媒体转载群
商务合作,请添加聪明牛牛【微信congmingtzz1/ 手机13262200706 】,添加备注“合作+机构名+姓名”具体商议
财报
2026-02-04
·微信
肺癌
1. Randomized Trial Links Time of Day to Immunotherapy Success.
给药时间显著影响肺癌免疫治疗效果。利用昼夜节律生物学优化治疗方案是提高癌症疗效的一种简单且低成本的方法。首个随机对照试验显示,早晨接受免疫检查点抑制剂治疗的晚期非小细胞肺癌患者生存期显著延长。该研究为临床实践中通过调整给药时间来改善癌症预后提供了前瞻性证据。
非小细胞肺癌 | 昼夜节律, 免疫反应 | 随机对照试验
Cancer discovery · 2026/02/02
原文链接:https://doi.org/10.1158/2159-8290.CD-NW2026-0010
2. NPM1 Mediates mRNA Sorting into Extracellular Vesicles via Specific RNA Motif Binding and Phase Separation.
NPM1通过相分离机制介导mRNA分选进入细胞外囊泡。mRNA如何被特异性选择并包装进细胞外囊泡的机制尚不完全清楚。研究鉴定出NPM1能结合特定RNA基序并诱导相分离,从而促进mRNA装载。该发现揭示了囊泡运输的新分子机制,并为非小细胞肺癌的治疗提供了潜在靶点。
非小细胞肺癌 | NPM1, 相分离 | 细胞外囊泡分析
Zhang Kaixiang · … · Zhang Ying · · 2026/02/03
该研究由细胞生物学清华大学张瑛团队完成 Advanced science (Weinheim, Baden-Wurttemberg, Germany)
原文链接:https://doi.org/10.1002/advs.202514852
3. Metabolic characterization of tumor-immune interactions by multiplexed immunofluorescence reveals spatial mechanisms of immunotherapy response in non-small cell lung carcinoma (NSCLC).
多重免疫荧光揭示非小细胞肺癌免疫治疗响应的空间代谢机制。尽管免疫检查点抑制剂改善了部分肺癌患者预后,但耐药性仍是主要挑战。研究利用多重免疫荧光技术分析患者活检样本,结合深度学习模型评估肿瘤及免疫细胞的功能与代谢状态。结果发现细胞间的空间邻近性及其代谢背景是预测无进展生存期的关键特征,模型预测准确率达0.8。这些空间见解深化了对肿瘤微环境的理解,为优化免疫治疗策略提供了新视角。
非小细胞肺癌(NSCLC) | 肿瘤微环境, 细胞空间相互作用, 代谢状态 | 多重免疫荧光(mIF), 深度学习
Monkman James · … · Kulasinghe Arutha · · 2026/02/03
该研究由空间生物学与肿瘤免疫昆士兰大学Arutha Kulasinghe团队完成 Nature communications
原文链接:https://doi.org/10.1038/s41467-026-68633-8
4. Lung cancer vaccines to enhance immune checkpoint inhibitor therapy: evidence and future perspectives.
肺癌疫苗助力克服免疫检查点抑制剂耐药。尽管ICI改变了肺癌治疗,但耐药性仍是限制其长期临床获益的主要障碍。本综述探讨了癌症疫苗如何通过诱导新抗原特异性免疫反应来增强ICI的抗肿瘤效能。联合应用疫苗与ICI有望成为未来肺癌个体化免疫治疗的重要方向。
肺癌免疫耐药 | 肿瘤新抗原, 免疫检查点 | 癌症疫苗, 联合疗法
Tang Zhiting · … · Li Tianhong · · 2026/02/03
该研究由肿瘤学加州大学戴维斯分校李天红团队完成 Journal of hematology & oncology
原文链接:https://doi.org/10.1186/s13045-026-01778-7
乳腺癌
1. Associations of the HER2DX Genomic Test with Biological and Pathologic Features in HER2-Positive Breast Cancer.
HER2DX基因检测预测乳腺癌病理完全缓解。HER2阳性乳腺癌治疗决策需要更精准的生物学评估工具。研究证实HER2DX评分与多种组织病理学特征显著相关,且是新辅助治疗后病理完全缓解的独立预测因子。该研究支持将HER2DX用于乳腺癌的个体化治疗决策,优化临床管理。
HER2阳性乳腺癌 | ERBB2, 免疫特征 | 基因组检测, 逻辑回归分析
Sanfeliu Esther · … · Prat Aleix · · 2026/02/04
该研究由转化基因组学巴塞罗那医院Prat Aleix团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-3123
2. Addition of Ipatasertib to Dual Anti-HER2 Maintenance Therapy in HER2-Positive Metastatic Breast Tumors with PIK3CA Mutations: The Phase Ib SOLTI-1507 IPATHER Trial.
Ipatasertib联合方案治疗PIK3CA突变乳腺癌。针对伴有PIK3CA突变的HER2阳性转移性乳腺癌,亟需探索更有效的维持治疗策略。Ib期临床试验显示,AKT抑制剂Ipatasertib联合双靶向治疗具有良好的安全性和初步疗效。该研究为PIK3CA突变型乳腺癌患者提供了一种安全且有前景的精准治疗新选择。
HER2阳性转移性乳腺癌 | PIK3CA突变, AKT抑制剂 | Ib期临床试验
Oliveira Mafalda · … · Saura Cristina · · 2026/02/04
该研究由肿瘤内科学瓦尔德希布伦肿瘤研究所Saura Cristina团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-2298
3. A Phase Ib Study of Sapacitabine and Olaparib in Patients with BRCA1/2-Mutated Metastatic Breast Cancer.
Sapacitabine联合奥拉帕利治疗BRCA突变乳腺癌。针对BRCA突变的转移性乳腺癌,探索PARP抑制剂与其他药物的联合方案具有重要意义。Ib期研究显示,该联合方案在PARP抑制剂初治患者中表现出50%的客观缓解率,尽管存在血液学毒性。该研究提示了联合用药的潜在获益,并为优化给药方案提供了临床依据。
BRCA突变乳腺癌 | PARP抑制剂, 同源重组修复缺陷 | Ib期临床试验
Lynce Filipa · … · Tolaney Sara M · · 2026/02/04
该研究由内科肿瘤学丹娜-法伯癌症研究所Tolaney Sara M团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-0571
4. PI3K inhibition in combination with tamoxifen in patients with metastatic HR+/HER2- breast cancer: clinical and circulating tumor DNA results.
PI3K抑制剂联合他莫昔芬治疗乳腺癌。针对内分泌治疗耐药的HR+/HER2-乳腺癌,亟需探索新型靶向联合疗法。POSEIDON临床试验显示taselisib联合方案可改善无进展生存期,且ctDNA肿瘤分数具有预后价值。尽管毒性限制了临床应用,但该研究为优化靶向治疗选择提供了生物标志物参考。
转移性乳腺癌 | PI3K通路, 内分泌耐药 | 临床II期试验, ctDNA分析
Voorthuis Rosie A B · … · Baird Richard D · · 2026/02/03
该研究由肿瘤学剑桥大学Baird Richard D团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-2833
5. ITGB1 Regulates Triple-Negative Breast Cancer Development by Modulating the Tumor Microenvironment.
肿瘤内在ITGB1通过调节微环境促进三阴性乳腺癌进展。理解癌细胞与免疫微环境的相互作用对于开发有效的免疫疗法至关重要。研究通过体内CRISPR筛选发现ITGB1通过重塑髓系细胞群来构建促癌微环境。靶向ITGB1的新型抗体显示出显著的抗肿瘤潜力。
三阴性乳腺癌 | ITGB1, 肿瘤微环境 | CRISPR筛选
Song Nuozi · … · Feng Mingye · · 2026/02/03
该研究由肿瘤免疫学希望之城国家医疗中心冯明野团队完成 Advanced science (Weinheim, Baden-Wurttemberg, Germany)
原文链接:https://doi.org/10.1002/advs.202513672
6. Genetically engineered cellular membrane-camouflaged nanoparticles amplify immune response against recurrent metastatic triple-negative breast cancer.
细胞膜包裹纳米颗粒增强三阴性乳腺癌免疫治疗。有氧糖酵解驱动肿瘤生长并抑制免疫,是复发转移性乳腺癌的治疗难点。研究开发了PD-1修饰的生物仿生纳米平台,封装糖酵解抑制剂3BP以实现代谢-免疫双重干预。该策略有效抑制了肿瘤复发和转移并延长生存期,为PD-L1阳性恶性肿瘤提供了新范式。
三阴性乳腺癌 | 糖酵解抑制, PD-L1/PD-1通路 | 细胞膜仿生纳米颗粒
Yang Yun · … · Qian Zhiyong · · 2026/01/31
该研究由生物治疗四川大学华西医院钱志勇团队完成 Biomaterials
原文链接:https://doi.org/10.1016/j.biomaterials.2026.124026
7. Cyclic di-GMP suppresses cancer metastasis by targeting proteasome 26S subunit non-ATPase 3 independently of STING.
环二鸟苷酸通过靶向PSMD3抑制癌症转移。癌症转移是导致死亡的主因,亟需开发能有效阻断转移过程的新型治疗药物。研究发现细菌信号分子c-di-GMP能直接结合PSMD3并阻断其与TBK1的相互作用,从而抑制NF-κB通路。该研究揭示了PSMD3在乳腺癌中的促癌新功能,并证实了c-di-GMP作为抗转移药物的巨大潜力。
三阴性乳腺癌, 癌症转移 | c-di-GMP, PSMD3-TBK1-NF-κB轴 | 蛋白质相互作用, 转移模型
Wang Jieqiong · … · Lu Hua · · 2026/02/04
该研究由生物化学与分子生物学杜兰大学医学院卢华团队完成 Signal transduction and targeted therapy
原文链接:https://doi.org/10.1038/s41392-025-02553-9
8. LAP2α drives breast tumorigenesis by mitigating replication stress.
LAP2α通过缓解复制压力驱动乳腺癌进展。RPA蛋白在基因组监控中起核心作用,但其在肿瘤进展中的具体调控机制仍不完全清楚。研究发现LAP2α能直接促进RPA加载至单链DNA,从而对抗复制压力诱导的损伤并促进肿瘤生长。该研究确立了LAP2α在乳腺癌发生中的关键地位,并将其识别为潜在的治疗干预靶点。
乳腺癌, 基因组稳定性 | LAP2α, RPA加载 | 复制压力分析, 肿瘤生长模型
Ma Yanhui · … · Bao Kaiwen · · 2026/02/03
该研究由基础医学天津医科大学鲍凯文团队完成 Cell death & disease
原文链接:https://doi.org/10.1038/s41419-026-08433-6
血液肿瘤
1. Allogeneic HSCT for consolidation in pediatric refractory or relapsed ALK-positive anaplastic large cell lymphoma.
异基因造血干细胞移植有效巩固儿童ALCL疗效。对于复发性ALK阳性间变性大细胞淋巴瘤,最佳的移植预处理方案仍需探讨。研究分析了57名患儿,发现无TBI的预处理方案与非亲缘供者结合ATLG可获得优异的生存率。该结果支持在儿童ALCL复发治疗中采用更优的移植策略。
间变性大细胞淋巴瘤 | 移植巩固治疗 | 异基因造血干细胞移植, 预处理方案
Knörr Fabian · … · Woessmann Wilhelm · · 2026/02/03
该研究由血液肿瘤学汉堡-埃彭多夫大学医学中心Woessmann Wilhelm团队完成 Blood advances
原文链接:https://doi.org/10.1182/bloodadvances.2025018774
2. Proteasome Subunit PSMD1 is a Key Therapeutic Target in Multiple Myeloma.
蛋白酶体亚基PSMD1成为多发性骨髓瘤治疗新靶点。PSMD1在多发性骨髓瘤中高表达且与预后不良密切相关。研究发现利用脂质纳米颗粒递送PSMD1 siRNA可显著抑制肿瘤生长并克服耐药性。该研究为开发针对骨髓瘤及其他恶性肿瘤的PSMD1靶向疗法奠定了基础。
多发性骨髓瘤 | PSMD1, 蛋白酶体抑制剂 | siRNA脂质纳米颗粒, 蛋白质组学
Du Ting · … · Anderson Kenneth C · · 2026/02/03
该研究由血液肿瘤学丹娜-法伯癌症研究所Anderson Kenneth C团队完成 Blood
原文链接:https://doi.org/10.1182/blood.2025029358
3. Efficacy and safety of autologous CD5-KO anti-CD5 CAR-T cells in relapsed/refractory CD5+ hematological malignancies.
CD5敲除的CAR-T细胞治疗CD5阳性血液肿瘤的临床研究。针对T细胞抗原的CAR-T疗法需防止同类相残。这项I期临床试验评估了CD5基因缺失的CT125A细胞,在复发/难治性患者中实现了85.7%的总缓解率。研究证实了该疗法的潜力,同时也指出了需关注的自身免疫相关副作用。
血液肿瘤 | CD5, CAR-T细胞疗法 | CRISPR基因敲除, I期临床试验
Cheng Jiali · … · Zhu Xiaojian · · 2026/02/02
该研究由血液学华中科技大学同济医院朱晓健团队完成 Cell reports. Medicine
原文链接:https://doi.org/10.1016/j.xcrm.2026.102584
4. DOT1L provides transcriptional memory through PRC1.1 antagonism.
DOT1L通过拮抗PRC1.1维持白血病转录记忆。DOT1L和Menin是白血病致癌蛋白的关键辅助因子,但其抑制剂发挥疗效的深层机制仍待阐明。研究发现DOT1L介导的H3K79甲基化能直接拮抗PRC1.1活性,抑制其导致的基因沉默,从而维持致癌基因的表达。该研究揭示了白血病中转录记忆的维持机制,为优化表观遗传疗法提供了新见解。
急性髓系白血病 (AML) | DOT1L, PRC1.1, H3K79甲基化 | 表观遗传抑制剂, 染色质免疫共沉淀
Neville Daniel · … · Gilan Omer · Nature cell biology · 2026/02/03
⭐ 该研究由血液病学澳大利亚莫纳什大学Gilan Omer团队完成
原文链接:https://doi.org/10.1038/s41556-025-01859-8
消化肿瘤
1. Glypican-3-Targeted PET Imaging for Precise Diagnosis of Hepatocellular Carcinoma: From Bench to Bedside.
新型GPC3靶向PET示踪剂实现肝细胞癌的精准诊断。肝细胞癌的早期精准诊断对预后至关重要,但现有影像技术在特异性上仍有不足。本研究开发了[68Ga]Ga-XH-06示踪剂,并在临床前及首例人体研究中证实了其高对比度显像能力。该技术为GPC3阳性肝癌的无创评估和精准管理提供了极具前景的临床工具。
肝细胞癌(HCC) | Glypican-3 (GPC3) | PET/MRI, 放射性示踪剂
Lin Zhaoguo · … · Lan Xiaoli · · 2026/02/04
该研究由核医学华中科技大学同济医学院附属协和医院兰晓莉团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-1856
2. Ibrutinib and PD-1 Blockade Potentiate Mesothelin-Targeting CAR T-cell Therapy in Preclinical Models of Pancreatic Cancer.
依鲁替尼与PD-1阻断剂可增强间皮素靶向CAR-T治疗胰腺癌的疗效。胰腺癌的免疫抑制环境和致密间质严重阻碍了CAR-T细胞的浸润与功能。研究通过临床前模型证明,依鲁替尼能促进CAR-T细胞扩增和Th1极化,联合PD-1阻断可进一步提升抗肿瘤活性。该研究为优化胰腺癌CAR-T疗法提供了可临床转化的联合策略。
胰腺导管腺癌(PDAC) | 间皮素(MSLN), 依鲁替尼, PD-1阻断 | CAR-T细胞疗法, 双靶向平台
Armstrong Alexander · … · Escobar Giulia · · 2026/02/04
该研究由肿瘤学马萨诸塞州总医院Escobar Giulia团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-2907
3. Distinct Spatially Resolved Tumor Microenvironment Trajectories Define Benefit from Ramucirumab plus Pembrolizumab in Refractory PD-L1+ Gastric Cancer.
空间解析的肿瘤微环境特征决定胃癌双靶向治疗的获益。VEGF与PD-1双靶向疗法在实体瘤中备受关注,但影响疗效的关键特征尚不明确。通过数字空间谱分析发现,响应者具有更高的基线VEGF信号和更近的瘤-免空间距离。该研究揭示了治疗压力下的微环境重塑规律,为胃癌精准治疗提供了空间生物标志物。
难治性PD-L1+胃癌 | VEGFR2, PD-1, 血管正常化 | 数字空间谱(DSP), 临床试验
Lim Sung Hee · … · Lee Jeeyun · · 2026/02/03
该研究由肿瘤学成均馆大学医学院Lee Jeeyun团队完成 Cancer immunology research
原文链接:https://doi.org/10.1158/2326-6066.CIR-25-0625
4. Tislelizumab and Hypofractionated Radiotherapy plus Nab-Paclitaxel/Gemcitabine as Conversion Therapy for BRPC/LAPC: A Phase II Trial with Dynamic Biomarker Monitoring.
替雷利珠单抗联合放化疗助力胰腺癌转化治疗。交界可切除或局部晚期胰腺癌的最佳转化治疗方案尚不明确。II期临床试验表明,THAG方案具有显著的临床活性和可控的安全性,并能提高根治性手术切除率。该研究结合动态生物标志物监测,为胰腺癌的精准转化治疗提供了重要依据。
胰腺癌 | PD-1抑制剂, IL6 | 转化治疗, 循环肿瘤DNA
Ni Jiayao · … · Du Juan · · 2026/02/04
该研究由肿瘤学南京鼓楼医院Du Juan团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-2461
5. Clinical Translation of Antibody Positron Emission Tomography for Cancer Imaging.
新型PET放射示踪剂助力肝细胞癌精准诊断。肝细胞癌缺乏精确的非侵入性诊断工具,影响了患者的早期发现与治疗。研究开发了针对GPC3的抗体片段示踪剂[68Ga]Ga-XH-06,成功实现了患者体内肿瘤的可视化。该技术满足了肝癌诊断的重要临床需求,具有显著的临床转化价值。
肝细胞癌 | GPC3 | 正电子发射断层扫描(PET), 放射性示踪剂
Seo Youngho · Flavell Robert R · · 2026/02/04
该研究由放射学与生物医学成像加州大学旧金山分校Flavell Robert R团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-3784
6. Pancreatic Cancer Organoids Recapitulate Chemotherapy Response and Identify a Potent Cytotoxic T-Cell Population.
胰腺癌类器官预测化疗反应并识别高效T细胞。胰腺导管腺癌预后极差,亟需个体化精准治疗方案。研究成功建立了患者来源的类器官(PDO),其药物敏感性与临床预后高度相关,并利用PDO筛选出具有强效抗肿瘤活性的γδ T细胞。该研究证实了PDO在临床相关时间内指导精准治疗及免疫疗法开发的巨大潜力。
胰腺导管腺癌 | 肿瘤浸润淋巴细胞(TIL) | 患者来源类器官(PDO), 药物筛选
Anderko Renee R · … · Zureikat Amer H · · 2026/02/04
该研究由外科肿瘤学匹兹堡大学Zureikat Amer H团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-1110
7. Transcriptomic landscape of transposable elements reveals LTR7-PLAAT4 as a potential oncogene and therapeutic target in pancreatic adenocarcinoma.
转座元件LTR7-PLAAT4作为胰腺癌新靶点的发现。转座元件(TEs)的失调会威胁基因组稳定性,并可能在癌症进展中发挥作用。研究通过分析胰腺癌类器官,鉴定出LTR7-PLAAT4融合转录本通过促进脂质蓄积推动肿瘤进展,并发现其衍生的免疫肽具有强免疫原性。该发现为胰腺癌的分子分型和免疫治疗提供了潜在的新靶点。
胰腺腺癌 | 转座元件, LTR7-PLAAT4 | 类器官分析, 免疫肽组学
Shi Meilong · … · Li Jing · · 2026/02/03
该研究由肿瘤学/免疫学海军军医大学/上海理工大学李静团队完成 Genome research
原文链接:https://doi.org/10.1101/gr.280528.125
8. Burden of primary liver cancer and underlying etiologies among young adults from 1990 to 2021 and modelled Projection to 2050.
全球中青年原发性肝癌负担及病因演变分析。尽管肝癌死亡率有所下降,但中青年群体的发病率和患病率在过去三十年间显著上升。研究通过分析1990-2021年全球数据发现,代谢风险和酒精摄入已逐渐取代乙肝成为肝癌的主要诱因。东亚地区负担最重,且男性吸烟相关风险远高于女性。该研究强调了早期筛查的重要性,并建议将资源分配从传统病毒防治转向代谢和行为风险干预。
原发性肝癌, 中青年健康 | 代谢风险, 酒精摄入, 乙肝病毒 | 流行病学分析, GBD数据
Liu Qi · … · Zhao Xiaofang · · 2026/01/31
该研究由肿瘤学郑州大学第一附属医院Zhao Xiaofang团队完成 Cancer letters
原文链接:https://doi.org/10.1016/j.canlet.2026.218272
9. TRIM47 Regulates Energy Metabolism via Glycolytic Reprogramming to Drive Hepatocellular Carcinoma Progression and Represents an Efficient Therapeutic Target.
TRIM47通过重塑糖代谢驱动肝癌进展。异常能量代谢是肝癌快速增殖的关键,但其调控机制仍需深入探讨。研究发现TRIM47通过泛素化降解FBP1来促进糖解重编程,从而驱动肝癌生长与转移。开发靶向TRIM47的纳米药物在动物模型中展现了良好的治疗潜力。
肝细胞癌 | TRIM47, FBP1泛素化, 糖解重编程 | siRNA纳米药物
Sun Weijie · … · Gao Yufeng · · 2026/02/03
该研究由传染病学安徽医科大学第一附属医院高勇峰团队完成 Advanced science (Weinheim, Baden-Wurttemberg, Germany)
原文链接:https://doi.org/10.1002/advs.202416996
10. TGFβ1 Activates Lnc-APUE to Promote Tumor Metastasis via the Alu Element-Driven STAU1-Mediated Decay of CDH1 mRNA.
揭示TGFβ1诱导肝癌转移的新机制。长非编码RNA中的Alu元素功能尚不明确,其在肿瘤转移中的作用亟待探索。研究发现TGFβ1激活的lnc-APUE通过Alu元素介导CDH1 mRNA降解,导致E-cadherin缺失并促进转移。该研究定义了调控癌症转移的全新信号轴。
肝癌转移 | lnc-APUE, Alu元素, STAU1介导的mRNA降解 | 异种移植模型
Li Song-Yang · … · Zhuang Shi-Mei · · 2026/02/03
该研究由肿瘤学中山大学庄诗美团队完成 Advanced science (Weinheim, Baden-Wurttemberg, Germany)
原文链接:https://doi.org/10.1002/advs.202518731
11. Recurrent resistance mutations to lirafugratinib inform treatment sequencing in FGFR2-driven tumors.
序贯使用不可逆FGFR抑制剂克服耐药。FGFR2驱动肿瘤在使用可逆抑制剂后易产生耐药突变,限制了后续治疗策略。研究通过ctDNA和外显子测序发现lirafugratinib耐药伴随特定激酶域突变,且与futibatinib具有互补性。该发现支持根据精准耐药突变检测开展序贯治疗,为胆管癌患者提供新方案。
肝内胆管癌 | FGFR2突变, 获得性耐药 | ctDNA监测, 序贯治疗
Facchinetti Francesco · … · Friboulet Luc · · 2026/02/03
该研究由肿瘤学古斯塔夫·鲁西研究所Friboulet Luc团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-4390
12. Uncertainty-guided Iterative Contrastive Fusion for Reliable Survival Prediction in Rectal Cancer.
迭代对比融合框架提升直肠癌生存预测可靠性。多模态影像与临床数据整合面临模态异质性和站点差异的挑战。研究提出UICSurv框架,通过不确定性引导的迭代对比学习捕捉跨站点多模态交互。该方法在生存预测和不确定性估计方面均表现出显著优势。
直肠癌 | 生存预测 | 迭代对比融合, 不确定性估计
Bao Qingsen · … · Shen Dinggang · · 2026/02/03
该研究由医学影像人工智能上海交通大学沈定刚团队完成 IEEE transactions on medical imaging
原文链接:https://doi.org/10.1109/TMI.2026.3660270
13. Biophysical approaches to cancer diagnostics: Collagen biomarkers in colorectal cancer.
胶原蛋白生物标志物助力结直肠癌生物物理诊断。细胞外基质中的胶原重塑是癌症进展的关键特征,直接影响组织的力学和光学属性。本文综述了利用光谱和光学技术原位探测胶原结构变化及其与宏观生物力学联系的最新进展。这些技术结合机器学习算法,有望显著提升肿瘤边缘检测等临床诊断性能。
结直肠癌 | 胶原重塑, 细胞外基质 | 多模态光谱技术, 机器学习
Biswas Shimul · Mahato Krishna Kishore · Physics of life reviews · 2026/01/29
⭐ 该研究由生物物理学马尼帕尔高等教育学院Krishna Kishore Mahato团队完成
原文链接:https://doi.org/10.1016/j.plrev.2026.01.009
14. Noninvasive imaging-based assessment of tumor-associated neutrophils for prognosis and immunotherapy response in gastric cancer: a multicenter study.
CT影像组学实现胃癌中性粒细胞无创评估。肿瘤相关中性粒细胞(TAN)促进胃癌进展,但传统活检无法实现连续监测。研究开发了基于CT影像的集成机器学习生物标志物EnmlbaRB,可准确映射TAN浸润状态。该工具能有效预测胃癌患者预后及免疫治疗反应,助力个性化精准治疗。
胃癌, 肿瘤相关中性粒细胞 | 免疫治疗反应, 预后分层 | 影像组学, 机器学习
Zheng Wenbo · … · Jiang Yuming · · 2026/02/01
该研究由放射肿瘤学维克森林大学医学院Jiang Yuming团队完成 Journal of advanced research
原文链接:https://doi.org/10.1016/j.jare.2026.01.082
15. Escherichia coli promotes colorectal cancer metastasis by maintaining enhancer-promoter loops through releasing neutrophil extracellular traps.
大肠杆菌通过诱导中性粒细胞胞外陷阱促进结直肠癌转移。肠道微生物在结直肠癌肝转移(CRCLM)中的作用机制尚未完全阐明。研究发现大肠杆菌在肝转移组织中富集,并通过RIPK2/HNRNPK通路诱导中性粒细胞形成胞外陷阱(NETs)。NETs进而通过增加癌细胞中TRPC1和NFATC3的表达,稳定STAT3相关的增强子-启动子环路,从而强化Tns1转录并促进转移。该研究阐明了细菌诱导的表观遗传修饰在肿瘤进展中的作用,为CRCLM的防治提供了新靶点。
结直肠癌肝转移(CRCLM) | 中性粒细胞胞外陷阱(NETs), 大肠杆菌, 增强子-启动子环路 | 2bRAD-M测序, 多组学分析
Pan Banglun · … · Tang Nanhong · · 2026/02/03
该研究由肝胆外科福建医科大学附属协和医院汤南鸿团队完成 Nature communications
原文链接:https://doi.org/10.1038/s41467-026-69005-y
16. BCAT1-elicited branched-chain amino acid catabolism fosters neutrophil-mediated pan-metastasis in pancreatic ductal adenocarcinoma.
BCAT1介导的支链氨基酸代谢促进胰腺癌全转移。胰腺导管腺癌(PDAC)具有极强的侵袭性,但其通过多种途径发生全转移的代谢免疫机制尚不明确。研究发现BCAT1在转移相关肿瘤细胞中高表达,通过催化亮氨酸代谢激活mTOR通路,进而招募中性粒细胞构建促转移微环境。该研究揭示了BCAT1+细胞与中性粒细胞间的代谢-免疫互作,为抑制PDAC全转移提供了潜在靶点。
胰腺导管腺癌 | BCAT1, 中性粒细胞招募 | 代谢-免疫互作分析
Xie Fan · … · Lao Xiang-Ming · · 2026/02/02
该研究由胰腺外科中山大学肿瘤防治中心Lao Xiang-Ming团队完成 Molecular therapy : the journal of the American Society of Gene Therapy
原文链接:https://doi.org/10.1016/j.ymthe.2026.01.039
17. Novel Combination of Irreversible Electroporation and Allogenic Chimeric Antigen Receptor T-Cell Therapy Synergizes Therapeutic Outcomes in a Preclinical Human Pancreatic Cancer Mouse Model.
不可逆电穿孔联合CAR-T疗法协同治疗胰腺癌。不可逆电穿孔(IRE)是治疗不可切除肿瘤的临床手段,但如何克服实体瘤对过继性细胞疗法的阻碍仍是难题。研究在胰腺癌小鼠模型中首次证明,IRE能改善肿瘤微环境并促进CAR-T细胞浸润,且不影响存活肿瘤细胞上的靶点表达。这种联合策略实现了两种临床技术的协同增效,为实体瘤的综合治疗提供了新方案。
胰腺癌 | 肿瘤微环境调节 | 不可逆电穿孔(IRE), CAR-T细胞疗法
Jacobs Edward · … · Davalos Rafael · · 2026/02/02
该研究由生物医学工程佐治亚理工学院Davalos Rafael团队完成 Research (Washington, D.C.)
原文链接:https://doi.org/10.34133/research.1105
18. RBM15 Enhances 5-Fluorouracil Drug Sensitivity and Suppresses Gastric Cancer Progression by Modulating N6-Methyladenosine Modification of ECT2-Dependent IGF2BP3.
RBM15通过调节ECT2的m6A修饰抑制胃癌进展并增强化疗敏感性。胃癌是全球癌症死亡的主要原因,寻找影响预后和化疗敏感性的分子标志物至关重要。研究发现RBM15在胃癌中高表达预示良好预后,其通过介导ECT2 mRNA的m6A修饰并影响IGF2BP3结合,从而抑制肿瘤迁移和增强5-FU敏感性。该研究揭示了RBM15/IGF2BP3-ECT2信号轴在胃癌中的调控作用,为胃癌治疗提供了新靶点。
胃癌 | m6A甲基化, RBM15/ECT2轴 | MeRIP-seq, 5-FU敏感性分析
Zhu Xingyu · … · Li Leping · · 2026/02/02
该研究由胃肠外科山东第一医科大学附属省立医院Li Leping团队完成 Research (Washington, D.C.)
原文链接:https://doi.org/10.34133/research.1108
妇科肿瘤
1. Repositioning GLP-1 Receptor Agonists in Endometrial Cancer: Molecular Rationale, Preclinical Insights, and Translational Opportunities.
GLP-1受体激动剂在子宫内膜癌中的应用前景。肥胖相关的子宫内膜癌发病率上升,急需针对代谢功能障碍和肿瘤生物学的新疗法。研究综述了GLP-1R激动剂通过激活cAMP-PKA等通路发挥抗增殖作用,并可能克服激素耐药。该类药物代表了靶向肥胖环境和肿瘤内在通路的一种极具潜力的治疗策略。
子宫内膜癌 | GLP-1受体, cAMP-PKA通路 | 药物重定位
Podder Vivek · … · Slomovitz Brian M · · 2026/02/04
该研究由妇科肿瘤学西奈山医疗中心Slomovitz Brian M团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-2819
2. Vitamin D and Uterine Fibroids: Insights into Pathophysiology and Therapeutic Potential.
维生素D在子宫肌瘤防治中的作用。子宫肌瘤是育龄女性高发的良性肿瘤,其发病机制与维生素D缺乏密切相关。综述阐述了维生素D通过抗增殖、诱导凋亡及调节酶活性来抑制肌瘤生长的多重机制。该研究肯定了维生素D作为管理和预防子宫肌瘤候选药物的潜力。
子宫肌瘤 | 维生素D受体, 细胞凋亡 | 临床综述
Ciebiera Michał · … · Al-Hendy Ayman · Endocrine reviews · 2026/02/03
⭐ 该研究由妇产科学芝加哥大学Al-Hendy Ayman团队完成
原文链接:https://doi.org/10.1210/endrev/bnaf043
3. Mapping CSC-Mediated Ovarian Cancer Chemoresistance via CXCR4-PET to Guide Precision Cisplatin Re-Sensitization Therapy.
CXCR4影像引导的卵巢癌精准治疗。针对癌症干细胞的疗法是减少耐药和复发的关键,但缺乏有效的靶向标记。研究证实CXCR4是卵巢癌干细胞的功能标志物,并开发了PET成像引导的精准化疗增敏方案。该模式为克服卵巢癌顺铂耐药提供了临床转化新路径。
卵巢癌耐药, 癌症干细胞 | CXCR4, 顺铂增敏 | PET成像, PDX模型
Feng Lixia · … · Cai Jing · · 2026/02/03
该研究由妇产科学华中科技大学同济医学院附属协和医院蔡静团队完成 Advanced science (Weinheim, Baden-Wurttemberg, Germany)
原文链接:https://doi.org/10.1002/advs.202521279
4. A 3D ovarian cancer metastasis model using a decellularised peritoneal matrix to study therapy response.
脱细胞腹膜基质构建卵巢癌转移3D模型。高级别浆液性卵巢癌的腹膜转移具有独特的肿瘤微环境,影响治疗响应。研究利用猪脱细胞腹膜基质构建了模拟人类生理的3D模型,支持肿瘤细胞生长并保持基质完整性。该平台可用于评估化疗及免疫疗法的敏感性,助力开发新型腹膜转移癌治疗方案。
卵巢癌腹膜转移 | 肿瘤微环境, 细胞外基质 | 脱细胞基质, 3D细胞培养模型
Gjerde Christiane Helgestad · … · McCormack Emmet · · 2026/02/02
该研究由肿瘤生物标志物卑尔根大学Emmet McCormack团队完成 EBioMedicine
原文链接:https://doi.org/10.1016/j.ebiom.2026.106135
5. The long noncoding RNA AC093895.1 promotes ovarian cancer formation and metastasis through a positive feedback network dependent on the transcription factor SOX4.
LncRNA AC093895.1通过SOX4反馈环路促进卵巢癌进展。卵巢癌的复发和转移是导致患者死亡的主要原因,寻找有效的预后标志物和治疗靶点至关重要。研究发现AC093895.1在卵巢癌中高表达,通过竞争性结合miR-1253上调SOX4,而SOX4反之促进其转录,形成正反馈环路。该研究揭示了AC093895.1在肿瘤生长和转移中的关键作用,为其作为卵巢癌预后生物标志物和治疗靶点提供了依据。
卵巢癌转移 | AC093895.1, SOX4, miR-1253 | 染色质免疫共沉淀(ChIP), 竞争性内源RNA(ceRNA)分析
Huang Bin · … · Lin Jiumao · · 2026/02/03
该研究由中西医结合肿瘤学福建中医药大学林久茂团队完成 Cell death & disease
原文链接:https://doi.org/10.1038/s41419-026-08429-2
6. Development and validation of an artificial intelligence-based model for diagnosing benign, borderline, and malignant adnexal masses.
深度学习模型提升附件肿块良恶性诊断准确率。准确区分附件肿块的良性、交界性与恶性对临床管理至关重要,但极具挑战性。研究开发了集成超声影像与临床指标的AI模型,在多中心验证中表现出与专家相当的诊断性能,并显著提升了放射科医师的一致性。该模型作为决策支持工具,在医疗资源匮乏地区具有重要的临床应用价值。
卵巢肿瘤, 附件肿块 | 影像组学特征 | 深度学习, 多模态数据融合
Wu Yingnan · … · Sun Litao · · 2026/02/03
该研究由超声医学浙江省人民医院孙立涛团队完成 NPJ precision oncology
原文链接:https://doi.org/10.1038/s41698-026-01320-5
神经肿瘤
1. SOX2 phosphorylation during mitosis limits genomic damage.
有丝分裂期间SOX2磷酸化可防止基因组损伤。先驱转录因子SOX2在干细胞和癌症中高表达,但其在细胞分裂中的调控机制不明。研究发现SOX2在有丝分裂时的磷酸化能限制其染色质结合,防止染色质异常开放。该研究揭示了SOX2在维持基因组稳定性方面的新功能,为癌症治疗提供了新靶点。
胶质母细胞瘤, 基因组稳定性 | SOX2, 磷酸化, 先驱转录因子 | 质谱分析, 染色质免疫沉淀
Williams Charles A C · … · Pollard Steven M · · 2026/02/02
该研究由再生医学爱丁堡大学Pollard Steven M团队完成 Genes & development
原文链接:https://doi.org/10.1101/gad.352664.125
2. Potent synthetic lethality between PLK1 and EYA family inhibitors in tumors of the central and peripheral nervous systems.
PLK1与EYA家族抑制剂在神经系统肿瘤中具有强效合成致死作用。EYA家族蛋白在多种神经系统癌症中异常表达,是潜在的治疗靶点。研究发现EYA能增加活性PLK1池,联合抑制两者可显著诱导有丝分裂停滞和细胞死亡,尤其针对癌症干细胞。该发现为胶质母细胞瘤和神经母细胞瘤提供了一种新型合成致死疗法。
神经母细胞瘤, 胶质母细胞瘤 | PLK1, EYA1/4, 合成致死 | 癌症干细胞模型, 多组学分析
Nelson Christopher B · … · Pickett Hilda A · · 2026/02/02
该研究由肿瘤学悉尼大学Pickett Hilda A团队完成 Genes & development
原文链接:https://doi.org/10.1101/gad.353271.125
3. Lymphotropic Virotherapy Induces DC and High Endothelial Venule Inflammation, Promoting the Antitumor Efficacy of Intratumor Virus Administration.
淋巴靶向病毒疗法通过重塑淋巴结炎症增强胶质瘤治疗效果。肿瘤引流淋巴结在抗肿瘤免疫中至关重要,但其在病毒疗法中的作用尚未被充分利用。研究发现通过颈部周淋巴灌注病毒可诱导淋巴结炎症重塑,显著提升T细胞激活和抗肿瘤效能。该研究将淋巴系统确立为病毒疗法的新靶点,为脑肿瘤免疫治疗提供了新策略。
胶质瘤, 肿瘤免疫监测 | I型干扰素(IFNI), 树突状细胞, 淋巴结重塑 | 溶瘤病毒疗法, 颈部周淋巴灌注
Ludwig Andrea L · … · Gromeier Matthias · · 2026/02/03
该研究由神经外科学杜克大学Gromeier Matthias团队完成 Cancer immunology research
原文链接:https://doi.org/10.1158/2326-6066.CIR-25-0756
4. RETRACTION: Histone Lactylation-Driven Upregulation of VRK1 Expression Promotes Stemness and Proliferation of Glioma Stem Cells.
撤回关于VRK1促进胶质瘤干细胞的研究。该文章因在基因筛选过程中发现非故意统计错误,导致VRK1作为候选基因的可靠性受损。作者与期刊达成一致撤回稿件,以确保科学记录的准确性。此撤稿强调了在多数据库基因筛选中统计严谨性的重要性。
胶质瘤干细胞 | 组蛋白乳酸化, VRK1 | 撤稿声明
Advanced science (Weinheim, Baden-Wurttemberg, Germany) · 2026/02/03
原文链接:https://doi.org/10.1002/advs.74235
5. Hijacking the Hydrogen Sulfide Axis: A Novel 4-Trifluoromethylquinoline Derivative Suppresses Glioblastoma via Cystathionine γ-Lyase Suppression.
新型喹啉衍生物通过抑制CTH活性抑制胶质母细胞瘤。CTH在胶质母细胞瘤中富集并与耐药性相关,但缺乏有效的抑制剂。研究鉴定出TKL002能直接靶向CTH,减少硫化氢产生并抑制NF-κB信号通路。该发现证明了TKL002作为克服胶质母细胞瘤侵袭性的治疗候选药物的潜力。
胶质母细胞瘤 | CTH, 硫化氢 | 喹啉衍生物, 点击化学
Luo Zhiqiong · … · Sun Baofei · · 2026/02/03
该研究由神经科学贵州医科大学孙宝飞团队完成 Journal of medicinal chemistry
原文链接:https://doi.org/10.1021/acs.jmedchem.5c03477
6. Niacin Modulates Immune Responses in a Phase I Dose-Escalation Clinical Trial of Newly Diagnosed Glioblastoma.
烟酸调节胶质母细胞瘤患者全身免疫。胶质母细胞瘤具有强烈的免疫抑制微环境,常规疗法和免疫治疗效果有限。本研究通过I期临床试验发现,烟酸能增加循环记忆T细胞和NK细胞频率,并调节促炎细胞因子水平。该研究为烟酸作为胶质母细胞瘤辅助治疗手段提供了初步证据,支持进一步临床评估。
胶质母细胞瘤 | 全身免疫调节, 促炎细胞因子 | I期临床试验
Poon Candice C · … · Yong V Wee · · 2026/02/03
该研究由神经科学卡尔加里大学V Wee Yong团队完成 Neurology(R) neuroimmunology & neuroinflammation
原文链接:https://doi.org/10.1212/NXI.0000000000200530
7. Childhood brain tumors instruct cranial hematopoiesis and immunotolerance.
儿童脑肿瘤诱导颅骨造血产生免疫耐受。脑肿瘤如何逃避免疫监视并重塑全身免疫系统是神经肿瘤学的重要课题。研究发现脑肿瘤通过颅骨骨髓中的抗原呈递细胞诱导调节性T细胞,形成免疫耐受环境。阻断相关细胞因子可恢复造血并诱导肿瘤退缩,为侵袭性脑肿瘤提供了新疗法。
儿童脑肿瘤 | 颅骨骨髓造血, 免疫耐受 | 基因工程小鼠模型
Cooper Elizabeth · … · Gilbertson Richard J · Nature genetics · 2026/02/03
⭐ 该研究由肿瘤学剑桥大学Richard J Gilbertson团队完成
原文链接:https://doi.org/10.1038/s41588-025-02499-2
罕见癌种
1. Phase II Trial of Cabozantinib in Combination with Nivolumab for Advanced Extrapancreatic Neuroendocrine Tumors.
卡博替尼联合纳武利尤单抗在晚期胰腺外神经内分泌肿瘤中疗效有限。卡博替尼被认为能改善肿瘤微环境,增强免疫治疗效果,但在epNET中的表现仍需验证。二期临床试验结果显示,尽管该组合安全性可控,但客观缓解率未达预期目标。该研究提示对于epNET患者,仍需探索更有效的免疫增强策略。
胰腺外神经内分泌肿瘤(epNET) | 多激酶抑制, PD-1抑制 | II期临床试验, 蛋白质组学分析
Perez Kimberly J · … · Chan Jennifer A · · 2026/02/04
该研究由肿瘤学丹娜-法伯癌症研究所Chan Jennifer A团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-2337
2. A Computational Pathology Model to Predict Docetaxel Benefit in Localized High-Risk and Metastatic Prostate Cancer.
AI病理模型预测前列腺癌多西他赛获益。临床缺乏预测性生物标志物,导致前列腺癌患者面临不必要的化疗毒性。研究开发并验证了基于AI的病理图像分类器APIC,通过分析肿瘤免疫空间交互和核异质性预测生存获益。该模型为前列腺癌的精准化疗决策提供了独立于临床因素的有效工具。
前列腺癌 | 肿瘤免疫空间交互 | 人工智能, 计算病理学
Medina Sebastian · … · Madabhushi Anant · · 2026/02/04
该研究由生物医学工程佐治亚理工学院Madabhushi Anant团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-3327
3. Automated Imaging as an Adjunct to Serum and Clinical Biomarkers: A New Validated Prediction Tool for Metastatic Castration-Resistant Prostate Cancer.
自动化影像工具提升前列腺癌风险分层。现有的前列腺癌预后模型多基于治疗前参数,缺乏动态影像信息的整合。研究开发了一种结合骨影像定量分析与血清标志物的预测模型,显著改善了转移性去势抵抗性前列腺癌的风险分类。该工具为临床提供了早期的疗效预测手段,有助于优化晚期患者的治疗方案。
转移性去势抵抗性前列腺癌 | 骨转移 | 放射组学, 自动化定量影像
Morris Michael J · … · Heller Glenn · · 2026/02/04
该研究由流行病学与生物统计学纪念斯隆-凯特琳癌症中心Heller Glenn团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-2890
4. FET-Rearranged Myoepithelial Tumors Are Clinically Heterogeneous and Epigenetically Distinct from PLAG1-Rearranged Adnexal and Salivary Gland Myoepithelial Tumors.
FET重排肌上皮肿瘤的表观遗传特征与临床异质性。肌上皮肿瘤在临床病理上具有高度异质性,其分类和命名存在争议。研究通过DNA甲基化谱分析发现,FET重排肿瘤在表观遗传上与PLAG1重排肿瘤完全不同,更接近肉瘤。该发现建议将FET重排的恶性肌上皮肿瘤重新分类为肉瘤,为精准诊断提供了分子依据。
肌上皮肿瘤 | FET重排, DNA甲基化 | 表观遗传谱分析
Michal Michael · … · Dermawan Josephine K · · 2026/02/04
该研究由病理学克利夫兰医学中心Dermawan Josephine K团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-2426
5. Polyarginine Peptide R11-Actin Interaction Induces a Domino Effect on Cytoskeleton Remodeling to Suppress Bladder Cancer Metastasis.
R11多肽通过重塑细胞骨架抑制膀胱癌转移。细胞骨架重塑是肿瘤转移的关键,但由于缺乏特异性靶点,针对肌动蛋白的药物开发面临巨大挑战。研究开发了靶向膀胱癌的R11多肽,其通过干扰肌动蛋白-波形蛋白轴引发骨架紊乱,显著抑制肿瘤细胞运动。该研究为骨架靶向治疗提供了精准材料策略,展示了R11组装体在治疗转移性膀胱癌中的应用前景。
膀胱癌, 肿瘤转移 | 肌动蛋白, 细胞骨架重塑 | 多肽组装, 纳米材料
Liu Zhenghong · … · Zhang Qi · · 2026/01/29
该研究由泌尿外科学浙江省人民医院Zhang Qi团队完成 Research (Washington, D.C.)
原文链接:https://doi.org/10.34133/research.1109
6. Intercellular Horizontal Transfer of TXNDC5 mRNA via Extracellular Vesicles Contributes to Tumor-Associated Macrophage-Mediated Prostate Cancer Metastasis.
M2巨噬细胞通过外泌体mRNA转移促进前列腺癌转移。肿瘤相关巨噬细胞在微环境中促进肿瘤进展,但其与癌细胞间具体的通讯机制仍有待阐明。研究发现M2巨噬细胞分泌的外泌体可将TXNDC5 mRNA水平转移至前列腺癌细胞,诱导其发生间充质样转变。单囊泡分析证实了这种水平转移的发生,且转移后的mRNA能翻译成功能蛋白。该研究揭示了外泌体介导的水平基因转移在肿瘤转移中的新作用,为前列腺癌治疗提供了新思路。
前列腺癌, 肿瘤转移 | TXNDC5, 外泌体, M2巨噬细胞 | 外泌体分析, 水平基因转移
Hu Cong · … · Xue Wei · · 2026/02/03
该研究由泌尿外科学上海交通大学医学院附属仁济医院Xue Wei团队完成 Advanced science (Weinheim, Baden-Wurttemberg, Germany)
原文链接:https://doi.org/10.1002/advs.202511052
7. A targetable developmental program co-regulates angiogenesis and immune evasion in melanoma.
HOXD13协同调控黑色素瘤血管生成与免疫逃逸。黑色素瘤需通过表观遗传和微环境重塑来逃避免疫检测,但血管生成与免疫排斥的协同机制尚不明确。研究发现转录因子HOXD13通过激活VEGFA和CD73等基因,在促进血管生成的同时抑制T细胞浸润。通过3D染色质分析证实,HOXD13编排了增强子-启动子接触以重塑脉管系统并提升免疫抑制。该研究揭示了HOXD13作为促血管生成和免疫抑制的双重轴心,为黑色素瘤的联合治疗提供了理论依据。
黑色素瘤, 免疫逃逸 | HOXD13, VEGFA, CD73 | 3D染色质分析, 转录组学
Berico Pietro · … · Hernando Eva · · 2026/02/03
该研究由肿瘤学纽约大学朗格尼医学中心Hernando Eva团队完成 Cancer discovery
原文链接:https://doi.org/10.1158/2159-8290.CD-24-1853
8. Notch1 O-GlcNAcylation drives tumor stemness and mechanoadaptation to a stiff microenvironment and promotes chordoma recurrence.
Notch1糖基化驱动脊索瘤机械适应与复发。脊索瘤术后复发率高,且复发组织的微环境硬度显著增加,其背后的机械感知机制不明。研究发现硬微环境通过EPHA2/LYN通路触发Notch1的O-GlcNAc修饰,进而增强肿瘤干性。该研究揭示了肿瘤细胞适应硬环境的分子模型,并指出MIR31缺失可作为复发监测的生物标志物。
脊索瘤复发 | Notch1 O-GlcNAcylation, 机械适应 | 机械力学模拟
Lian Chengjie · … · Liu Lei · · 2026/02/03
该研究由肿瘤学重庆医科大学刘雷团队完成 The Journal of clinical investigation
原文链接:https://doi.org/10.1172/JCI194378
9. Targeting RRBP1 reverses immune evasion and enhances immunotherapy efficacy via the CXCL10-CXCR3 axis in bladder cancer.
靶向RRBP1可逆转膀胱癌免疫逃逸。炎症与免疫系统的交互作用在肿瘤进展中至关重要,但关键调控基因仍待挖掘。研究通过多组学筛选鉴定出RRBP1为关键调控因子,其缺失可激活CXCL10-CXCR3轴并重塑免疫微环境。抑制RRBP1能显著增强抗PD-L1疗效,为膀胱癌免疫治疗提供了新靶点。
膀胱癌, 免疫逃逸 | RRBP1, CXCL10-CXCR3轴 | 单细胞测序, CRISPR筛选
Shen Chengquan · … · Niu Haitao · · 2026/02/03
该研究由泌尿外科学青岛大学附属医院牛海涛团队完成 Journal for immunotherapy of cancer
原文链接:https://doi.org/10.1136/jitc-2025-013809
10. Noninvasive prediction of occult pT3a upstaging in localized ccRCC with radiogenomic insights and prognostic relevance.
深度学习框架RENALNet精准预测肾癌病理升级。局限性透明细胞肾细胞癌的隐匿性病理升级会显著影响预后,但术前准确预测极具挑战。研究开发了基于CT影像的3D深度学习模型RENALNet,不仅预测准确率高,且其风险评分与肿瘤侵袭性通路及预后密切相关。该影像基因组学框架为肾癌的精准手术决策和风险分层提供了有力支持。
透明细胞肾细胞癌 (ccRCC) | 影像基因组学, 肿瘤侵袭性 | 3D深度学习, CT影像分析
Li Shichao · … · Li Zhen · · 2026/02/03
该研究由放射医学华中科技大学同济医院李震团队完成 NPJ precision oncology
原文链接:https://doi.org/10.1038/s41698-026-01315-2
11. Personalized ctDNA analysis for detection of residual disease and recurrence in surgically treated HNSCC patients.
个性化ctDNA监测助力头颈癌术后复发早期预警。头颈鳞状细胞癌(HNSCC)预后改善有限,亟需灵敏的方法检测微小残留病灶和早期复发。研究通过纵向监测血浆和唾液ctDNA,发现其能提前临床诊断多达500天识别复发,并揭示了高排泄量与肿瘤侵袭性通路的关联。该研究证实了ctDNA监测在指导HNSCC精准治疗和改善预后中的巨大价值。
头颈鳞状细胞癌 (HNSCC) | 循环肿瘤DNA (ctDNA), 分子残留病灶 (MRD) | 液体活检, 纵向监测
Flach Susanne · … · Baumeister Philipp · · 2026/02/03
该研究由耳鼻喉头颈外科学德国慕尼黑大学Baumeister Philipp团队完成 NPJ precision oncology
原文链接:https://doi.org/10.1038/s41698-026-01309-0
泛癌种
1. Long-term Survival and Molecular Biomarker Evaluation of a Phase II Cetuximab and Nivolumab Clinical Trial in Recurrent/Metastatic Head and Neck Cancer.
西妥昔单抗联合纳武利尤单抗显著提升p16阴性头颈癌患者生存。复发或转移性头颈鳞癌预后极差,亟需有效的联合免疫治疗方案。二期临床试验长期随访显示,该联合疗法在p16阴性患者中表现出更优的生存获益和更高的响应率。该研究为头颈癌的精准免疫治疗提供了分子生物标志物依据。
头颈部鳞状细胞癌(HNSCC) | EGFR通路, PD-1抑制, p16状态 | 临床试验, 液体活检(cfDNA)
Chaudhary Ritu · … · Chung Christine H · · 2026/02/04
该研究由肿瘤学莫菲特癌症中心Chung Christine H团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-2201
2. ERRγ impedes neuroendocrine prostate cancer development.
核受体ERRγ缺失驱动前列腺癌向神经内分泌型转化。神经内分泌前列腺癌(NEPC)是晚期癌症的致死亚型,其转化机制尚不明确。研究发现ERRγ缺失会激活神经内分泌转录程序,而恢复其功能可抑制肿瘤生长。该研究揭示了ERRγ作为NEPC抑制因子的作用,并提出了针对性的联合用药策略。
神经内分泌前列腺癌(NEPC) | ERRγ, 谱系转化, EZH2/RET抑制 | 类器官模型, 异种移植
Li Ting · … · Giguère Vincent · · 2026/02/02
该研究由肿瘤学麦吉尔大学Giguère Vincent团队完成 Genes & development
原文链接:https://doi.org/10.1101/gad.353024.125
3. Intratumoral Three-Cell-Type Clusters Are a Conserved Feature of Endogenous Antitumor Immunity.
瘤内DC:Th:CTL三细胞簇是内源性抗肿瘤免疫的保守特征。瘤内免疫细胞间的空间相互作用对治疗响应至关重要,但其组织规律尚不清晰。通过整合百万级空间单细胞谱,研究发现这种三细胞簇在多种实体瘤中广泛存在且与生存率正相关。该发现强调了空间生态位在介导抗肿瘤免疫中的核心地位。
肿瘤免疫微环境 | 细胞空间相互作用, T细胞克隆扩张 | 空间转录组学, 空间蛋白质组学
Damle Sheela R · … · Pillarisetty Venu G · · 2026/02/03
该研究由外科学华盛顿大学Pillarisetty Venu G团队完成 Cancer immunology research
原文链接:https://doi.org/10.1158/2326-6066.CIR-25-0062
4. Cergutuzumab Amunaleukin in Combination with Atezolizumab in Patients with Carcinoembryonic Antigen-Positive Advanced/Metastatic Solid Tumors.
Cergutuzumab Amunaleukin联合阿替利珠单抗在实体瘤中表现出免疫激活效应。该研究评估了新型免疫细胞因子CA联合PD-L1抑制剂治疗CEA阳性实体瘤的安全性和活性。临床试验显示该联合方案安全性良好,能诱导外周血T细胞和NK细胞增殖,但抗肿瘤活性有限。该研究为免疫细胞因子的临床开发提供了重要的药代与药效学数据。
CEA阳性晚期实体瘤 | IL-2变体, CEA靶向, PD-L1抑制 | Ib期临床试验, 药代动力学(PK)
Melero Ignacio · … · Lassen Ulrik · · 2026/02/04
该研究由肿瘤学哥本哈根大学医院Lassen Ulrik团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-2440
5. Accelerating Drug Development in Hormone-Naïve Prostate Cancer through Multimodal Therapy Strategies: The MetaCURE Phase 2 Randomized Clinical Trial.
多模态治疗策略加速激素敏感性前列腺癌的药物开发。为了快速评估新药疗效,MetaCURE试验建立了一个结合手术、放疗和新型内分泌药物的框架。研究证实了该多模态方案的安全性,并利用病理完全缓解等中间终点实现了疗效的快速读出。该成果为前列腺癌的适应性临床试验设计提供了重要范式。
激素敏感性前列腺癌 | 雄激素受体抑制, 多模态治疗 | 随机临床试验, 中间终点评估
McHugh Deaglan · … · Scher Howard I · · 2026/02/04
该研究由肿瘤学纪念斯隆-凯特琳癌症中心Scher Howard I团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-2679
6. HPV Integration in Head and Neck Cancer: Downstream Splicing Events and Expression Ratios Linked with Poor Outcomes.
HPV整合诱导的剪接事件与头颈癌不良预后密切相关。HPV整合是驱动癌症进展的关键事件,但其与临床结果的直接联系仍有争议。通过大规模转录组分析,研究鉴定了受整合影响的致癌基因网络,并发现特定的HPV-人类融合转录本可作为预后标志物。该研究为HPV相关头颈癌的精准肿瘤学提供了新的分子靶点和生物标志物。
HPV相关头颈鳞癌 | HPV整合, 异常剪接, 致癌基因网络 | 单细胞转录组测序, 荟萃分析
Li Shiting · … · Sartor Maureen A · · 2026/02/04
该研究由生物信息学密歇根大学Sartor Maureen A团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-0645
7. Facts and Hopes in Harnessing Macrophage-Mediated Antibody-Dependent Cellular Phagocytosis for Cancer Immunotherapy.
巨噬细胞介导的ADCP在癌症免疫治疗中的潜力。虽然NK细胞介导的ADCC广为人知,但巨噬细胞介导的抗体依赖性细胞吞噬作用(ADCP)在免疫治疗中的作用日益凸显。本文综述了肿瘤相关巨噬细胞在ADCP中的核心地位,以及FcγR多态性和抗体工程对疗效的影响。该研究强调了开发选择性增强ADCP策略的重要性,为癌症免疫治疗提供了新方向。
癌症免疫治疗 | 抗体依赖性细胞吞噬作用(ADCP), 肿瘤相关巨噬细胞 | 单克隆抗体工程
Armero Maria · … · Galvez-Cancino Felipe · · 2026/02/04
该研究由免疫调节牛津大学Galvez-Cancino Felipe团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-0434
8. Cytokine-Engineered Chimeric Antigen Receptor-T Cell Therapy: How to Balance the Efficacy and Toxicity.
细胞因子工程化CAR-T细胞治疗的疗效与毒性平衡。CAR-T疗法在实体瘤中受限于免疫抑制微环境,引入细胞因子虽能增强疗效但常伴随严重毒副作用。本综述详细探讨了细胞因子增强CAR-T功能的机制,并分析了细胞因子释放综合征等毒性的病理生理特征。文章介绍了先进的细胞因子工程化设计,旨在改善T细胞的浸润与持久性。文章强调了优化细胞因子产生的重要性,旨在最大化患者获益并降低临床应用风险。
实体瘤, 免疫治疗毒性 | CAR-T, 细胞因子, CRS/ICANS | 综述分析, 细胞工程
Zhang Xinru · … · Yuan Wei-En · · 2026/02/03
该研究由药剂学上海交通大学Yuan Wei-En团队完成 Advanced science (Weinheim, Baden-Wurttemberg, Germany)
原文链接:https://doi.org/10.1002/advs.202518547
9. Deep Learning-Powered Scalable Cancer Organ Chip for Cancer Precision Medicine.
深度学习赋能的可扩展癌症器官芯片。现有功能精准肿瘤学平台面临成本高、周期长及微环境模拟不全等挑战。研究开发了全热塑性注塑成型的器官芯片,并结合深度学习模型进行无标记表型分析。该系统为高通量、患者相关的药物测试提供了可靠的精准医疗框架。
癌症精准医疗, 药物敏感性 | 肿瘤微环境模拟 | 器官芯片, 深度学习
Yuan Yu-Chieh · … · Bai Haiqing · · 2026/02/03
该研究由生物工程Xellar Biosystems团队完成 Advanced science (Weinheim, Baden-Wurttemberg, Germany)
原文链接:https://doi.org/10.1002/advs.202516660
10. CD3ɛ Nanobody-Engineered Extracellular Vesicles Driving In Vivo Generation of BiTE-Secreting CAR-Ts for Solid Tumor Therapy With Memory Response and Minimal Immunogenicity.
纳米抗体修饰外泌体助力体内生成CAR-T。体内生成CAR-T细胞可规避体外制备的复杂性,但合成载体存在免疫原性风险。研究利用CD3ε纳米抗体修饰外泌体,精准递送转基因以在体内产生双抗分泌型CAR-T。该平台在增强抗肿瘤记忆响应的同时,显著降低了免疫原性风险。
实体瘤治疗 | 体内CAR-T生成, CD3ε靶向 | 细胞外囊泡, 纳米抗体工程
Huang Shi-Wei · … · Cho Der-Yang · · 2026/02/03
该研究由细胞治疗中国医药大学附设医院Cho Der-Yang团队完成 Advanced science (Weinheim, Baden-Wurttemberg, Germany)
原文链接:https://doi.org/10.1002/advs.202519440
11. Oncolytic Probiotics with Molecular Pili for Solid Tumor Therapy.
研发靶向实体瘤的溶瘤益生菌。细胞和病毒疗法在实体瘤治疗中常受限于免疫抑制和毒性。研究筛选出具有溶瘤活性的乳杆菌,并通过分子菌毛修饰增强其在肿瘤组织中的聚集。该策略在结直肠癌和黑色素瘤模型中展现了显著的抑瘤效果和良好的安全性。
实体瘤治疗 | 钙内流诱导凋亡, 活性氧爆发 | 溶瘤益生菌
Ge Haodong · … · Zheng Minhua · · 2026/02/03
该研究由普通外科学上海交通大学医学院附属瑞金医院郑民华团队完成 Advanced science (Weinheim, Baden-Wurttemberg, Germany)
原文链接:https://doi.org/10.1002/advs.202517989
12. Longitudinal ctDNA monitoring for post-surgical disease surveillance in patients with stage I-IIIB melanoma.
纵向ctDNA监测预测黑色素瘤术后复发。早期检测复发对I-IIIB期黑色素瘤患者的预后评估和治疗决策至关重要。研究通过个性化ctDNA检测发现,术后任何时间点阳性均与复发风险显著增加及生存期缩短相关。该结果证实了ctDNA作为独立预后因子的价值,强调了其在疾病长期监测中的临床意义。
黑色素瘤 | 术后复发监测 | mPCR-NGS, ctDNA检测
Ansstas George · … · Tan Alan · · 2026/02/03
该研究由肿瘤学范德堡大学Tan Alan团队完成 Clinical cancer research : an official journal of the American Association for Cancer Research
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-3643
13. Repression of PRMT activities sensitize human homologous recombination-proficient ovarian and breast cancer cells to PARP inhibitor treatment.
抑制PRMT增强HR正常肿瘤对PARP抑制剂敏感性。临床上亟需寻找能使同源重组修复正常的肿瘤对PARP抑制剂产生反应的增敏策略。通过药物筛选发现,抑制PRMT1/4/5可下调DNA修复基因并诱导BRCAness状态,从而协同增强DNA损伤。该研究为卵巢癌和乳腺癌的联合用药提供了强有力的临床开发依据。
卵巢癌, 乳腺癌 | PRMT抑制, DNA损伤修复 | 药物筛选, 表观遗传调节
Zhang Youyou · … · Hu Xiaowen · · 2026/02/03
该研究由肿瘤学宾夕法尼亚大学Hu Xiaowen团队完成 eLife
原文链接:https://doi.org/10.7554/eLife.99225
14. Overexpression of the ERG oncogene in prostate cancer identifies candidates for PARP inhibitor-based radiosensitization.
ERG癌基因表达介导前列腺癌放射增敏。前列腺癌放疗疗效受限于肿瘤的DNA修复能力,亟需开发肿瘤特异性的增敏策略。研究发现ERG阳性肿瘤依赖PARP1介导的末端连接修复,使用奥拉帕利可显著增强其放疗敏感性。该发现为ERG融合基因阳性的前列腺癌患者提供了精准放疗联合靶向治疗的新方案。
前列腺癌 | ERG癌基因, PARP1-EJ修复 | 放射增敏, 类器官模型
Köcher Sabrina · … · Mansour Wael Y · · 2026/02/03
该研究由放射肿瘤学汉堡-埃彭多夫大学医学中心Mansour Wael Y团队完成 The Journal of clinical investigation
原文链接:https://doi.org/10.1172/JCI194949
15. Personalized Cancer Vaccine Shows Durable Benefit at 5 Years.
个性化癌症疫苗展现五年持久疗效。KEYNOTE-942研究的五年随访数据证实,mRNA新抗原疫苗联合免疫治疗可显著降低复发风险。数据显示该联合方案将黑色素瘤患者的死亡或复发风险降低了约49%,且疗效具有持久性。这一结果增强了对个性化疫苗策略的信心,为即将开展的三期临床试验奠定了基础。
黑色素瘤 | 新抗原疫苗, 免疫持久性 | mRNA疫苗, 联合疗法
Cancer discovery · 2026/02/02
原文链接:https://doi.org/10.1158/2159-8290.CD-NW2026-0009
16. An interpretable molecular framework for predicting cancer driver missense mutations.
可解释分子框架MutaPheno助力鉴定癌症驱动错义突变。准确预测错义突变的功能影响是癌症研究中的重大挑战。研究基于12万个变体开发了集成34个分子特征的随机森林模型。MutaPheno在预测驱动突变方面表现优异,为发现新驱动因子和开发靶向疗法提供了有力工具。
癌症驱动突变 | 错义突变 | 随机森林, 生物信息学
Yang Yan · … · Li Minghui · · 2026/02/03
该研究由生物信息学苏州大学李明晖团队完成 Proceedings of the National Academy of Sciences of the United States of America
原文链接:https://doi.org/10.1073/pnas.2524289123
17. Exploring the Vasculitis-Tumors Link: Epidemiological Patterns, Mechanistic Insights, and Clinical Implications.
综述系统阐述血管炎与恶性肿瘤之间的流行病学及机制联系。血管炎与肿瘤的关联具有重要的临床意义,但其致病机制较为复杂。本文总结了不同亚型血管炎的肿瘤风险、临床特征及潜在致病路径。研究结果为风湿科和肿瘤科医生在鉴别诊断及风险管理方面提供了实用指导。
血管炎 | 肿瘤相关性 | 系统综述
Shi Xiaofei · … · Chu Cong-Qiu · · 2026/02/03
该研究由风湿免疫学俄勒冈健康与科学大学Chu Cong-Qiu团队完成 Advanced science (Weinheim, Baden-Wurttemberg, Germany)
原文链接:https://doi.org/10.1002/advs.202521056
18. Pharmacological activation of cGAS-STING pathway to reverse cancer drug resistance.
药理激活cGAS-STING通路以逆转肿瘤耐药。肿瘤细胞常通过抑制cGAS-STING信号通路来逃避免疫监视并产生耐药。本文综述了肿瘤通过表观遗传和代谢等手段抑制该通路的机制。评价了利用小分子激动剂恢复化疗及免疫治疗敏感性的最新策略,为克服治疗障碍提供参考。
肿瘤耐药 | cGAS-STING通路, 先天免疫 | 小分子激动剂
Wang Yumin · … · Wang Hongquan · · 2026/02/01
该研究由老年医学北京大学航天临床医学院王洪权团队完成 Pharmacology & therapeutics
原文链接:https://doi.org/10.1016/j.pharmthera.2026.108991
19. HB023: A glutamine antagonist prodrug boosting antitumor lmmunity via PD-L1 suppression and mitochondrial membrane remodeling.
谷氨酰胺拮抗剂前药HB023通过抑制PD-L1增强抗肿瘤免疫。谷氨酰胺剥夺虽能抑制肿瘤代谢,但常诱导PD-L1表达导致免疫逃逸。研究开发了偶联JQ1的前药HB023,能同时引发代谢压力并下调PD-L1。该双重功能策略协同激活了先天与适应性免疫,显著提升了免疫治疗效果。
肿瘤免疫逃逸 | 谷氨酰胺代谢, PD-L1抑制 | 前药设计, 代谢-免疫联合治疗
Zhuang Junyan · … · Wei Gaofei · · 2026/02/01
该研究由细胞代谢西北工业大学魏高飞团队完成 Journal of advanced research
原文链接:https://doi.org/10.1016/j.jare.2026.01.077
20. Design and Synthesis of KRASG12C Inhibitors for Antitumor Evaluation Harboring Combination Therapy with Nrf2, PARP-7, and Pan-USP Inhibitors to Alleviate Drug Resistance Synergistically.
新型KRASG12C抑制剂联合用药缓解耐药。KRASG12C突变在多种癌症中高发,但单一抑制剂易产生耐药性。研究合成了一系列新型抑制剂,其中化合物7q在体内外均表现出优异的抗肿瘤活性。7q与Nrf2或USP抑制剂联用可产生协同效应,为克服KRAS突变肿瘤耐药提供了新方案。
KRASG12C突变肿瘤, 药物耐药 | KRASG12C, 联合用药 | 药物合成, 异种移植模型
Tian Liangliang · … · Chen Lixue · · 2026/02/03
该研究由药理学大连医科大学陈立学团队完成 Journal of medicinal chemistry
原文链接:https://doi.org/10.1021/acs.jmedchem.5c02703
21. Immune checkpoint inhibitor-related pneumonitis: current advances and the putative role of mesenchymal stem cell therapy.
间充质干细胞有望治疗免疫检查点抑制剂相关肺炎。免疫检查点抑制剂常引发严重的肺部不良反应,现有治疗手段存在局限。本文综述了相关肺炎的进展,并提出利用间充质干细胞的免疫调节特性进行治疗。该研究强调了MSC疗法在改善并发症管理中的潜力,并指出了未来临床研究的方向。
免疫检查点抑制剂相关肺炎, 免疫相关不良反应 | 免疫调节, 组织修复 | 间充质干细胞疗法
Li Zekang · … · Wu Chen · · 2026/02/03
该研究由肿瘤学苏州大学附属第三医院吴晨团队完成 Cell death & disease
原文链接:https://doi.org/10.1038/s41419-026-08440-7
22. Expanding the DNA damaging potential of artificial metallo-nucleases with click chemistry.
利用点击化学扩展人工金属核酸酶的DNA损伤潜力。设计具有高效DNA损伤能力的人工金属核酸酶(AMNs)是抗癌药物研发的重要方向。研究利用点击化学生成了一系列三点击(Tri-Click)配体,并筛选出具有高活性的铜配合物Cu3-TC-Py。该配合物在单分子成像和细胞实验中展现出极强的DNA结合与损伤能力,能有效诱导人类及细菌细胞内的DNA断裂。研究确立了Cu3-TC-Py作为一种新型抗癌先导化合物的潜力,为开发基于DNA损伤的化疗药物提供了新策略。
癌症治疗 | DNA损伤, 人工金属核酸酶 | 点击化学(CuAAC), 单分子成像
Gibney Alex · … · Kellett Andrew · · 2026/02/03
该研究由药物化学都柏林城市大学Andrew Kellett团队完成 Nature communications
原文链接:https://doi.org/10.1038/s41467-026-68911-5
23. Human-AI teaming to improve accuracy and efficiency of eligibility criteria prescreening for oncology trials: a randomized evaluation trial using retrospective electronic health records.
人机协作提升肿瘤临床试验入组预筛查的准确性与效率。临床试验预筛查过程繁琐且易出错,是限制患者入组的关键瓶颈。研究开展了一项随机对照试验,比较了纯人工筛查与AI语言模型辅助筛查在355名癌症患者中的表现。结果显示,人机协作组在准确性上显著优于纯人工组(76.5% vs 71.1%),尤其在生物标志物和分期判定方面提升明显,但筛查时间未见缩短。该研究证明了AI在辅助临床决策中的潜力,为优化肿瘤临床试验流程提供了实证支持。
肿瘤临床试验入组 | 临床决策支持, 自动化预筛查 | 神经符号AI, 随机非劣效性试验
Parikh Ravi B · … · Emanuel Ezekiel J · · 2026/02/03
该研究由医学伦理与健康政策宾夕法尼亚大学Ravi B Parikh团队完成 Nature communications
原文链接:https://doi.org/10.1038/s41467-026-68873-8
24. Generalisation of automatic tumour segmentation in histopathological whole-slide images across multiple cancer types.
跨癌种自动肿瘤分割通用模型的开发与验证。深度学习有望辅助病理学家进行肿瘤分割,但模型的跨癌种泛化能力仍需验证。研究利用超2万张全视野数字化切片开发了通用模型,在多种外部队列中验证其Dice系数均超过80%。该研究证明了单一通用模型在不同癌种和样本制备条件下的临床应用潜力。
肿瘤分割, 数字化病理 | 深度学习泛化性 | 全视野数字化切片 (WSI), 卷积神经网络
Skrede Ole-Johan · … · Kleppe Andreas · · 2026/02/04
该研究由肿瘤信息学挪威奥斯陆大学医院Kleppe Andreas团队完成 NPJ precision oncology
原文链接:https://doi.org/10.1038/s41698-026-01311-6
25. The second mitochondrial activator of caspases (SMAC) regulates growth, inflammation and mitochondrial integrity in cancer cells.
SMAC蛋白在非凋亡癌细胞中调节生长与炎症。SMAC传统上被认为是促凋亡蛋白,但其在未发生细胞死亡的癌细胞中的功能尚不清楚。研究发现SMAC能自发释放至胞质,通过激活STING信号调节干扰素反应,并影响线粒体形态及肿瘤的迁移和转移能力。该研究揭示了SMAC在癌症生物学中的多重非凋亡功能,提示其可作为预后标志物。
肿瘤转移, 炎症反应 | SMAC, STING信号通路 | 基因敲除, 异种移植模型
Amer Tarek N · … · Häcker Georg · · 2026/02/03
该研究由医学微生物学德国弗莱堡大学Häcker Georg团队完成 Cell death and differentiation
原文链接:https://doi.org/10.1038/s41418-026-01667-z
26. Brain-cancer interactions outside the CNS.
脑-外周肿瘤环路调控癌症进展。神经系统如何调节外周肿瘤的发生与演变是当前生物医学领域的研究热点。本综述总结了脑与外周肿瘤间的双向神经通讯网络,揭示了其对肿瘤微环境及全身代谢的影响。深入理解这一机制为开发基于神经调节的癌症整合治疗策略提供了新方向。
脑-外周肿瘤相互作用 | 神经-免疫-代谢轴 | 逆行追踪技术, 神经调节治疗
Li Weihan · … · Xue Junli · · 2026/02/03
该研究由肿瘤学同济大学薛俊丽团队完成 Oncogene
原文链接:https://doi.org/10.1038/s41388-026-03684-1
27. Global and regional cancer burden attributable to modifiable risk factors to inform prevention.
全球近四成癌症病例可归因于可控风险。明确可预防风险因素对癌症负担的影响是制定全球公共卫生政策的关键。研究估算了2022年30种风险因素导致的癌症负担,发现吸烟、感染和饮酒是主要诱因。强化对可控暴露因素的干预仍是全球癌症预防工作的核心任务。
癌症预防 | 可控风险因素 | 流行病学负担评估
Fink Hanna · … · Soerjomataram Isabelle · Nature medicine · 2026/02/03
⭐ 该研究由癌症流行病学国际癌症研究机构(IARC)Isabelle Soerjomataram团队完成
原文链接:https://doi.org/10.1038/s41591-026-04219-7
28. Targeting extrachromosomal DNA in human cancers.
靶向染色体外DNA助力癌症精准治疗。染色体外DNA(ecDNA)介导的癌基因扩增是导致肿瘤进化和耐药的关键机制。本综述系统阐述了ecDNA的生物学特性及其在临床诊断和治疗中的潜在脆弱性。提出未来应通过化学蛋白质组学等手段开发针对ecDNA的创新疗法。
肿瘤耐药与进化 | 染色体外DNA(ecDNA) | 化学蛋白质组学, 临床诊断
Wong Ivy Tsz-Lo · … · Mischel Paul S · Nature reviews. Drug discovery · 2026/02/03
⭐ 该研究由病理学斯坦福大学Paul S Mischel团队完成
原文链接:https://doi.org/10.1038/s41573-025-01369-0
29. Fusobacterium nucleatum manipulates host autophagy to promote its intracellular survival and treatment resistance in nasopharyngeal carcinoma.
具核梭杆菌通过抑制自噬促进鼻咽癌耐药。胞内微生物在肿瘤进展中的作用日益受到关注,但其生存及促癌机制尚不明确。研究发现具核梭杆菌通过降解RAB7A抑制宿主自噬流,从而实现胞内生存并诱导治疗耐药。该成果为鼻咽癌提供了新的微生物预后指标及潜在的治疗干预靶点。
鼻咽癌耐药 | 具核梭杆菌, 自噬抑制 | 转录组学分析
Wang Jing-Yun · … · Liu Na · · 2026/02/04
该研究由肿瘤学中山大学肿瘤防治中心刘娜团队完成 Molecular cancer
原文链接:https://doi.org/10.1186/s12943-026-02581-1
30. Stable Cyclometalated Gold(III) Complex Engaging Isoquinoline Derivative and Disulfur Ligand Elicits Necroptosis-Dependent Immunogenic Cell Death.
金(III)配合物通过诱导坏死性凋亡触发免疫原性细胞死亡。金属配合物在肿瘤化疗免疫治疗中具有潜力,但亟需开发能通过非凋亡途径诱导免疫原性细胞死亡(ICD)的新型诱导剂。研究设计了一种稳定的环金属化金(III)配合物Au-1,其通过抑制硫氧还蛋白还原酶引发ROS介导的坏死性凋亡,进而激活抗肿瘤免疫。该研究为设计新型金属类ICD诱导剂及联合免疫检查点阻断治疗提供了新策略。
肿瘤免疫治疗 | 坏死性凋亡, 免疫原性细胞死亡(ICD) | 金(III)配合物
Zhang Wei · … · Liang Hong · · 2026/02/03
该研究由药物资源化学广西师范大学Liang Hong团队完成 Journal of medicinal chemistry
原文链接:https://doi.org/10.1021/acs.jmedchem.5c03654
31. AACR Pediatric Cancer Progress Report 2025.
2025年AACR儿童癌症进展报告发布。美国癌症研究协会(AACR)发布首份儿童癌症报告,总结了过去十年在分子靶向治疗和基因组学领域的重大突破。报告指出,尽管诊疗手段不断进步,罕见及侵袭性儿童癌症仍面临有效疗法匮乏和医疗资源分配不均的挑战。该报告强调了加强联邦投资和国际合作的紧迫性,旨在加速攻克儿童癌症。
儿童癌症 | 分子靶向治疗 | 进展报告
Primm Kristin M · … · Sengupta Rajarshi · Clinical cancer research : an official journal of the American Association for Cancer Research · 2026/02/04
原文链接:https://doi.org/10.1158/1078-0432.CCR-25-4722
32. TISSUE-SPECIFIC SPATIAL REGULATION OF INNATE IMMUNE CHECKPOINTS IN CANCER.
癌症中先天免疫检查点的组织特异性空间调节。自然杀伤(NK)细胞和先天淋巴细胞(ILC)是组织稳态的早期传感器,其功能受局部微环境和免疫检查点的严密调控。本文探讨了组织特异性信号如何塑造癌症中NK/ILC的细胞状态和效应程序,以及空间重新分布对免疫反应的影响。针对先天免疫检查点的靶向治疗有望为实体瘤的免疫治疗提供新的策略和补充。
实体瘤免疫逃逸 | 先天免疫检查点, NK/ILC细胞 | 空间免疫调节分析
Stojanovic Ana · … · Cerwenka Adelheid · · 2026/02/04
该研究由免疫学海德堡大学Cerwenka Adelheid团队完成 Cancer discovery
原文链接:https://doi.org/10.1158/2159-8290.CD-25-2112
声明:本文使用了AI进行翻译和总结
信使RNA疫苗临床研究免疫疗法
2026-02-03
下面内容由ReviewMaker生成,想使用的可以私聊:
414391587@qq.com
科研人的“外挂”来了!ReviewMaker 3.0:从文献调研到万字综述,我们的AI工具帮您搞定一个基因的全面总结(以铁死亡核心分子GPX4为例)Abstract
KRAS是人类癌症中最常见的突变癌基因之一,尤其在胰腺癌、肺癌和结直肠癌中高发。在过去四十年中,由于其蛋白表面光滑、与GTP亲和力高,KRAS一直被认为是“不可成药”的靶点。然而,随着对KRAS生物学功能、调控机制以及其与肿瘤微环境相互作用的深入理解,特别是近期针对KRAS G12C突变体的特异性抑制剂的成功开发,靶向KRAS治疗进入了一个新纪元。本综述旨在系统性地梳理KRAS蛋白的分子生物学特征,包括其复杂的翻译后修饰、膜定位、二聚化以及基因表达调控;深入探讨KRAS在不同肿瘤类型中的致癌作用,特别是野生型等位基因和不同突变亚型所扮演的独特角色;全面解析KRAS如何通过重塑细胞信号网络、代谢途径以及肿瘤微环境来驱动肿瘤的发生发展和免疫逃逸。此外,本综述将重点介绍当前靶向KRAS的各类前沿治疗策略,涵盖直接抑制剂、蛋白降解剂、下游通路抑制剂、合成致死策略及免疫疗法。最后,我们将剖析KRAS靶向治疗中出现的内在性和获得性耐药机制,并展望未来通过联合治疗和开发泛KRAS抑制剂来克服这些挑战的前景。1. 引言 (Introduction)
RAS (Rat Sarcoma) 基因家族是首批被发现的人类癌基因,其中,Kirsten大鼠肉瘤病毒癌基因同源物 (Kirsten rat sarcoma viral oncogene homolog, KRAS) 自1982年被确认为首个在人类癌症中异常激活的癌基因以来,便一直处于癌症研究的核心地位 [1]。在过去的四十年里,对KRAS的研究极大地加深了我们对肿瘤发生和发展的理解。KRAS基因的激活突变是人类癌症中最常见的致癌驱动事件之一,在多种恶性实体瘤中广泛存在。尤其是在一些最致命的癌症中,KRAS突变频率极高,例如,接近95%的胰腺导管腺癌 (pancreatic ductal adenocarcinoma, PDAC)、约40%的结直肠癌 (colorectal cancer, CRC) 以及约33%的肺腺癌 (lung adenocarcinoma, LUAD) 均由KRAS突变驱动 [1, 2, 3]。
由于其蛋白表面光滑、缺乏明显的药物结合口袋,以及对GTP的高亲和力,KRAS在长达四十年的时间里被认为是“不可成药”(undruggable) 的靶点,这使得针对KRAS突变肿瘤的靶向治疗开发举步维艰 [4, 1]。然而,这一困境在近年被彻底打破。2013年,科学家们在KRAS G12C突变体蛋白的GDP结合状态下发现了一个可供药物结合的变构口袋(Switch-II口袋),这一里程碑式的发现为直接靶向KRAS突变体打开了大门 [5]。基于此,一系列共价抑制剂被成功开发出来,它们能够特异性地与KRAS G12C突变体中的半胱氨酸残基共价结合,将其锁定在非活性的GDP结合状态。其中,Sotorasib (AMG510) 和 Adagrasib (MRTX849) 在临床试验中取得了令人鼓舞的结果,Sotorasib更是成为首个获得美国食品药品监督管理局 (FDA) 批准上市的KRAS G12C抑制剂,标志着KRAS靶点从“不可成药”到“可成药”的历史性转变 [5, 4]。
这一突破不仅为携带KRAS G12C突变的癌症患者带来了新的希望,也极大地激发了针对其他KRAS突变亚型(如G12D、G12V等)以及泛KRAS抑制剂的研发热情。本文旨在全面综述KRAS的研究进展,将从KRAS的分子生物学特性、致癌机制、复杂的信号调控网络、多样化的治疗策略(包括直接和间接抑制剂)、药物耐药机制的探索以及未来的临床研究展望等方面进行深入探讨,以期为理解KRAS在癌症中的作用及开发更有效的治疗策略提供参考。2. KRAS蛋白的分子生物学与调控 (Molecular Biology and Regulation of the KRAS Protein)
KRAS蛋白作为一种小GTP酶,在细胞信号转导中扮演着分子开关的关键角色,其功能受到从基因转录到蛋白质降解等多个层面的精密调控。对这一复杂调控网络的深入理解,为开发靶向KRAS的治疗策略提供了重要理论基础。
2.1 基因转录、剪接与翻译调控
KRAS基因表达的调控机制总结名称内容/功能引用(PMID)G-四链体 (G-quadruplex, G4)位于KRAS启动子区域,作为转录调控因子影响KRAS表达。[6, 7]小檗碱 (berberine) / 黄连素 (coptisine)结合并稳定KRAS启动子的G4结构,从而降低KRAS的mRNA水平。[7]APE1作为G4结合蛋白,促进KRAS启动子G4结构折叠,调控KRAS表达。[8]可变剪接 (Alternative splicing)产生KRAS4A和KRAS4B两种蛋白异构体,它们在致癌过程中可能发挥不同作用。[9]miR-96直接靶向并抑制KRAS的mRNA表达,在胰腺癌中发挥抑癌功能。[10]稀有密码子偏好性 (Rare codon bias)限制KRAS蛋白的翻译效率和表达水平,对肿瘤发生有复杂影响。[11, 12]
KRAS蛋白的表达首先在转录水平受到调控。其启动子区域的富含GC的核酸酶超敏元件(NHPPE)能够形成G-四链体(G-quadruplex, G4)结构,这种特殊的二级结构作为转录调控因子,影响KRAS的表达 [6, 7]。研究发现,小分子配体如小檗碱(berberine)和黄连素(coptisine)能结合并稳定这种G4结构,从而显著降低癌细胞中KRAS的mRNA水平 [7]。此外,DNA修复酶APE1也被发现是一种G4结合蛋白,能够促进KRAS启动子G4结构的折叠,并通过影响转录因子MAZ的结合来调控KRAS的表达 [8]。
在转录后水平,KRAS基因通过可变剪接产生两种主要的蛋白异构体:KRAS4A和KRAS4B。这两种异构体在C末端高变区(HVR)存在差异,导致它们经历不同的翻译后修饰,并可能在致癌过程中发挥不同作用。例如,KRAS4A被证明在肺癌发生中尤为重要 [9]。此外,microRNA(miRNA)也参与KRAS的表达调控,例如miR-96可直接靶向KRAS的mRNA,抑制其表达,从而在胰腺癌中发挥抑癌基因的功能 [10]。
在翻译水平,KRAS基因的密码子偏好性也对其蛋白表达量有重要影响。KRAS基因富含稀有密码子,这天然地限制了其蛋白的翻译效率和表达水平。这种低表达特性在肿瘤发生中起着复杂的作用,一方面,过高水平的野生型KRAS蛋白反而具有肿瘤抑制活性;另一方面,肿瘤细胞在获得耐药性的过程中,可能需要克服这种密码子偏好性限制,通过增加KRAS拷贝数等方式来提高突变KRAS蛋白的表达量 [11, 12]。2.2 翻译后修饰与亚细胞定位
表1. 调控KRAS亚细胞定位的关键分子及其功能名称内容/功能引用(PMID)法尼基化修饰KRAS锚定到细胞膜的第一步。[13]PDEδ识别并捕获错误定位到内膜系统的KRAS。[14, 15]Arl2辅助PDEδ将KRAS释放到核周膜区域。[14, 15]SNARE蛋白 (SNAP23, SNAP29, VAMP3)介导囊泡运输,将KRAS重新转运并富集到质膜。[14, 16]SNORD50A/B负向调控KRAS的质膜定位,抑制其与SNARE蛋白的邻近性。[16]鞘糖脂 (Glycosphingolipids)维持KRAS在质膜上的定位和纳米级空间组织。[17]
KRAS蛋白的功能高度依赖于其在细胞质膜上的正确定位,而这一过程由一系列复杂的翻译后修饰(Post-Translational Modifications, PTMs)和蛋白转运机制共同调控。新合成的KRAS蛋白首先在胞质中经历法尼基化修饰,这是其锚定到细胞膜上的第一步 [13]。然而,仅靠法尼基化不足以实现其向质膜的特异性富集。
KRAS蛋白的亚细胞定位是一个动态循环过程。错误定位到内质网等内膜系统的KRAS可被胞质中的可溶性蛋白PDEδ识别并“捕获”,随后在Arl2蛋白的辅助下被释放到核周膜区域 [14, 15]。之后,KRAS通过静电相互作用被捕获在再循环内体(recycling endosome),最终通过SNARE蛋白(如SNAP23, SNAP29, VAMP3)介导的囊泡运输途径被重新转运并富集到质膜上 [14, 16]。这一过程受到小GTP酶超家族成员SNORD50A/B的负向调控,它们通过抑制KRAS与SNARE蛋白的邻近性来拮抗其质膜定位 [16]。此外,细胞代谢状态也深刻影响KRAS的定位,例如糖酵解通路衍生的特定鞘糖脂(glycosphingolipids)对于维持KRAS在质膜上的定位和纳米级空间组织至关重要 [17]。2.3 蛋白稳定性与降解
调控KRAS蛋白稳定性的关键分子及其功能名称内容/功能引用(PMID)JOSD2去泛素化酶,直接与KRAS突变体作用并使其稳定,形成正反馈回路加速肿瘤生长。[18]NDRG3 / USP9X支架蛋白NDRG3促进去泛素化酶USP9X与KRAS的相互作用,从而上调KRAS蛋白表达。[19]AIMP2-DX2通过阻断E3连接酶Smurf2与KRAS的接触,阻止其被泛素化降解,从而稳定KRAS。[13]LZTR1介导经典的泛素依赖性降解通路。其功能受损会导致野生型KRAS水平升高。[20]REGγ (NRF2/REGγ通路)KRAS突变通过NRF2上调REGγ,增强ATP和泛素非依赖性的蛋白酶体降解通路。[21]
KRAS蛋白的丰度受到泛素-蛋白酶体系统的严密调控,其稳定性是决定其致癌活性的关键因素。多种去泛素化酶(DUBs)被发现能够移除KRAS上的泛素链,从而保护其免于降解,延长其半衰期。例如,在结直肠癌中,去泛素化酶JOSD2可以直接与多种KRAS突变体相互作用并使其稳定。有趣的是,KRAS突变体又能反过来抑制JOSD2的E3泛素连接酶CHIP的活性,形成一个JOSD2/KRAS正反馈回路,从而显著加速肿瘤生长 [18]。另一项研究发现,支架蛋白NDRG3能够促进去泛素化酶USP9X与KRAS的特异性相互作用,从而上调KRAS蛋白的表达 [19]。
相反,一些蛋白则通过促进KRAS的泛素化降解来发挥肿瘤抑制作用。例如,肿瘤抑制因子AIMP2的剪接变体AIMP2-DX2,在KRAS法尼基化之前与其结合,通过竞争性阻断E3泛素连接酶Smurf2与KRAS的接触,阻止其被泛素化介导的降解,从而增强KRAS驱动的肿瘤发生 [13]。此外,LZTR1介导的降解通路也至关重要,该通路的受损会导致野生型KRAS蛋白水平升高,从而驱动对RAS抑制剂的内源性耐药 [20]。除了经典的泛素依赖性降解,研究还发现KRAS突变可通过NRF2上调REGγ,增强ATP和泛素非依赖性的蛋白酶体降解通路,这为泛KRAS抑制剂的开发提供了新思路 [21]。2.4 磷酸化、二聚化与其他相互作用调控
调控KRAS活性的关键分子与机制总结名称内容/功能引用(PMID)PKC / Ser-181磷酸化蛋白激酶C(PKC)磷酸化KRAS的Ser-181位点,此修饰对突变KRAS的致癌功能至关重要。[22]HNRNPA2B1与KRAS G12V相互作用,调节其磷酸化水平,影响胰腺癌细胞的KRAS依赖性。[23]Src激酶磷酸化KRAS的Tyr32和Tyr64位点,导致GTPase循环停滞并削弱与下游效应子的结合。[24]SHP2对KRAS进行去磷酸化,恢复其正常的GTPase循环动态。[24]野生型KRAS等位基因通过形成异源二聚体抑制突变KRAS的致癌活性;其存在是MEK抑制剂耐药性的原因之一。[25, 26, 27]野生型KRAS杂合性丢失(LOH)增强突变KRAS信号(尤其是MAPK通路),促进肿瘤起始,并使肿瘤对MEK抑制剂敏感。[26, 27, 25]SHANK3支架蛋白,与激活的KRAS结合,通过与RAF竞争来限制MAPK/ERK信号,抑制它可诱导“信号超载”凋亡。[28]
KRAS蛋白的活性还受到磷酸化、二聚化以及与其他蛋白相互作用的精细调控。
磷酸化修饰:磷酸化是调节KRAS活性的重要机制。位于其C末端高变区的丝氨酸181(Ser-181)位点的磷酸化被证明对突变KRAS的致癌功能至关重要。蛋白激酶C(PKC)可催化该位点的磷酸化,磷酸化模拟突变体S181D展现出更强的成瘤能力,而不可磷酸化突变体S181A则无法诱导肿瘤形成 [22]。此外,异质性核糖核蛋白HNRNPA2B1能与KRAS G12V相互作用,并调节其磷酸化水平,进而影响胰腺癌细胞的KRAS依赖性 [23]。酪氨酸磷酸化也扮演着重要角色,Src激酶可磷酸化KRAS的Tyr32和Tyr64位点,导致其构象改变,GTPase循环停滞并削弱与下游效应子的结合;而蛋白酪氨酸磷酸酶SHP2则能对KRAS进行去磷酸化,恢复其正常的GTPase循环动态 [24]。
二聚化与等位基因相互作用:KRAS蛋白可以在质膜上形成同源或异源二聚体,这一过程深刻影响其信号输出。野生型(WT)KRAS等位基因通常对突变KRAS的致癌活性具有抑制作用,这种抑制效应部分是通过形成突变型-野生型异源二聚体来实现的 [25]。当肿瘤细胞发生杂合性丢失(LOH),即野生型KRAS等位基因缺失时,突变KRAS的致癌信号(尤其是MAPK通路)会显著增强,从而促进肿瘤的起始 [26, 27]。有趣的是,野生型KRAS的缺失虽然加速了肿瘤起始,但在某些模型中却可能抑制肿瘤的进展和转移 [27, 26]。这种复杂的双重作用凸显了KRAS等位基因剂量在肿瘤不同阶段扮演的不同角色。同时,野生型KRAS的存在是导致MEK抑制剂耐药性的原因之一,而野生型等位基因的缺失则会使肿瘤细胞对MEK抑制剂变得敏感 [25, 26, 27]。
其他相互作用蛋白:除了经典的上下游分子,许多新发现的相互作用蛋白也参与调控KRAS的功能。例如,支架蛋白SHANK3能与激活状态的KRAS(包括突变体)结合,通过与RAF竞争结合位点来限制下游MAPK/ERK信号的过度激活,将信号维持在一个最佳水平。因此,抑制SHANK3会导致ERK信号“过载”,从而诱导KRAS突变癌细胞的凋亡,这为治疗KRAS突变癌症提供了一种全新的“信号超载”策略 [28]。
综上所述,KRAS蛋白的生命周期受到一个多层次、动态且高度整合的调控网络所控制。从基因表达的源头,到翻译后修饰、蛋白稳定性、亚细胞穿梭,再到与其他分子的复杂相互作用,每一个环节都为KRAS的功能提供了精细的调校机制。这些复杂的调控节点不仅揭示了KRAS致癌活性的分子基础,也为开发新型、多角度的抗癌疗法开辟了广阔的前景。3. KRAS在肿瘤发生发展中的多维角色 (The Multidimensional Role of KRAS in Tumorigenesis and Progression)
KRAS在肿瘤发生发展中的多维角色及相关分子机制总结名称/因素内容/功能引用(PMID)Kras G12D (内源性水平)引起细胞增殖和运动能力增强,但不足以完成恶性转化。[29]KrasG12D (结肠上皮)导致增生性息肉样结构,但不扩增干细胞池,恶性潜能较低。[30]PIK3CA 突变与 KRAS(G12D) 协同作用,显著加速肺肿瘤的发生。[31]AP-1/IL-1α/p62/NF-κB 信号轴在胰腺癌中,KrasG12D通过此信号轴持续激活NF-κB通路。[32]KRAS 抑制/敲除在胶质母细胞瘤和胰腺癌模型中导致肿瘤消退,体现“癌基因成瘾”。[33, 34]PI3K 依赖的 MAPK 信号通路作为KRAS抑制的耐药机制,维持胰腺癌细胞存活。[35]YAP1 信号通路激活后可绕过对KRAS的依赖,恢复肿瘤细胞的生存和增殖。[36, 37]FOSL1 (转录因子)连接KRAS致癌信号与有丝分裂机器(如AURKA),是KRAS驱动癌症的关键脆弱性。[38]KRAS 突变亚型 (G12C, G12V, G12D)在蛋白质行为、信号转导和临床预后方面表现出显著差异。[39]KRAS 拷贝数增加增强糖酵解转换和代谢重编程。[40]野生型 KRAS 等位基因缺失增强致癌信号,促进所有RAS亚型激活,加速白血病发生。[41]代谢重编程 (葡萄糖)KRAS刺激葡萄糖摄取,并将其导入己糖胺生物合成途径(HBP)和戊糖磷酸途径(PPP)。[34]MAPK-MYC-RPIA 信号轴KRAS通过此信号轴上调核苷酸的生物合成。[42]FASN (脂肪酸合酶)被KRAS激活,促进脂肪生成。[43]ELOVL6 (脂肪酸延长酶)调控KRAS-G12V蛋白的稳定性。[44]巨噬细胞吞饮作用 (Macropinocytosis)KRAS突变细胞通过此作用获取铜,并上调ATP7A以避免铜毒性。[45]PCSK9被KRAS上调以调控胆固醇代谢,促进GGPP积累。[46]CD47 ("别吃我"信号)KRAS通过PI3K/STAT3/miR-34a轴上调CD47,抑制巨噬细胞吞噬,促进免疫逃逸。[47]COX2/PGE2KRAS诱导COX2表达,其产物PGE2促进对免疫检查点抑制剂的抵抗。[48]KRAS C118位点氧化一种翻译后修饰,介导KRAS的氧化还原依赖性激活。[49]PIP5K1A (互作蛋白)作为KRAS特异性的互作蛋白,对其功能进行精细调控。[50]PROTAC 降解剂靶向GTP负载的KRAS(on)状态的新型治疗策略。[51]广谱 KRAS 抑制剂新一代靶向KRAS的治疗方法。[52, 53]
KRAS基因的激活突变是多种人类癌症中最常见的驱动因素之一,其在肿瘤的起始、进展和维持中扮演着复杂而多维的角色。深入理解KRAS突变如何从不同层面重塑细胞生物学行为,对于开发有效的治疗策略至关重要。
在肿瘤起始阶段,KRAS突变通常被认为是关键的“第一次打击”,但其本身往往不足以完成恶性转化。例如,在小鼠体细胞中通过基因敲入方式引入内源性水平的Kras G12D突变,虽然能引起细胞增殖和运动能力增强等显著的表型变化,但并不足以使细胞完全转化 [29]。在结肠上皮中,激活KrasG12D突变可导致类似于人类增生性息肉(HPPs)的锯齿状隐窝结构和分化异常,但并不会扩增干细胞池,这解释了为何携带KRAS突变的HPPs恶性潜能较低 [30]。这些发现表明,KRAS突变需要与其他遗传或环境因素协同作用才能驱动肿瘤的全面发展。例如,PIK3CA基因的激活突变能够与KRAS(G12D)协同,显著加速和增强肺肿瘤的发生 [31]。同样,在胰腺癌的发生过程中,KrasG12D需要通过激活AP-1诱导IL-1α,进而启动IL-1α/p62前馈循环来持续激活NF-κB信号通路 [32]。
对于已形成的肿瘤,许多研究证实其生长和存活高度依赖于持续的KRAS信号,这一现象被称为“癌基因成瘾”(oncogene addiction)。在胶质母细胞瘤和晚期胰腺导管腺癌(PDAC)的小鼠模型中,抑制或敲除突变KRAS的表达能够导致肿瘤细胞凋亡、肿瘤消退,并显著延长生存期 [33, 34]。然而,KRAS的“成瘾性”并非绝对。研究发现,一部分胰腺癌细胞在KRAS功能完全丧失后仍能存活,这些细胞通过激活磷脂酰肌醇3-激酶(PI3K)依赖性的MAPK信号通路来维持其生存 [35]。更有趣的是,KRAS的缺失反而揭示了其在抑制转移相关基因表达中的作用 [35]。此外,肿瘤细胞可以通过激活YAP1信号通路来绕过对KRAS的依赖,从而在KRAS被抑制后恢复生存和增殖能力 [36, 37]。这些发现揭示了肿瘤细胞为抵抗KRAS靶向治疗而演化出的复杂适应性机制。
KRAS突变通过广泛的转录和信号重编程来发挥其致癌功能。突变KRAS能够触发一个特定的转录印记,该印记可用于对不同来源的人类癌症进行分类 [29]。例如,转录因子FOSL1被确定为KRAS驱动的肺癌和胰腺癌中的一个关键脆弱性,它将KRAS致癌信号与有丝分裂机器(如AURKA激酶)联系起来,揭示了新的治疗靶点 [38]。值得注意的是,不同的KRAS突变亚型(如G12C、G12V、G12D)在蛋白质行为、信号转导和临床预后方面表现出显著差异 [39]。此外,突变KRAS的拷贝数也至关重要,拷贝数增加的细胞表现出更强的糖酵解转换和代谢重编程 [40]。野生型KRAS等位基因的缺失也能增强致癌信号,促进所有RAS亚型的激活,从而加速白血病的发生 [41]。
在代谢层面,KRAS突变对肿瘤细胞的代谢网络进行了系统性重塑,以满足其快速增殖的能量和生物合成需求。致癌性KRAS通过刺激葡萄糖摄取,并将葡萄糖中间产物导入己糖胺生物合成途径(HBP)和戊糖磷酸途径(PPP),来维持肿瘤生长 [34]。特别是,KRAS通过MAPK-MYC-RPIA信号轴上调核苷酸的生物合成 [42]。KRAS还能激活脂肪酸合酶(FASN),促进脂肪生成 [43],并通过调控脂肪酸延长酶ELOVL6的活性来影响自身蛋白的稳定性,抑制ELOVL6可诱导KRAS-G12V降解 [44]。更有研究发现,KRAS突变细胞通过巨噬细胞吞饮作用(macropinocytosis)获取铜,并上调铜外排蛋白ATP7A以避免铜毒性,从而对铜的生物利用度产生特殊依赖 [45]。同时,KRAS突变还通过上调PCSK9来调控胆固醇代谢,促进对KRAS激活至关重要的GGPP的积累 [46]。
KRAS的致癌作用还延伸至肿瘤细胞外部,通过重塑肿瘤微环境(TME)来促进免疫逃逸。致癌KRAS信号通过PI3K/STAT3通路抑制miR-34a的表达,从而上调其靶基因CD47的表达。CD47作为一种“别吃我”信号,能够抑制巨噬细胞对肿瘤细胞的吞噬作用,帮助肿瘤逃避先天免疫监视 [47]。此外,KRAS还能诱导COX2的表达,后者通过其产物前列腺素E2(PGE2)促进对免疫检查点抑制剂(ICB)的抵抗 [48]。这些发现表明,KRAS突变不仅驱动细胞内信号,还主动营造了一个免疫抑制的微环境。
综上所述,KRAS在肿瘤中的角色是多维度的,涉及细胞自主性的增殖、存活、代谢和转录重编程,以及与肿瘤微环境的复杂互作。KRAS蛋白的不同突变亚型、表达水平、翻译后修饰(如C118位点的氧化还原依赖性激活) [49] 乃至其特异性的互作蛋白(如PIP5K1A) [50] 都对其功能产生了精细的调控。对这些多维角色的深入理解,不仅揭示了KRAS驱动癌症的复杂性,也为开发新型治疗策略,如靶向GTP负载的KRAS(on)状态的PROTAC降解剂 [51] 或广谱KRAS抑制剂 [52, 53],提供了新的理论基础和靶点。4. KRAS与肿瘤微环境的相互作用 (Interaction between KRAS and the Tumor Microenvironment)
KRAS突变调控肿瘤微环境的关键分子与机制名称内容/功能引用(PMID)炎症与细胞因子网络NF-κB核心炎症枢纽,被致癌性KRAS激活,驱动炎性表型。[54, 55]IL-1α被KRAS G12D诱导,激活NF-κB并形成正反馈回路,持续放大炎症信号。[32]IL-1β来源于宿主髓系细胞,加剧KRAS突变肿瘤对IKKα的依赖,促进CXCL1分泌。[56]CCL5 / IL-6由KRAS信号通过TBK1/IKKε调控,形成支持肿瘤发生的自分泌细胞因子回路。[57]CXCR2配体 (KC, MIP-2)由肿瘤细胞高表达,招募中性粒细胞和内皮细胞,促进炎症和血管生成。[58]Twist被KRAS上调,抑制p16 INK4A,从而抑制炎症相关的细胞衰老,帮助肿瘤启动。[59, 60]基质与细胞外基质重塑IGF1R/AXL-AKT轴在癌相关成纤维细胞(CAFs)中被激活,向肿瘤细胞发出往复信号,调控其增殖与凋亡。[61]TFCP2 / BMP4 / WNT5BKRAS激活TFCP2,上调BMP4/WNT5B,将CAFs转化为促进血管生成的富脂CAFs。[62]KRAS-ITGA3-SRC轴介导KRAS信号与细胞外基质(ECM)的相互作用,影响早期肺癌的细胞状态。[63]免疫逃逸PD-L1 / CXCL10 / CXCL11KRAS G12D突变导致其表达/分泌减少,造成免疫抑制微环境。[64]CD47"别吃我"信号,被KRAS通过PI3K/STAT3/miR-34a轴上调,抑制巨噬细胞吞噬。[47]CSF2 / 乳酸由KRAS突变细胞分泌,将巨噬细胞重编程为促肿瘤的M2样肿瘤相关巨噬细胞(TAMs)。[65]COX2 / PGE2KRAS上调COX2,通过前列腺素E2(PGE2)介导对免疫检查点抑制剂的耐药。[48]circATXN7被KRAS激活的环状RNA,使肿瘤特异性T细胞对活化诱导的细胞死亡更敏感。[66]治疗耐药FGFR1 / AXL (RTKs)被TME中的生长因子激活,介导对KRAS G12C抑制剂的适应性耐药。[67, 68, 69]整合素 / FAK其信号通路是重要的耐药机制,抑制FAK可与KRAS抑制剂产生协同效应。[70]细胞外囊泡 (EVs)将致癌性KRAS蛋白或mRNA传递给周围细胞,增强化疗耐药性。[71]
KRAS突变的致癌作用远不止于细胞自主性效应。越来越多的证据表明,KRAS突变通过重塑肿瘤微环境(Tumor Microenvironment, TME),主动建立一个有利于肿瘤生长、血管生成、侵袭和免疫逃逸的“生态位”。这种相互作用是双向的,即KRAS突变细胞改变TME,而TME反过来又影响肿瘤细胞的行为和治疗反应 [72, 73]。
4.1 KRAS驱动的炎症和细胞因子网络
KRAS突变肿瘤的一个显著特征是其炎性表型。致癌性KRAS能够通过多种机制激活NF-κB信号通路,这是调控炎症反应的核心枢纽 [54, 55]。例如,在胰腺导管腺癌(PDAC)中,KRAS G12D突变可激活AP-1,诱导IL-1α的表达,进而激活NF-κB,并形成IL-1α/p62正反馈回路,持续放大炎症信号 [32]。在恶性胸腔积液中,宿主髓系细胞来源的IL-1β能够加剧KRAS突变肿瘤细胞对IKKα的依赖,促进CXCL1的分泌,从而加剧局部炎症 [56]。此外,KRAS信号还通过TBK1/IKKε激酶调控自分泌的CCL5和IL-6,形成一个支持肿瘤发生的细胞因子回路 [57]。在KRAS诱导的肺腺癌模型中,肿瘤细胞高表达趋化因子受体CXCR2的配体(如KC和MIP-2,人CXCL8的同源物),这些配体招募中性粒细胞和内皮细胞,促进炎症和血管生成 [58]。值得注意的是,外界因素如高脂饮食可通过上调环氧合酶-2(COX2)加剧炎症反应,从而激活致癌性Kras并诱导PDAC的发生 [74]。然而,KRAS的作用也具有复杂性,在肿瘤起始阶段,它可以通过上调Twist抑制p16 INK4A的表达,从而抑制炎症相关的细胞衰老,使细胞得以逃脱生长停滞,启动肿瘤发生 [59, 60]。
4.2 KRAS对基质和细胞外基质的重塑
肿瘤基质是TME的重要组成部分,但研究表明,基质细胞本身通常不携带其上皮肿瘤细胞中的KRAS突变 [75]。然而,KRAS突变细胞能主动与基质细胞进行“异型细胞信号交流”(heterocellular signaling)。例如,PDAC中的KRAS G12D信号能够作用于癌相关成纤维细胞(Cancer-Associated Fibroblasts, CAFs),而活化的CAFs又通过IGF1R/AXL-AKT轴等通路向肿瘤细胞发出往复信号,从而调控肿瘤细胞的增殖、凋亡和线粒体功能 [61]。在结直肠癌(CRC)中,致癌性KRAS通过激活转录因子TFCP2,上调BMP4和WNT5B的表达,将CAFs转化为富含脂质的CAFs。这些富脂CAFs转而分泌VEGFA,促进肿瘤血管生成和疾病进展 [62]。此外,TGF-β信号通路也参与其中;在Kras驱动的胰腺癌前病变模型中,Tgfbr1单倍剂量不足会减少纤维化和淋巴细胞浸润,显示了其在肿瘤早期发展中的复杂作用 [76]。KRAS信号还可通过KRAS-ITGA3-SRC轴等途径与细胞外基质(ECM)相互作用,影响早期肺癌的细胞状态 [63]。
4.3 KRAS介导的免疫逃逸
KRAS突变肿瘤通常表现为免疫抑制性或“冷”肿瘤微环境,这为肿瘤逃避免疫监视提供了便利。研究表明,KRAS的致癌依赖性(oncogenic dependency)而非突变本身,在塑造肿瘤免疫应答中起主导作用;降低KRAS活性可以促进T细胞向肿瘤内浸润 [77, 78]。KRAS通过多种机制实现免疫逃逸。首先,特定KRAS突变亚型(如NSCLC中的G12D)与PD-L1的低表达以及T细胞趋化因子(如CXCL10/CXCL11)分泌减少相关,导致免疫抑制 [64]。其次,KRAS突变可通过PI3K/STAT3/miR-34a轴上调肿瘤细胞表面的“别吃我”信号CD47,从而抑制巨噬细胞的吞噬作用 [47]。第三,KRAS突变细胞通过分泌集落刺激因子2(CSF2)和乳酸,将巨噬细胞重编程为促进肿瘤生长的M2样肿瘤相关巨噬细胞(TAMs) [65]。第四,KRAS可通过上调COX2,经由前列腺素E2(PGE2)介导对免疫检查点抑制剂(ICB)的耐药 [48]。最近还发现,KRAS激活的环状RNA(circATXN7)能使肿瘤特异性T细胞对活化诱导的细胞死亡更加敏感,从而促进免疫逃逸 [66]。这些发现为联合KRAS抑制剂与免疫疗法(如PD-1/PD-L1阻断剂或Kras肽疫苗)提供了理论基础 [79, 80, 81]。
4.4 TME介导的治疗耐药
TME是KRAS突变肿瘤产生治疗耐药性的关键因素。当KRAS信号通路被抑制时,来自TME的旁路信号可以帮助肿瘤细胞存活。例如,对KRAS G12C抑制剂的适应性耐药常常涉及由TME中的生长因子(如FGF、GAS6)激活的受体酪氨酸激酶(RTK),如FGFR1和AXL,从而重新激活下游通路 [67, 68, 69]。整合素和黏着斑激酶(FAK)介导的信号也是一个重要的耐药机制,抑制FAK可与KRAS抑制剂产生协同抗癌效应 [70]。在多发性骨髓瘤中,骨髓微环境为KRAS突变细胞提供了庇护所,而靶向KRAS的寡核苷酸药物可在该微环境中发挥抗肿瘤作用 [82]。此外,肿瘤细胞还能通过细胞外囊泡(EVs)将致癌性KRAS蛋白或mRNA传递给周围细胞,从而增强化疗耐药性 [71]。这些由TME介导的耐药机制凸显了联合靶向KRAS和TME关键节点的治疗策略的重要性。5. 靶向KRAS突变癌症的治疗策略 (Therapeutic Strategies for KRAS-Mutant Cancers)
KRAS基因突变是人类癌症中最常见的致癌驱动因素之一,尤其在胰腺癌、肺癌和结直肠癌中高发。然而,由于其蛋白表面光滑、缺乏深层疏水性口袋,以及与GTP的亲和力极高,KRAS在过去几十年中一直被认为是“不可成药”的靶点。近年来,随着对KRAS生物学功能和结构的深入理解,以及新药研发技术的突破,针对KRAS突变癌症的治疗策略取得了革命性进展。本节将系统梳理直接抑制KRAS、靶向其上下游通路、利用合成致死策略以及其他新兴疗法的最新研究进展。5.1 直接抑制KRAS蛋白
直接抑制KRAS蛋白的策略总结名称内容/功能引用(PMID)共价抑制剂 (Sotorasib)靶向KRAS G12C。与GDP结合状态下暴露的SII-P口袋中的C12残基共价结合,将KRAS锁定在无活性状态。[83, 84, 85]非共价抑制剂 (MRTX1133)靶向G12C以外的突变(如G12D)。非共价结合SII-P口袋,通过与H95等非保守残基相互作用实现选择性。不同突变亚型(如Q61H)在药物敏感性上存在差异。[86, 87, 88]大分子抑制剂 (DARPins)结合KRAS变构区域(α3/loop7/α4界面),抑制其与效应蛋白的相互作用及二聚化。[89]大分子抑制剂 (Monobodies)选择性结合活性状态的突变KRAS(如G12D, G12V/G12C),发挥抗肿瘤活性。[90, 91]蛋白降解剂 (PROTACs)基于PROTAC技术(如DARPin-VHL融合),选择性诱导突变KRAS蛋白的降解。[92]稳定α螺旋肽模拟SOS1螺旋,直接抑制KRAS的核苷酸交换。[93]聚集诱导肽基于KRAS的易聚集区域(APRs)设计,诱导突变KRAS错误折叠并失活。[94]基因编辑 (CRISPR)利用碱基编辑技术在基因层面直接校正KRAS致癌突变。[95]
直接靶向KRAS蛋白的策略在经历了长期瓶颈后,终于迎来了重大突破,主要集中在对特定突变亚型的抑制剂开发上。
等位基因特异性共价抑制剂:针对KRAS G12C突变的抑制剂是这一领域的里程碑。KRAS G12C突变体在GDP结合状态下,其12位突变的半胱氨酸残基附近会暴露一个“switch-II”口袋(SII-P)[83]。利用这一结构特征,科学家开发了能与该半胱氨酸共价结合的小分子抑制剂,将KRAS G12C锁定在无活性的GDP结合状态,从而阻断其下游信号传导 [84]。Sotorasib (AMG 510) 是首个获批上市的KRAS G12C抑制剂,其I期临床试验证实了在KRAS G12C突变的晚期实体瘤患者中的安全性与抗肿瘤活性,尤其在非小细胞肺癌(NSCLC)患者中,客观缓解率达到32.2%,疾病控制率高达88.1% [85]。这一成功激发了对其他KRAS突变亚型抑制剂的探索。
非共价抑制剂与泛KRAS抑制剂:由于G12C以外的突变(如G12D、G12V)不含可共价结合的半胱氨酸,开发非共价抑制剂成为新的方向。研究发现,SII-P作为KRAS蛋白上的一个优先药物结合位点,在多种突变体(包括G12、G13、Q61突变)中均可被非共价小分子配体靶向,且这种结合不完全依赖于GDP状态 [86]。MRTX1133是首个进入临床研究的强效、选择性KRAS G12D非共价抑制剂。其对KRAS的选择性主要依赖于与KRAS蛋白上非保守的第95位组氨酸(H95)的相互作用,而HRAS和NRAS在该位点为酪氨酸,从而实现了对KRAS亚型的特异性识别 [87]。此外,针对KRAS Q61H突变的研究发现,该突变使KRAS脱离上游调控,并对SHP2抑制剂产生耐药性,提示了不同突变亚型在信号调控和药物敏感性上的差异 [88]。
新型直接抑制策略:除了小分子抑制剂,其他创新的直接抑制策略也在不断涌现。
大分子抑制剂:设计的锚定蛋白重复蛋白(DARPins)能够结合到KRAS蛋白变构区域的α3/loop7/α4界面(H95残基附近),特异性地抑制KRAS与效应蛋白的相互作用及下游信号,甚至抑制KRAS在细胞膜上的二聚化 [89]。同样,针对KRAS G12D或G12V/G12C的单体蛋白(monobodies)也被开发出来,它们能选择性结合活性状态的突变KRAS,在体内外均显示出抗肿瘤活性 [90, 91]。
蛋白降解剂:基于PROTAC(蛋白水解靶向嵌合体)技术的KRAS降解剂是另一个前沿方向。例如,将KRAS特异性的DARPin与VHL E3连接酶融合构建的大分子降解剂,可以选择性地诱导突变KRAS蛋白的降解,从而在KRAS突变肿瘤中发挥抗癌作用 [92]。
其他创新方法:利用稳定α螺旋肽(如模拟SOS1螺旋的肽)直接抑制KRAS的核苷酸交换 [93];或利用从KRAS蛋白序列中发现的易聚集区域(APRs)设计的合成肽,诱导突变KRAS蛋白错误折叠并丧失功能,从而抑制肿瘤生长 [94]。此外,CRISPR碱基编辑技术也被用于直接校正肿瘤细胞中的KRAS致癌突变,为基因治疗提供了新思路 [95]。5.2 靶向KRAS上下游通路和合成致死策略
靶向KRAS的间接治疗策略总结名称内容/功能引用(PMID)上游靶点:ERBB抑制剂在KRAS驱动的肺腺癌中,抑制对肿瘤发生和发展至关重要的ERBB家族RTK网络。[96]上游靶点:FGFR2 + EGFR 双重阻断在KRAS突变的胰腺癌模型中,协同作用,显著减少癌前病变形成。[97]下游靶点:MEK抑制剂 + BCL-XL抑制剂在多种KRAS突变癌细胞中诱导强烈的细胞凋亡,在动物模型中实现肿瘤消退。[98]下游靶点:G12C抑制剂 + (mTOR/IGF1R/MEK)抑制剂在KRAS G12C突变肺癌中,联合用药比单一用药显示出更强的疗效和特异性。[99]下游靶点:PI3K + MAPK通路联合抑制针对PIK3CA共突变导致的MEK抑制剂耐药性,凸显联合靶向的必要性。[100]合成致死:PLK1 + ROCK 联合抑制通过上调p21诱导KRAS突变细胞特异性的G2/M期阻滞和凋亡。[101]合成致死:CHK1 + MK2 联合抑制在KRAS突变细胞中引发有丝分裂灾难,显示强大的协同抗癌效应。[102]合成致死:TAK1 抑制在KRAS依赖的结直肠癌中,通过抑制Wnt信号通路促进细胞凋亡。[103]合成致死:GSK3 抑制在KRAS依赖性肿瘤中,通过上调c-Myc和β-catenin发挥抗肿瘤作用。[104]合成致死:BCL6 抑制在KRAS突变肺癌中,抑制其维持DNA复制的功能,导致DNA损伤和生长停滞。[105]合成致死:Ubc9 抑制/敲低抑制对KRAS驱动的肿瘤转化至关重要的SUMO化E2连接酶,从而抑制结直肠癌生长。[106]合成致死:UHRF1 敲除在KRAS突变肺癌中,导致全局DNA去甲基化,重新激活抑癌基因,抑制肿瘤生长。[107]验证失败的靶点:STK33 抑制(警示案例) 后续研究表明抑制其激酶活性对KRAS依赖性癌细胞的存活没有影响。[108, 109]
由于直接抑制KRAS蛋白(尤其是非G12C突变)仍面临挑战,且耐药性问题突出,靶向其信号通路的上下游组分或寻找其特异性的脆弱性(即合成致死)是重要的补充和联合治疗策略。
靶向上游信号:传统观念认为,KRAS突变会使其摆脱上游信号的调控。然而,越来越多的证据表明这种独立性并非绝对。例如,在KRAS驱动的肺腺癌中,ERBB家族受体酪氨酸激酶(RTKs)网络对于肿瘤的发生和发展至关重要,使用多靶点ERBB抑制剂可以有效抑制肿瘤形成 [96]。同样,在KRAS突变的胰腺癌模型中,FGFR2与EGFR协同作用,双重阻断这两个受体可显著减少癌前病变的形成 [97]。这些发现提示,抑制上游RTK信号输入是削弱KRAS致癌活性的有效途径。
靶向下游信号通路:KRAS主要通过RAF-MEK-ERK和PI3K-AKT-mTOR两条下游通路发挥作用。单独抑制MEK或PI3K通路往往效果有限,因为通路之间存在复杂的反馈激活机制。因此,联合用药成为主流策略。例如,MEK抑制剂与抗凋亡蛋白BCL-XL的抑制剂(如Navitoclax)联用,可在多种KRAS突变癌细胞中诱导强烈的细胞凋亡,并在动物模型中实现显著的肿瘤消退 [98]。对于KRAS G12C突变肺癌,将G12C抑制剂与mTOR、IGF1R和MEK抑制剂联合使用,比单一用药显示出更强的疗效和特异性 [99]。此外,PIK3CA的共突变是导致KRAS突变肿瘤对MEK抑制剂耐药的重要原因,这也凸显了联合靶向PI3K和MAPK通路的必要性 [100]。
合成致死策略:合成致死是指两个非致死性基因突变/抑制的组合导致细胞死亡的现象。针对KRAS突变背景,通过高通量筛选(如RNAi或CRISPR筛选)已发现多个合成致死靶点 [110]。
细胞周期与DNA损伤修复激酶:KRAS突变细胞常表现出内在的基因毒性压力,使其对细胞周期检查点激酶抑制剂敏感。研究发现,联合抑制PLK1和ROCK可通过上调p21诱导KRAS突变细胞特异性的G2/M期阻滞和凋亡 [101]。类似地,联合抑制CHK1和MK2可在KRAS突变细胞中引发有丝分裂灾难,显示出强大的协同抗癌效应 [102]。
信号通路节点:转化生长因子β活化激酶1(TAK1)在KRAS依赖的结直肠癌中是关键的生存激酶,抑制TAK1可通过抑制Wnt信号通路促进细胞凋亡 [103]。糖原合成酶激酶3(GSK3)也被证明是KRAS依赖性肿瘤生长所必需的,抑制GSK3可上调c-Myc和β-catenin,从而发挥抗肿瘤作用 [104]。
转录与表观遗传调控因子:B细胞淋巴瘤因子6(BCL6)作为KRAS-MAPK通路的下游靶点,在维持KRAS突变肺癌细胞的DNA复制中起关键作用,抑制BCL6可导致DNA损伤和生长停滞 [105]。SUMO化修饰通路中的E2连接酶Ubc9对于KRAS驱动的肿瘤转化至关重要,敲低Ubc9可有效抑制结直肠癌的生长 [106]。表观遗传调控因子UHRF1在KRAS突变肺癌中也是一个特异性脆弱性靶点,敲除UHRF1导致全局DNA去甲基化,重新激活抑癌基因,从而抑制肿瘤生长 [107]。
失败的靶点:值得注意的是,并非所有最初发现的潜在靶点都经得起验证。丝氨酸/苏氨酸激酶STK33曾被认为是KRAS的合成致死伴侣,但后续多项研究,包括使用RNAi和特异性小分子抑制剂(如BRD-8899),均表明抑制STK33的激酶活性对KRAS依赖性癌细胞的存活没有影响 [108, 109]。这为合成致死策略的靶点验证提供了重要警示。5.3 靶向KRAS依赖的其他脆弱性
靶向KRAS依赖的其他脆弱性的策略总结名称内容/功能引用(PMID)SLC7A5氨基酸转运蛋白,KRAS突变结直肠癌细胞依赖其满足蛋白质合成需求。[111]SLC7A11 / GSH合成胱氨酸/谷氨酸反向转运蛋白及谷胱甘肽合成途径,与KRAS突变肺腺癌细胞存在合成致死关系。[112]ATF4 / 天冬酰胺合成KRAS通过ATF4通路调控天冬酰胺合成,使肿瘤细胞对天冬酰胺酶敏感。[113]GLUT1 / 高剂量维生素C转运氧化型维生素C(DHA),导致ROS积累并抑制糖酵解,选择性杀死KRAS突变细胞。[114]多胺代谢 / 铁死亡逃逸KRAS突变肿瘤的依赖机制,联合靶向可增强KRAS抑制剂疗效。[115, 116]PDEδ / ORP5/ORP8负责将KRAS转运至质膜的蛋白,抑制其功能可阻断KRAS信号。[117]USP5 / Ubc9 (去泛素化/SUMO化)调控KRAS稳定性和活性的翻译后修饰相关酶,具有治疗潜力。[118, 106]KLF5转录因子,KRAS在肠道肿瘤发生中的重要下游介质。[119]RASH3D19介导正反馈环路以再激活RAS通路,靶向其可克服KRAS抑制剂的适应性耐药。[120]
除了经典的信号通路,KRAS突变还导致肿瘤细胞在代谢、蛋白稳态和生存环境等方面产生新的依赖性,这些依赖性也构成了潜在的治疗靶点。
靶向代谢重编程:KRAS突变肿瘤细胞表现出独特的代谢重编程特征。
氨基酸代谢:KRAS突变结直肠癌细胞对氨基酸转运蛋白SLC7A5高度依赖,以满足其旺盛的蛋白质合成需求 [111]。在肺腺癌中,KRAS突变细胞对胱氨酸/谷氨酸反向转运蛋白SLC7A11和谷胱甘肽(GSH)合成途径也表现出合成致死依赖性 [112]。此外,KRAS通过ATF4通路调控天冬酰胺的生物合成,使肿瘤细胞对天冬酰胺酶敏感 [113]。
糖酵解与氧化应激:KRAS突变细胞表现出增强的糖酵解。利用这一特性,高剂量的维生素C可通过其氧化形式(DHA)被GLUT1转运蛋白大量摄入,耗尽细胞内GSH,导致活性氧(ROS)积累并抑制糖酵解关键酶GAPDH,从而选择性地杀死KRAS突变细胞 [114]。
其他代谢通路:KRAS突变肿瘤还可能依赖于多胺代谢和铁死亡逃逸等机制,联合靶向这些通路与KRAS抑制剂可增强疗效 [115, 116]。
靶向KRAS蛋白定位与翻译后修饰:KRAS蛋白必须正确定位到细胞膜上才能发挥功能。抑制其膜定位过程是另一条思路。例如,抑制负责将KRAS从内膜系统转运至质膜的蛋白(如PDEδ、ORP5/ORP8)可以有效阻断KRAS信号 [117]。此外,一些翻译后修饰,如泛素化和SUMO化,也参与调控KRAS的稳定性和活性,靶向相关的酶(如去泛素化酶USP5或SUMO化连接酶Ubc9)已显示出治疗潜力 [118, 106]。
靶向协同通路与反馈机制:KRAS突变肿瘤的生长不仅依赖于其自身,还依赖于其他协同通路。例如,转录因子KLF5是KRAS在肠道肿瘤发生中的重要下游介质 [119]。同时,KRAS抑制剂的使用会激活多种反馈通路,导致耐药。例如,RASH3D19介导了一个正反馈环路,促进RAS通路的再激活,靶向RASH3D19有望克服对KRAS抑制剂的适应性耐药 [120]。
综上所述,针对KRAS突变癌症的治疗策略已经从“不可成药”的困境中走出,进入了以直接抑制剂为核心,结合靶向合成致死、代谢重编程和免疫微环境等多维度联合治疗的新时代。尽管耐药性仍然是巨大挑战,但对KRAS信号网络和肿瘤脆弱性的深入理解,正不断为开发更有效、更持久的治疗方案提供新的机遇。6. KRAS靶向治疗的耐药机制 (Mechanisms of Resistance to KRAS-Targeted Therapy)
表1. KRAS靶向治疗的耐药机制总结名称内容/功能引用(PMID)基因组/转录组层面KRAS等位基因扩增突变型Kras等位基因拷贝数增加,导致靶蛋白水平升高,使抑制剂浓度不足。[121, 122]KRAS二次获得性突变改变药物结合口袋的构象或激活KRAS,使其不依赖GTP/GDP循环,从而阻止抑制剂结合。[123]旁路激活:上游RTKEGFR、FGFR、IGF-IR等受体酪氨酸激酶被激活,绕过KRAS直接激活下游MAPK通路。[124, 125, 126]旁路激活:PI3K/AKT/mTORPIK3CA等基因的共存突变或反馈性激活,使肿瘤生长不依赖于MEK/ERK信号。[127, 128, 129, 130]旁路激活:YAP1转录共激活因子YAP1扩增和激活,通过SDC1增强营养获取和多条RTK通路激活。[37, 131]旁路激活:LCK/PAK1/β-Catenin在KRAS依赖的非小细胞肺癌中,形成信号网络作为潜在的旁路通路。[132]转录/转录后调控:eIF5A-PEAK1eIF5A-PEAK1正反馈环路异常上调,导致KRAS癌蛋白积累。[133]转录/转录后调控:lncRNA ST8SIA6-AS1通过调控PLK1/c-Myc轴赋予肿瘤原发性和获得性耐药。[134]转录/转录后调控:NRF2KRAS上调转录因子NRF2,可能赋予细胞对靶向治疗的抗性。[135]非基因组层面表型转换:上皮-间质转化(EMT)发生EMT的细胞通过IGFR-IRS1通路维持PI3K信号的持续激活,导致耐药。[136]表型转换:细胞谱系分化肿瘤细胞分化为对药物不敏感的静息状态(如AT1样细胞)或耐药的“经典”上皮状态。[137, 138]代谢重编程:向氧化磷酸化(OXPHOS)转变细胞从依赖糖酵解转向依赖线粒体OXPHOS的药物抵抗状态,与ZBTB11上调有关。[139]代谢重编程:保护性自噬抑制KRAS/ERK通路会增强细胞的自噬通量,作为一种适应性生存机制。[140, 141]代谢重编程:铁死亡逃逸肿瘤细胞通过上调ALDH1A1或FSP1来拮抗药物诱导的铁死亡过程,从而产生耐药。[142, 143]
近年来,针对KRAS G12C突变体的共价抑制剂(如Sotorasib和Adagrasib)的问世,标志着KRAS靶向治疗取得了历史性突破,打破了KRAS“不可成药”的魔咒 [123, 144]。然而,临床实践表明,与许多靶向药物一样,患者不可避免地会产生原发性或获得性耐药,导致肿瘤复发和疾病进展,这极大地限制了这些药物的长期疗效 [145, 125]。深入理解KRAS靶向治疗的耐药机制,对于开发克服耐药的联合治疗策略至关重要。这些机制复杂多样,涉及从基因组改变到细胞表型可塑性、代谢重编程以及肿瘤微环境的动态互作等多个层面。6.1 基因组和转录组层面的耐药机制
6.1.1 KRAS基因自身的改变最直接的耐药机制源于KRAS基因本身的改变。其中,KRAS等位基因扩增是一个关键的获得性耐药机制。在小鼠模型中,对Sotorasib产生耐药的肿瘤表现出突变型Kras等位基因的扩增,导致靶蛋白水平升高,从而使抑制剂浓度不足以完全抑制其活性 [121]。临床数据也表明,KRAS突变等位基因的剂量增加与晚期疾病和不良预后相关 [122]。此外,虽然罕见,但在治疗压力下,KRAS可能出现二次获得性突变,这些突变可能改变药物结合口袋的构象,阻止抑制剂结合,或激活KRAS使其不依赖于GTP/GDP循环 [123]。
6.1.2 旁路信号通路的激活当KRAS本身被有效抑制时,肿瘤细胞可通过激活平行的“旁路”信号通路来维持其增殖和生存。
MAPK通路的再激活:这是最常见的耐药机制之一。即使KRAS被抑制,下游的MAPK通路(RAF-MEK-ERK)也可能通过多种方式被重新激活。上游受体酪氨酸激酶(RTK)的激活,如EGFR、FGFR或IGF-IR,能够绕过KRAS直接激活RAF-MEK-ERK信号 [124]。例如,在KRAS突变的结直肠癌中,PI3K信号通路主要受IGF-IR等RTK调控,而非KRAS本身,这为旁路激活提供了基础 [124]。一项对KRAS G12C抑制剂产生临床耐药的快速尸检研究发现,耐药肿瘤中MAPK通路被再激活,但并未在KRAS或其下游中介物中发现新的突变,这表明非基因突变机制(如RTK的配体依赖性激活)也参与其中 [125, 126]。
PI3K/AKT/mTOR通路的激活:PI3K/AKT/mTOR是另一条关键的生存通路。在KRAS突变肿瘤中,常常伴随PIK3CA等该通路相关基因的突变。这些共存突变可以使肿瘤生长不依赖于MEK/ERK信号,从而导致对MEK抑制剂的原发性耐药 [127, 128]。即使在没有PIK3CA突变的情况下,对KRAS的抑制也可能通过反馈机制激活PI3K/AKT通路 [129]。例如,在KRAS诱导的胰腺肿瘤中,sst2的缺失可通过激活PI3K信号促进肿瘤生长 [130]。
其他信号通路的激活:研究发现,转录共激活因子YAP1的扩增和激活是KRAS抑制后肿瘤复发的重要机制 [37]。在获得性耐药模型中,YAP1激活驱动了Syndecan-1 (SDC1)的表面定位恢复,增强了巨噬胞饮介导的营养获取和多条RTK通路的激活,从而维持肿瘤生存 [131]。此外,在KRAS依赖的非小细胞肺癌中,以LCK、PAK1和β-Catenin为中心的信号网络也被证明是潜在的旁路通路 [132]。
6.1.3 转录和转录后调控耐药性也可能源于转录和翻译水平的适应性变化。例如,一个由翻译延伸因子eIF5A和激酶PEAK1组成的正反馈调节环路可以控制KRAS癌蛋白的积累,该环路的异常上调可能导致耐药 [133]。此外,长链非编码RNA(lncRNA)如ST8SIA6-AS1可通过调控PLK1/c-Myc轴赋予肿瘤对KRAS抑制剂的原发性和获得性耐药 [134]。癌基因KRAS本身也能通过上调NRF2等转录因子赋予细胞对化疗的抗性,这一机制也可能参与靶向治疗的耐药过程 [135]。6.2 非基因组层面的耐药机制
6.2.1 细胞状态可塑性与表型转换肿瘤内部的异质性和细胞状态的可塑性是耐药的主要来源。
上皮-间质转化(EMT):EMT已被证实是KRAS G12C抑制剂在非小细胞肺癌中产生原发性和获得性耐药的原因之一。发生EMT的细胞即使在KRAS G12C被抑制的情况下,仍能通过IGFR-IRS1通路维持PI3K信号的持续激活 [136]。
细胞谱系分化和状态转换:在KRAS驱动的肺腺癌中,KRAS抑制剂可诱导肿瘤细胞向一种静息的、类似肺泡I型(AT1)细胞的状态分化,这些细胞对药物不敏感,并在停药后迅速增殖,导致肿瘤复发 [137]。类似地,在胰腺癌中,对KRAS的抑制会富集处于“经典”上皮状态的癌细胞,这些细胞是耐药并导致疾病复发的“储备库”,而“基底样”状态的细胞则对抑制剂敏感 [138]。
6.2.2 代谢重编程代谢重编程是肿瘤细胞应对治疗压力的重要适应性策略。
向氧化磷酸化(OXPHOS)的转变:研究表明,胰腺导管腺癌(PDAC)细胞在接受KRAS抑制剂治疗后,会迅速发生代谢重编程,从依赖糖酵解转向依赖线粒体氧化磷酸化(OXPHOS)的药物抵抗状态。这一过程与转录因子ZBTB11的上调有关,通过降解ZBTB11可以逆转该代谢状态并恢复对KRAS抑制剂的敏感性 [139]。
自噬的保护性作用:抑制KRAS或其下游的ERK信号通路会意外地增强细胞的自噬通量,这是一种细胞在代谢压力下的生存机制。因此,自噬成为一种适应性耐药机制。联合使用ERK抑制剂和自噬抑制剂(如氯喹)在KRAS驱动的胰腺癌模型中显示出显著的协同抗肿瘤效果 [140, 141]。
铁死亡的逃逸:KRAS抑制剂可诱导铁死亡,但肿瘤细胞可通过上调醛脱氢酶1家族成员A1(ALDH1A1)或铁死亡抑制蛋白1(FSP1)来拮抗这一过程,从而产生耐药性 [142, 143]。
综上所述,KRAS靶向治疗的耐药机制是多维度、动态且相互关联的复杂网络。从KRAS基因自身的改变,到旁路信号通路的激活,再到细胞表型和代谢状态的深刻重塑,共同构成了肿瘤逃避药物抑制的屏障。这些发现不仅解释了单一靶向药物疗效有限的原因,也为未来的治疗策略指明了方向,即必须采取多靶点、多途径的联合治疗方案,例如将KRAS抑制剂与MEK抑制剂、SHP2抑制剂、自噬抑制剂或针对特定代谢脆弱性的药物联用,以期实现更持久和更深度的临床缓解 [140, 146]。7. 临床转化与未来展望 (Clinical Translation and Future Perspectives)
KRAS靶向治疗及未来策略总结策略/靶点内容/功能引用(PMID)直接抑制剂KRAS G12C抑制剂 (Sotorasib, Adagrasib)共价抑制KRAS G12C突变体,在NSCLC、CRC、PDAC等癌种中显示出临床疗效。[154], [155], [156]KRAS G12D抑制剂 (临床前)通过与突变蛋白Asp12残基形成盐桥来特异性靶向KRAS G12D。[157]间接靶向与联合策略靶向合成致死伙伴 (CNK1, EZH2, WT1)抑制KRAS突变细胞所依赖的特定蛋白,如抑制CNK1的PH结构域或调控细胞衰老与增殖的WT1;基于等位基因特异性联合使用EZH2抑制剂。[158], [159], [160]靶向蛋白相互作用/复合物破坏KRAS与下游效应蛋白(如RAF)的相互作用或KRAS自身的二聚化,或稳定其“预激活”中间状态。[161], [162], [163]靶向野生型KRAS在某些KRAS野生型肿瘤(如HCC)中,上调的野生型KRAS也可驱动肿瘤进展,具有治疗价值。[164]未来展望与挑战克服耐药性揭示耐药机制(如旁路激活、次级突变),开发合理的联合用药策略以延缓或克服耐药。[165], [166]广谱/泛KRAS疗法 (PROTACs)开发能覆盖G12D、G12V等多种突变类型的抑制剂或基于蛋白质降解技术的降解剂。[167], [168], [169]联合免疫疗法 (ICB)将KRAS抑制剂与免疫检查点抑制剂(ICB)联合,重塑肿瘤免疫微环境,产生协同抗肿瘤效应。[170], [171]新型治疗模式 (疫苗, TCR-T)开发靶向KRAS突变新抗原的治疗性疫苗和过继性T细胞疗法(TCR-T)。[170], [172]
KRAS作为首个被发现的人类肿瘤基因,在过去四十年中一直是癌症研究的焦点。然而,由于其蛋白表面光滑、缺乏深层疏水性口袋以及对GTP的高亲和力,KRAS曾一度被认为是“不可成药”的靶点 [123, 147]。随着对KRAS生物学功能的深入理解和药物研发技术的突破,特别是KRAS G12C共价抑制剂的成功,KRAS靶向治疗进入了一个全新的时代。本节将总结KRAS研究的临床意义,并对未来的研究方向和治疗前景进行展望。7.1 KRAS作为临床生物标志物的意义
KRAS突变在临床实践中的首要应用是作为预测性生物标志物。早在2008年,一项里程碑式的研究就证实,只有KRAS野生型(wild-type, WT)的转移性结直肠癌(mCRC)患者才能从抗EGFR单克隆抗体(如帕尼单抗)治疗中获益 [148]。类似地,在非小细胞肺癌(NSCLC)中,KRAS突变也被发现与EGFR酪氨酸激酶抑制剂(TKI,如吉非替尼和厄洛替尼)的原发性耐药相关 [149]。这些发现确立了KRAS突变检测在指导靶向治疗中的关键地位,避免了无效治疗带来的毒副作用和经济负担。然而,KRAS在NSCLC中作为预测标志物的价值曾一度存在争议,部分原因是早期研究样本量较小以及KRAS突变在NSCLC中的异质性 [150, 151]。
除了作为预测标志物,KRAS突变的预后价值也日益受到关注。不同KRAS突变亚型对患者预后的影响存在差异。例如,一项针对肝内胆管癌(ICC)的研究表明,不同的KRAS突变亚型与患者术后的总生存期和无病生存期显著相关,提示精细化的突变分型对于评估患者风险具有重要意义 [152]。此外,KRAS突变常与其他基因突变(如BRAF)同时存在,这种复杂的突变模式可能共同影响肿瘤的生物学行为和患者预后,提示在临床决策中需要考虑肿瘤基因组的全景信息 [153]。7.2 KRAS靶向治疗的临床转化
直接抑制剂的突破:KRAS G12C抑制剂(如Sotorasib和Adagrasib)的问世,彻底改变了KRAS突变肿瘤的治疗格局 [154]。这些药物在携带KRAS G12C突变的NSCLC和CRC患者中显示出显著的临床疗效 [155]。尽管KRAS G12C突变在胰腺导管腺癌(PDAC)中仅占1%-2%,但针对这一靶点的精准治疗同样展现了巨大潜力,为这部分难治性肿瘤患者带来了新的希望 [156]。然而,G12C突变仅占所有KRAS突变的一部分,开发针对G12D、G12V等更常见突变类型的抑制剂是当前亟待解决的挑战。已有研究设计出可通过与突变蛋白Asp12残基形成盐桥来特异性靶向KRAS G12D的抑制剂,并在临床前模型中验证了其潜力 [157]。
间接靶向与联合治疗策略:在直接抑制剂取得突破的同时,间接靶向KRAS信号通路的策略也在不断发展。
靶向合成致死伙伴:通过抑制KRAS突变细胞所依赖的特定蛋白来杀死肿瘤细胞是一种有效的策略。例如,抑制支架蛋白CNK1的PH结构域可选择性地阻断KRAS突变细胞的生长 [158]。另一项研究发现,EZH2的表达受到KRAS信号的调控,且调控通路因KRAS突变亚型(G12C依赖MEK-ERK,而G12D依赖PI3K-AKT)而异,这为基于等位基因特异性的联合用药(如EZH2抑制剂联合MEK或PI3K抑制剂)提供了理论依据 [159]。此外,调控KRAS驱动的细胞衰老与增殖平衡的转录因子WT1也可能成为新的治疗靶点 [160]。
靶向蛋白相互作用与复合物:KRAS功能的发挥依赖于其在细胞膜上的正确定位和与下游效应蛋白(如RAF)的相互作用。高分辨率的冷冻电镜技术揭示了KRAS与BRAF、MEK1和14-3-3形成的自抑制复合物结构,发现KRAS的结合本身不足以激活BRAF,还需要膜募集过程。这提示,稳定这种“预激活”中间状态可能成为一种新的治疗策略,而非仅仅阻断结合 [161]。对KRAS在膜上形成同源二聚体及其与RAF相互作用的深入研究,也为开发破坏这些关键蛋白复合物的小分子药物提供了结构基础 [162, 163]。
靶向非经典KRAS驱动的肿瘤:研究发现,在某些KRAS野生型的肿瘤中,如肝细胞癌(HCC),野生型KRAS蛋白的表达上调同样可以驱动肿瘤进展并导致对索拉非尼的耐药。这表明,靶向野生型KRAS在特定癌症中也具有重要的临床价值 [164]。7.3 未来展望与挑战
尽管KRAS靶向治疗取得了历史性突破,但仍面临诸多挑战,同时也为未来的研究指明了方向。
克服耐药性:与所有靶向药物一样,KRAS抑制剂的耐药性是限制其长期疗效的主要障碍。未来的研究需深入揭示耐药的分子机制,包括旁路激活、次级突变以及肿瘤微环境重塑等,并据此开发合理的联合用药策略以延缓或克服耐药 [165, 166]。
开发广谱及等位基因特异性疗法:当前临床获益主要局限于KRAS G12C突变患者。开发能够覆盖G12D、G12V等更多突变类型的“泛KRAS”抑制剂,或针对不同突变亚型的特异性药物,是扩大受益人群的关键 [167, 168]。基于蛋白质降解技术(如PROTACs)的KRAS降解剂,能够有效降解多种KRAS突变体,展现了广阔的应用前景 [169]。
与免疫疗法联合:越来越多的证据表明,KRAS突变能够塑造一个免疫抑制性的肿瘤微环境。因此,将KRAS抑制剂与免疫检查点抑制剂(ICB)等免疫疗法相结合,有望重塑肿瘤免疫微环境,产生协同抗肿瘤效应,是目前极具潜力的研究方向 [170, 171]。
探索新型治疗模式:除了小分子抑制剂和降解剂,其他创新的治疗方式也在积极探索中,例如靶向KRAS突变新抗原的治疗性疫苗和过继性T细胞疗法(TCR-T),这些策略旨在利用人体自身免疫系统精准清除携带KRAS突变的肿瘤细胞 [170, 172]。
深化对KRAS生物学的理解:KRAS突变不仅仅是一个简单的“开关”,其致癌过程复杂且具有组织和细胞谱系特异性。例如,在造血系统恶性肿瘤中,致癌性Kras突变本身不足以导致恶性转化,还需要二次打击事件的协同作用,且其起始靶细胞为造血干细胞 [173]。深入理解KRAS在不同肿瘤类型、不同突变亚型以及与肿瘤微环境相互作用中的独特生物学功能,将为开发更精准、更有效的治疗策略提供新的思路。
总之,KRAS研究已经从基础科学的探索成功迈向临床应用的转化。随着KRAS G12C抑制剂的成功,我们有理由相信,通过开发更广谱的抑制剂、设计更智能的联合治疗方案以及探索全新的治疗模式,人类终将攻克KRAS这个曾经“不可战胜”的癌症靶点,为广大癌症患者带来福音。8. 结论 (Conclusion)
作为发现最早、突变频率最高的癌基因之一,KRAS在过去几十年中一直是肿瘤研究的焦点。得益于分子生物学、基因组学和化学生物学的飞速发展,我们对KRAS突变体的分子结构、信号转导网络、代谢重编程及其在肿瘤发生发展中的核心驱动作用有了前所未有的深刻理解 [174, 175]。研究揭示了KRAS突变不仅通过经典的MAPK和PI3K通路驱动细胞增殖,还通过调控非编码RNA [176]、蛋白质泛素化修饰 [177]、表观遗传 [178] 和氧化还原稳态 [179] 等多种机制,在不同组织和细胞起源中启动并维持肿瘤的恶性表型 [180, 181, 182]。
在经历了漫长的“不可成药”时期后,针对KRAS G12C突变体的共价抑制剂(如Sotorasib和Adagrasib)的成功开发和临床应用,无疑是KRAS靶向治疗乃至整个精准医疗领域的里程碑式突破 [85, 183, 184]。这些药物的出现首次证明了直接靶向KRAS突变体是可行的,为攻克这一顽固靶点带来了希望。
然而,临床实践和基础研究迅速揭示了严峻的耐药性问题。无论是内源性耐药还是获得性耐药,都极大地限制了直接KRAS抑制剂的长期疗效 [185, 186]。耐药机制极为复杂,包括KRAS基因本身的扩增、旁路信号通路的激活(如受体酪氨酸激酶RTK)、细胞状态的可塑性(如上皮-间质转化)以及对化疗药物的交叉耐药性增强等 [187, 188]。此外,KRAS突变肿瘤细胞还能通过上调替代表错修复通路来应对DNA损伤,进一步增加了治疗难度 [189]。
为了克服耐药并开发更持久的治疗策略,未来的研究必须转向一个更宏观和系统的视角。整合基因组学、蛋白质组学、代谢组学等多组学数据,将有助于我们绘制出KRAS驱动的肿瘤全景图谱 [190, 191]。这不仅能揭示新的治疗靶点,更能让我们深入理解KRAS如何重塑整个肿瘤生态系统(Tumor Ecosystem),包括其与免疫细胞、基质细胞、代谢物乃至微生物群的复杂互作网络 [192, 193]。例如,KRAS突变能够通过重编程肿瘤代谢(如增强糖酵解和谷氨酰胺分解)来支持自身生长,同时也为靶向代谢通路提供了新的治疗契机 [194, 195]。在此基础上,开发更智能、更有效的个体化联合治疗策略是未来的核心方向,例如将直接KRAS抑制剂与靶向其下游或旁路信号通路的药物(如RAF/MEK抑制剂)相结合 [196],或者联合靶向肿瘤生态系统中关键节点的疗法,以及靶向KRAS活性状态的组合疗法等 [197]。
综上所述,尽管挑战重重,但随着我们对KRAS生物学复杂性的认知不断深化,以及创新疗法的不断涌现,我们有理由相信,通过多学科交叉融合和个体化精准施策,人类终将攻克KRAS突变这一顽固的癌症驱动因素。Supplementary Studies / Further Reading补充研究/延伸阅读
除了本文先前章节中详细讨论的核心主题外,还有大量研究从不同角度扩展了我们对KRAS生物学及其在癌症中作用的理解。这些研究涵盖了共突变、非经典KRAS驱动机制、新的调控网络以及创新的治疗策略,为未来的研究和临床应用提供了重要线索。
一个关键的研究方向是探索KRAS突变与其他基因改变的相互作用及其临床意义。一项针对早期可切除非小细胞肺癌(NSCLC)的大型汇总分析表明,TP53与KRAS的共突变状态在接受辅助化疗或观察的患者中,并未显示出对总生存期或无病生存期的显著预后影响 [198]。然而,在局部晚期(III期)非鳞状NSCLC中,情况有所不同。一项多中心回顾性研究发现,在接受同步放化疗和度伐利尤单抗(durvalumab)巩固治疗的患者中,携带KRAS突变的患者其中位无进展生存期(PFS)显著短于KRAS野生型患者,这表明KRAS突变可能是该治疗方案下的一个不良预后因素 [199]。从机制上看,KRAS突变常与KEAP1功能丧失性突变在约20%的肺腺癌(LUAD)中同时发生。研究证明,这种共存状态是导致KRAS突变型LUAD在免疫和代谢方面表现出显著异质性的一个关键驱动因素,为理解肿瘤微环境和制定个性化治疗策略提供了新视角 [200]。
尽管KRAS突变在约90%的胰腺癌中起着核心驱动作用,但仍有部分胰腺癌为KRAS野生型。一项利用基因工程小鼠模型的研究揭示,p53和SMAD4的联合缺失可以在没有致癌性KRAS突变的情况下,诱导腺鳞癌亚型胰腺癌的自发形成,这为KRAS野生型胰腺癌的发生提供了重要的替代致病机制 [201]。此外,KRAS在血液系统恶性肿瘤中的作用也日益受到关注 [202]。例如,在多发性骨髓瘤(MM)中,泛素特异性蛋白酶2(USP2)被鉴定为一种新型的KRAS去泛素化酶。通过药物或基因手段抑制USP2可导致KRAS降解,从而抑制MM细胞的增殖,这提示靶向KRAS降解途径可能成为治疗相关血液肿瘤的新策略 [203]。
近年来,对KRAS信号通路上下游调控的精细机制研究取得了显著进展。研究发现,F-box蛋白FBXL6能够通过促进野生型和突变型KRAS的多聚泛素化来激活其功能,揭示了一个新的KRAS上游调控因子 [204]。在KRAS突变的结直肠癌(CRC)中,丙酮酸脱氢酶磷酸酶催化亚基1(PDP1)被发现是一个关键的进展调节因子,它作为支架蛋白增强BRAF和MEK1的相互作用,从而激活MAPK信号通路 [205]。另一项研究发现,在LUAD中,KRAS/PI3K轴驱动了通用转录因子III C亚基6(GTF3C6)的表达,后者通过调节FAK磷酸化促进肿瘤发生 [206]。此外,转录因子Miz1通过抑制原钙粘蛋白Pcdh10的表达来促进KRAS驱动的肺肿瘤发生 [207]。更有开创性的工作表明,通过在活细胞上进行蛋白质靶向的聚糖编辑,可以直接破坏KRAS信号传导,为靶向KRAS提供了全新的化学干预手段 [208]。
KRAS的功能状态与细胞内其他关键生物学过程紧密相连。例如,在LUAD中,致癌性KRAS的“成瘾”状态差异性地影响着MTH1(一种8-氧代鸟嘌呤核苷三磷酸酶)的表达和活性,而MTH1对于防止氧化核苷酸掺入基因组至关重要。抑制KRAS可导致MTH1表达下降,这揭示了KRAS状态与细胞氧化还原稳态之间的联系 [209]。在胰腺癌中,研究发现KRAS/ABHD17C/ALOX15B轴通过促进铁死亡逃逸来驱动肿瘤进展,为KRAS突变型胰腺癌提供了新的治疗靶点 [210]。
对KRAS结构和药物反应的深入研究也为治疗策略的开发提供了宝贵信息。一项基于核磁共振的研究揭示,效应蛋白(如RAF1的RBD-CRD结构域)的结合会依次改变KRAS在细胞膜上的二聚化模式,从而为RAS介导的RAF激活机制提供了新的结构见解 [211]。早期的研究已经显示出基于KRAS状态的差异化治疗潜力。例如,多激酶抑制剂索拉非尼(Sorafenib)在KRAS野生型NSCLC细胞中主要靶向B-RAF,而在KRAS突变细胞中则主要靶向C-RAF,尽管它在两种细胞中均能抑制生长 [212]。另一项研究表明,内源性突变KRAS(D13)的缺失会降低结直肠癌细胞对溶瘤病毒ReovirusT3D和奥沙利铂诱导的凋亡敏感性,但对TRAIL诱导的凋亡无影响,这提示特定KRAS突变亚型在决定治疗反应中的复杂作用 [213]。利用KRAS突变肿瘤增强的巨噬胞饮作用,研究人员开发了靶向该途径的肽-多西他赛偶联药物,实现了对胰腺癌细胞的旁观者杀伤效应,为泛KRAS突变癌症的治疗提供了新思路 [214]。最后,在临床诊断方面,对循环肿瘤DNA(ctDNA)中KRAS突变的检测显示出巨大的应用潜力,一项研究表明,治疗前在晚期NSCLC患者的ctDNA中检测到KRAS突变具有重要的临床价值 [215]。这些补充研究共同描绘了一幅更加完整和复杂的KRAS生物学图景,并不断为攻克KRAS驱动的癌症带来新的希望。References
Kristina Drizyte-Miller, Taiwo Talabi, Ashwin Somasundaram, Adrienne D Cox, Channing J Der KRAS: the Achilles' heel of pancreas cancer biology. J Clin Invest. 2025;135(16). PMID: 40829181. doi: 10.1172/JCI191939.
Joshua H Cook, Giorgio E M Melloni, Doga C Gulhan, Peter J Park, Kevin M Haigis The origins and genetic interactions of KRAS mutations are allele- and tissue-specific. Nat Commun. 2021;12(1):1808. PMID: 33753749. doi: 10.1038/s41467-021-22125-z.
Gongmin Zhu, Lijiao Pei, Hongwei Xia, Qiulin Tang, Feng Bi Role of oncogenic KRAS in the prognosis, diagnosis and treatment of colorectal cancer. Mol Cancer. 2021;20(1):143. PMID: 34742312. doi: 10.1186/s12943-021-01441-4.
Lamei Huang, Zhixing Guo, Fang Wang, Liwu Fu KRAS mutation: from undruggable to druggable in cancer. Signal Transduct Target Ther. 2021;6(1):386. PMID: 34776511. doi: 10.1038/s41392-021-00780-4.
Kaushal Parikh, Giuseppe Banna, Stephen V Liu, Alex Friedlaender, Aakash Desai, Vivek Subbiah, et al. Drugging KRAS: current perspectives and state-of-art review. J Hematol Oncol. 2022;15(1):152. PMID: 36284306. doi: 10.1186/s13045-022-01375-4.
Susanna Cogoi, Luigi E Xodo G-quadruplex formation within the promoter of the KRAS proto-oncogene and its effect on transcription. Nucleic Acids Res. 2006;34(9):2536-49. PMID: 16687659.
Kai-Bo Wang, Yushuang Liu, Jinzhu Li, Chengmei Xiao, Yingying Wang, Wei Gu, et al. Structural insight into the bulge-containing KRAS oncogene promoter G-quadruplex bound to berberine and coptisine. Nat Commun. 2022;13(1):6016. PMID: 36224201. doi: 10.1038/s41467-022-33761-4.
Young Seok Cho, Gui Chul Kim, Hye Min Lee, Byoungmo Kim, Ha Rin Kim, Seung Woo Chung, et al. Albumin metabolism targeted peptide-drug conjugate strategy for targeting pan-KRAS mutant cancer. J Control Release. 2022;344:26-38. PMID: 35202743. doi: 10.1016/j.jconrel.2022.02.026.
Minh D To, Christine E Wong, Anthony N Karnezis, Reyno Del Rosario, Roberto Di Lauro, Allan Balmain Kras regulatory elements and exon 4A determine mutation specificity in lung cancer. Nat Genet. 2008;40(10):1240-4. PMID: 18758463. doi: 10.1038/ng.211.
Shuangni Yu, Zhaohui Lu, Changzheng Liu, Yunxiao Meng, Yihui Ma, Wugan Zhao, et al. miRNA-96 suppresses KRAS and functions as a tumor suppressor gene in pancreatic cancer. Cancer Res. 2010;70(14):6015-25. PMID: 20610624. doi: 10.1158/0008-5472.CAN-09-4531.
Moiez Ali, Erin Kaltenbrun, Gray R Anderson, Sarah Jo Stephens, Sabrina Arena, Alberto Bardelli, et al. Codon bias imposes a targetable limitation on KRAS-driven therapeutic resistance. Nat Commun. 2017;8:15617. PMID: 28593995. doi: 10.1038/ncomms15617.
Nicole L K Pershing, Benjamin L Lampson, Jason A Belsky, Erin Kaltenbrun, David M MacAlpine, Christopher M Counter Rare codons capacitate Kras-driven de novo tumorigenesis. J Clin Invest. 2015;125(1):222-33. PMID: 25437878. doi: 10.1172/JCI77627.
Dae Gyu Kim, Yongseok Choi, Yuno Lee, Semi Lim, Jiwon Kong, JaeHa Song, et al. AIMP2-DX2 provides therapeutic interface to control KRAS-driven tumorigenesis. Nat Commun. 2022;13(1):2572. PMID: 35546148. doi: 10.1038/s41467-022-30149-2.
Malte Schmick, Nachiket Vartak, Björn Papke, Marija Kovacevic, Dina C Truxius, Lisaweta Rossmannek, et al. KRas localizes to the plasma membrane by spatial cycles of solubilization, trapping and vesicular transport. Cell. 2014;157(2):459-471. PMID: 24725411. doi: 10.1016/j.cell.2014.02.051.
Gunther Zimmermann, Björn Papke, Shehab Ismail, Nachiket Vartak, Anchal Chandra, Maike Hoffmann, et al. Small molecule inhibition of the KRAS-PDEδ interaction impairs oncogenic KRAS signalling. Nature. 2013;497(7451):638-42. PMID: 23698361. doi: 10.1038/nature12205.
Yonglu Che, Zurab Siprashvili, Joanna R Kovalski, Tiffany Jiang, Glenn Wozniak, Lara Elcavage, et al. KRAS regulation by small non-coding RNAs and SNARE proteins. Nat Commun. 2019;10(1):5118. PMID: 31712554. doi: 10.1038/s41467-019-13106-4.
Junchen Liu, Ransome van der Hoeven, Walaa E Kattan, Jeffrey T Chang, Dina Montufar-Solis, Wei Chen, et al. Glycolysis regulates KRAS plasma membrane localization and function through defined glycosphingolipids. Nat Commun. 2023;14(1):465. PMID: 36709325. doi: 10.1038/s41467-023-36128-5.
Tao Yuan, Yue Liu, Ruilin Wu, Meijia Qian, Weihua Wang, Yonghao Li, et al. Josephin Domain Containing 2 (JOSD2) inhibition as Pan-KRAS-mutation-targeting strategy for colorectal cancer. Nat Commun. 2025;16(1):3623. PMID: 40240366. doi: 10.1038/s41467-025-58923-y.
Han Koo, Kyung Chan Park, Hyun Ahm Sohn, Minho Kang, Dong Joon Kim, Zee-Yong Park, et al. Anti-proteolytic regulation of KRAS by USP9X/NDRG3 in KRAS-driven cancer development. Nat Commun. 2025;16(1):628. PMID: 39819877. doi: 10.1038/s41467-024-54476-8.
Tonci Ivanisevic, Yan Ma, Emiel Van Boxel, Wout Magits, Benoit Lechat, Zeynep Koçberber, et al. Intrinsic resistance to RAS inhibitors is driven by dysregulation of KRAS degradation. Nat Commun. 2025;17(1):411. PMID: 41397976. doi: 10.1038/s41467-025-67109-5.
Shihui Shen, Qiansen Zhang, Yuhan Wang, Hui Chen, Shuangming Gong, Yun Liu, et al. Targeting ubiquitin-independent proteasome with small molecule increases susceptibility in pan-KRAS-mutant cancer models. J Clin Invest. 2025;135(6). PMID: 40091835. doi: 10.1172/JCI185278.
Carles Barceló, Noelia Paco, Mireia Morell, Blanca Alvarez-Moya, Neus Bota-Rabassedas, Montserrat Jaumot, et al. Phosphorylation at Ser-181 of oncogenic KRAS is required for tumor growth. Cancer Res. 2014;74(4):1190-9. PMID: 24371225. doi: 10.1158/0008-5472.CAN-13-1750.
Carles Barceló, Julia Etchin, Marc R Mansour, Takaomi Sanda, Mireia M Ginesta, Victor J Sanchez-Arévalo Lobo, et al. Ribonucleoprotein HNRNPA2B1 interacts with and regulates oncogenic KRAS in pancreatic ductal adenocarcinoma cells. Gastroenterology. 2014;147(4):882-892.e8. PMID: 24998203. doi: 10.1053/j.gastro.2014.06.041.
Yoshihito Kano, Teklab Gebregiworgis, Christopher B Marshall, Nikolina Radulovich, Betty P K Poon, Jonathan St-Germain, et al. Tyrosyl phosphorylation of KRAS stalls GTPase cycle via alteration of switch I and II conformation. Nat Commun. 2019;10(1):224. PMID: 30644389. doi: 10.1038/s41467-018-08115-8.
Chiara Ambrogio, Jens Köhler, Zhi-Wei Zhou, Haiyun Wang, Raymond Paranal, Jiaqi Li, et al. KRAS Dimerization Impacts MEK Inhibitor Sensitivity and Oncogenic Activity of Mutant KRAS. Cell. 2018;172(4):857-868.e15. PMID: 29336889. doi: 10.1016/j.cell.2017.12.020.
Sigrid K Fey, Arafath K Najumudeen, Dale M Watt, Laura M Millett, Catriona A Ford, Kathryn Gilroy, et al. KRAS Loss of Heterozygosity Promotes MAPK-Dependent Pancreatic Ductal Adenocarcinoma Initiation and Induces Therapeutic Sensitivity to MEK Inhibition. Cancer Res. 2025;85(2):251-262. PMID: 39412982. doi: 10.1158/0008-5472.CAN-23-2709.
Arafath K Najumudeen, Sigrid K Fey, Laura M Millett, Catriona A Ford, Kathryn Gilroy, Nuray Gunduz, et al. KRAS allelic imbalance drives tumour initiation yet suppresses metastasis in colorectal cancer in vivo. Nat Commun. 2024;15(1):100. PMID: 38168062. doi: 10.1038/s41467-023-44342-4.
Johanna Lilja, Jasmin Kaivola, James R W Conway, Joni Vuorio, Hanna Parkkola, Pekka Roivas, et al. SHANK3 depletion leads to ERK signalling overdose and cell death in KRAS-mutant cancers. Nat Commun. 2024;15(1):8002. PMID: 39266533. doi: 10.1038/s41467-024-52326-1.
Sabrina Arena, Claudio Isella, Miriam Martini, Ario de Marco, Enzo Medico, Alberto Bardelli Knock-in of oncogenic Kras does not transform mouse somatic cells but triggers a transcriptional response that classifies human cancers. Cancer Res. 2007;67(18):8468-76. PMID: 17875685.
Ying Feng, Guido T Bommer, Jenny Zhao, Maranne Green, Evan Sands, Yali Zhai, et al. Mutant KRAS promotes hyperplasia and alters differentiation in the colon epithelium but does not expand the presumptive stem cell pool. Gastroenterology. 2011;141(3):1003-1013.e1-10. PMID: 21699772. doi: 10.1053/j.gastro.2011.05.007.
Shon Green, Christy L Trejo, Martin McMahon PIK3CA(H1047R) Accelerates and Enhances KRAS(G12D)-Driven Lung Tumorigenesis. Cancer Res. 2015;75(24):5378-91. PMID: 26567140. doi: 10.1158/0008-5472.CAN-15-1249.
Jianhua Ling, Ya'an Kang, Ruiying Zhao, Qianghua Xia, Dung-Fang Lee, Zhe Chang, et al. KrasG12D-induced IKK2/β/NF-κB activation by IL-1α and p62 feedforward loops is required for development of pancreatic ductal adenocarcinoma. Cancer Cell. 2012;21(1):105-20. PMID: 22264792. doi: 10.1016/j.ccr.2011.12.006.
Sheri L Holmen, Bart O Williams Essential role for Ras signaling in glioblastoma maintenance. Cancer Res. 2005;65(18):8250-5. PMID: 16166301.
Haoqiang Ying, Alec C Kimmelman, Costas A Lyssiotis, Sujun Hua, Gerald C Chu, Eliot Fletcher-Sananikone, et al. Oncogenic Kras maintains pancreatic tumors through regulation of anabolic glucose metabolism. Cell. 2012;149(3):656-70. PMID: 22541435. doi: 10.1016/j.cell.2012.01.058.
Mandar Deepak Muzumdar, Pan-Yu Chen, Kimberly Judith Dorans, Katherine Minjee Chung, Arjun Bhutkar, Erin Hong, et al. Survival of pancreatic cancer cells lacking KRAS function. Nat Commun. 2017;8(1):1090. PMID: 29061961. doi: 10.1038/s41467-017-00942-5.
Diane D Shao, Wen Xue, Elsa B Krall, Arjun Bhutkar, Federica Piccioni, Xiaoxing Wang, et al. KRAS and YAP1 converge to regulate EMT and tumor survival. Cell. 2014;158(1):171-84. PMID: 24954536. doi: 10.1016/j.cell.2014.06.004.
Avnish Kapoor, Wantong Yao, Haoqiang Ying, Sujun Hua, Alison Liewen, Qiuyun Wang, et al. Yap1 activation enables bypass of oncogenic Kras addiction in pancreatic cancer. Cell. 2014;158(1):185-197. PMID: 24954535. doi: 10.1016/j.cell.2014.06.003.
Adrian Vallejo, Naiara Perurena, Elisabet Guruceaga, Pawel K Mazur, Susana Martinez-Canarias, Carolina Zandueta, et al. An integrative approach unveils FOSL1 as an oncogene vulnerability in KRAS-driven lung and pancreatic cancer. Nat Commun. 2017;8:14294. PMID: 28220783. doi: 10.1038/ncomms14294.
Nathan T Ihle, Lauren A Byers, Edward S Kim, Pierre Saintigny, J Jack Lee, George R Blumenschein, et al. Effect of KRAS oncogene substitutions on protein behavior: implications for signaling and clinical outcome. J Natl Cancer Inst. 2012;104(3):228-39. PMID: 22247021. doi: 10.1093/jnci/djr523.
Emma M Kerr, Edoardo Gaude, Frances K Turrell, Christian Frezza, Carla P Martins Mutant Kras copy number defines metabolic reprogramming and therapeutic susceptibilities. Nature. 2016;531(7592):110-3. PMID: 26909577. doi: 10.1038/nature16967.
G Kong, Y-I Chang, A Damnernsawad, X You, J Du, E A Ranheim, et al. Loss of wild-type Kras promotes activation of all Ras isoforms in oncogenic Kras-induced leukemogenesis. Leukemia. 2016;30(7):1542-51. PMID: 27055865. doi: 10.1038/leu.2016.40.
Naiara Santana-Codina, Anjali A Roeth, Yi Zhang, Annan Yang, Oksana Mashadova, John M Asara, et al. Oncogenic KRAS supports pancreatic cancer through regulation of nucleotide synthesis. Nat Commun. 2018;9(1):4945. PMID: 30470748. doi: 10.1038/s41467-018-07472-8.
Arvin M Gouw, Livia S Eberlin, Katherine Margulis, Delaney K Sullivan, Georgia G Toal, Ling Tong, et al. Oncogene KRAS activates fatty acid synthase, resulting in specific ERK and lipid signatures associated with lung adenocarcinoma. Proc Natl Acad Sci U S A. 2017;114(17):4300-4305. PMID: 28400509. doi: 10.1073/pnas.1617709114.
Xiyue Hu, Ranjit Singh Atwal, Sophie Xiao, Sharif U Ahmed, Zongjie Wang, Chenlin Zhao, et al. ELOVL6 activity attenuation induces mutant KRAS degradation. Nat Chem Biol. 2025. PMID: 40954224. doi: 10.1038/s41589-025-01998-x.
Léo Aubert, Neethi Nandagopal, Zachary Steinhart, Geneviève Lavoie, Sami Nourreddine, Jacob Berman, et al. Copper bioavailability is a KRAS-specific vulnerability in colorectal cancer. Nat Commun. 2020;11(1):3701. PMID: 32709883. doi: 10.1038/s41467-020-17549-y.
Chi Chun Wong, Jian-Lin Wu, Fenfen Ji, Wei Kang, Xiqing Bian, Huarong Chen, et al. The cholesterol uptake regulator PCSK9 promotes and is a therapeutic target in APC/KRAS-mutant colorectal cancer. Nat Commun. 2022;13(1):3971. PMID: 35803966. doi: 10.1038/s41467-022-31663-z.
Huanhuan Hu, Rongjie Cheng, Yanbo Wang, Xiaojun Wang, Jianzhuang Wu, Yan Kong, et al. Oncogenic KRAS signaling drives evasion of innate immune surveillance in lung adenocarcinoma by activating CD47. J Clin Invest. 2023;133(2). PMID: 36413402. doi: 10.1172/JCI153470.
Jesse Boumelha, Andrea de Castro, Nourdine Bah, Hongui Cha, Sophie de Carné Trécesson, Sareena Rana, et al. CRISPR-Cas9 Screening Identifies KRAS-Induced COX2 as a Driver of Immunotherapy Resistance in Lung Cancer. Cancer Res. 2024;84(14):2231-2246. PMID: 38635884. doi: 10.1158/0008-5472.CAN-23-2627.
Lu Huang, John Carney, Diana M Cardona, Christopher M Counter Decreased tumorigenesis in mice with a Kras point mutation at C118. Nat Commun. 2014;5:5410. PMID: 25394415. doi: 10.1038/ncomms6410.
Hema Adhikari, Christopher M Counter Interrogating the protein interactomes of RAS isoforms identifies PIP5K1A as a KRAS-specific vulnerability. Nat Commun. 2018;9(1):3646. PMID: 30194290. doi: 10.1038/s41467-018-05692-6.
Vesna Vetma, Ilaria Puoti, Natalia K Karolak, Sohini Chakraborti, Emelyne Diers, Enrico Girardi, et al. Identification of a Highly Cooperative PROTAC Degrader Targeting GTP-Loaded KRAS(On) Alleles. J Am Chem Soc. 2025;147(45):41367-41378. PMID: 41166656. doi: 10.1021/jacs.5c10354.
Juanjuan Feng, Xuanzheng Xiao, Xinting Xia, Jian Min, Weiying Tang, Xinyi Shi, et al. A pan-KRAS inhibitor and its derived degrader elicit multifaceted anti-tumor efficacy in KRAS-driven cancers. Cancer Cell. 2025;43(10):1866-1884.e12. PMID: 40780213. doi: 10.1016/j.ccell.2025.07.006.
Johannes Popow, William Farnaby, Andreas Gollner, Christiane Kofink, Gerhard Fischer, Melanie Wurm, et al. Targeting cancer with small-molecule pan-KRAS degraders. Science. 2024;385(6715):1338-1347. PMID: 39298590. doi: 10.1126/science.adm8684.
David A Barbie, Pablo Tamayo, Jesse S Boehm, So Young Kim, Susan E Moody, Ian F Dunn, et al. Systematic RNA interference reveals that oncogenic KRAS-driven cancers require TBK1. Nature. 2009;462(7269):108-12. PMID: 19847166. doi: 10.1038/nature08460.
Lina Yang, Yunjiao Zhou, Yinghua Li, Juan Zhou, Yougen Wu, Yunqing Cui, et al. Mutations of p53 and KRAS activate NF-κB to promote chemoresistance and tumorigenesis via dysregulation of cell cycle and suppression of apoptosis in lung cancer cells. Cancer Lett. 2015;357(2):520-6. PMID: 25499080. doi: 10.1016/j.canlet.2014.12.003.
Antonia Marazioti, Ioannis Lilis, Malamati Vreka, Hara Apostolopoulou, Argyro Kalogeropoulou, Ioanna Giopanou, et al. Myeloid-derived interleukin-1β drives oncogenic KRAS-NF-κΒ addiction in malignant pleural effusion. Nat Commun. 2018;9(1):672. PMID: 29445180. doi: 10.1038/s41467-018-03051-z.
Zehua Zhu, Amir R Aref, Travis J Cohoon, Thanh U Barbie, Yu Imamura, Shenghong Yang, et al. Inhibition of KRAS-driven tumorigenicity by interruption of an autocrine cytokine circuit. Cancer Discov. 2014;4(4):452-65. PMID: 24444711. doi: 10.1158/2159-8290.CD-13-0646.
Marie Wislez, Nobukazu Fujimoto, Julie G Izzo, Amy E Hanna, Dianna D Cody, Robert R Langley, et al. High expression of ligands for chemokine receptor CXCR2 in alveolar epithelial neoplasia induced by oncogenic kras. Cancer Res. 2006;66(8):4198-207. PMID: 16618742.
Kyoung Eun Lee, Dafna Bar-Sagi Oncogenic KRas suppresses inflammation-associated senescence of pancreatic ductal cells. Cancer Cell. 2010;18(5):448-58. PMID: 21075310. doi: 10.1016/j.ccr.2010.10.020.
Yong Li, Yi He, Junya Peng, Zhendong Su, Zeyao Li, Bingjie Zhang, et al. Mutant Kras co-opts a proto-oncogenic enhancer network in inflammation-induced metaplastic progenitor cells to initiate pancreatic cancer. Nat Cancer. 2021;2(1):49-65. PMID: 35121887. doi: 10.1038/s43018-020-00134-z.
Christopher J Tape, Stephanie Ling, Maria Dimitriadi, Kelly M McMahon, Jonathan D Worboys, Hui Sun Leong, et al. Oncogenic KRAS Regulates Tumor Cell Signaling via Stromal Reciprocation. Cell. 2016;165(4):910-20. PMID: 27087446. doi: 10.1016/j.cell.2016.03.029.
Wen-Hao Hsu, Kyle A LaBella, Yiyun Lin, Ping Xu, Rumi Lee, Cheng-En Hsieh, et al. Oncogenic KRAS Drives Lipofibrogenesis to Promote Angiogenesis and Colon Cancer Progression. Cancer Discov. 2023;13(12):2652-2673. PMID: 37768068. doi: 10.1158/2159-8290.CD-22-1467.
Aaron L Moye, Antonella Fm Dost, Robert Ietswaart, Shreoshi Sengupta, VanNashlee Ya, Chrystal Aluya, et al. Early-stage lung cancer is driven by a transitional cell state dependent on a KRAS-ITGA3-SRC axis. EMBO J. 2024;43(14):2843-2861. PMID: 38755258. doi: 10.1038/s44318-024-00113-5.
Chengming Liu, Sufei Zheng, Zhanyu Wang, Sihui Wang, Xinfeng Wang, Lu Yang, et al. KRAS-G12D mutation drives immune suppression and the primary resistance of anti-PD-1/PD-L1 immunotherapy in non-small cell lung cancer. Cancer Commun (Lond). 2022;42(9):828-847. PMID: 35811500. doi: 10.1002/cac2.12327.
Huashan Liu, Zhenxing Liang, Chi Zhou, Ziwei Zeng, Fengwei Wang, Tuo Hu, et al. Mutant KRAS triggers functional reprogramming of tumor-associated macrophages in colorectal cancer. Signal Transduct Target Ther. 2021;6(1):144. PMID: 33833221. doi: 10.1038/s41392-021-00534-2.
Chi Zhou, Wenxin Li, Zhenxing Liang, Xianrui Wu, Sijing Cheng, Jianhong Peng, et al. Mutant KRAS-activated circATXN7 fosters tumor immunoescape by sensitizing tumor-specific T cells to activation-induced cell death. Nat Commun. 2024;15(1):499. PMID: 38216551. doi: 10.1038/s41467-024-44779-1.
Eusebio Manchado, Susann Weissmueller, John P Morris, Chi-Chao Chen, Ramona Wullenkord, Amaia Lujambio, et al. A combinatorial strategy for treating KRAS-mutant lung cancer. Nature. 2016;534(7609):647-51. PMID: 27338794.
Kenji Morimoto, Tadaaki Yamada, Soichi Hirai, Yuki Katayama, Sarina Fukui, Ryo Sawada, et al. AXL signal mediates adaptive resistance to KRAS G12C inhibitors in KRAS G12C-mutant tumor cells. Cancer Lett. 2024;587:216692. PMID: 38342232. doi: 10.1016/j.canlet.2024.216692.
Pingping Hou, Avnish Kapoor, Qiang Zhang, Jiexi Li, Chang-Jiun Wu, Jun Li, et al. Tumor Microenvironment Remodeling Enables Bypass of Oncogenic KRAS Dependency in Pancreatic Cancer. Cancer Discov. 2020;10(7):1058-1077. PMID: 32341020. doi: 10.1158/2159-8290.CD-19-0597.
Baoyuan Zhang, Yan Zhang, Jiangwei Zhang, Ping Liu, Bo Jiao, Zaiqi Wang, et al. Focal Adhesion Kinase (FAK) Inhibition Synergizes with KRAS G12C Inhibitors in Treating Cancer through the Regulation of the FAK-YAP Signaling. Adv Sci (Weinh). 2021;8(16):e2100250. PMID: 34151545. doi: 10.1002/advs.202100250.
Xinyuan Liu, Jiaqi Yang, Sicong Huang, Yifan Hong, Yupeng Zhu, Jianing Wang, et al. Pancreatic cancer-derived extracellular vesicles enhance chemoresistance by delivering KRAS. Mol Ther. 2025;33(3):1134-1153. PMID: 39810420. doi: 10.1016/j.ymthe.2025.01.023.
Patrícia Dias Carvalho, Carlos F Guimarães, Ana P Cardoso, Susana Mendonça, Ângela M Costa, Maria J Oliveira, et al. KRAS Oncogenic Signaling Extends beyond Cancer Cells to Orchestrate the Microenvironment. Cancer Res. 2018;78(1):7-14. PMID: 29263151. doi: 10.1158/0008-5472.CAN-17-2084.
Shaima'a Hamarsheh, Olaf Groß, Tilman Brummer, Robert Zeiser Immune modulatory effects of oncogenic KRAS in cancer. Nat Commun. 2020;11(1):5439. PMID: 33116132. doi: 10.1038/s41467-020-19288-6.
Bincy Philip, Christina L Roland, Jaroslaw Daniluk, Yan Liu, Deyali Chatterjee, Sobeyda B Gomez, et al. A high-fat diet activates oncogenic Kras and COX2 to induce development of pancreatic ductal adenocarcinoma in mice. Gastroenterology. 2013;145(6):1449-58. PMID: 23958541. doi: 10.1053/j.gastro.2013.08.018.
Huseyin Baloglu, Nuri Yigit Stromal cells do not share KRAS mutations that the epithelial component harbors in colorectal carcinomas. Cancer Lett. 2011;308(1):118-21. PMID: 21624768. doi: 10.1016/j.canlet.2011.04.021.
Kevin Adrian, Matthew J Strouch, Qinghua Zeng, Morgan R Barron, Eric C Cheon, Akilesh Honasoge, et al. Tgfbr1 haploinsufficiency inhibits the development of murine mutant Kras-induced pancreatic precancer. Cancer Res. 2009;69(24):9169-74. PMID: 19951995. doi: 10.1158/0008-5472.CAN-09-1705.
Jinyu Li, Stephen D'Amico, Varvara Kirillov, Oleksi Petrenko, Nancy C Reich Oncogenic dependency plays a dominant role in the immune response to cancer. Proc Natl Acad Sci U S A. 2023;120(41):e2308635120. PMID: 37782788. doi: 10.1073/pnas.2308635120.
Irene Ischenko, Stephen D'Amico, Manisha Rao, Jinyu Li, Michael J Hayman, Scott Powers, et al. KRAS drives immune evasion in a genetic model of pancreatic cancer. Nat Commun. 2021;12(1):1482. PMID: 33674596. doi: 10.1038/s41467-021-21736-w.
Ling Xie, Chunwei Xu, Han Si, Xiaoshuang Fu, Xiangcai Yang, Jianan Jin, et al. Preclinical Characterization and Clinical Activity of RNK08954, a Highly Selective and Orally Bioavailable KRAS G12D Inhibitor. Cancer Discov. 2026. PMID: 41557983. doi: 10.1158/2159-8290.CD-25-1346.
Jing Pan, Qi Zhang, Katie Palen, Li Wang, Lifen Qiao, Bryon Johnson, et al. Potentiation of Kras peptide cancer vaccine by avasimibe, a cholesterol modulator. EBioMedicine. 2019;49:72-81. PMID: 31680003. doi: 10.1016/j.ebiom.2019.10.044.
Ander Puyalto, María Rodríguez-Remírez, Inés López, Irati Macaya, Elizabeth Guruceaga, María Olmedo, et al. Trametinib sensitizes KRAS-mutant lung adenocarcinoma tumors to PD-1/PD-L1 axis blockade via Id1 downregulation. Mol Cancer. 2024;23(1):78. PMID: 38643157. doi: 10.1186/s12943-024-01991-3.
Antonio Sacco, Cinzia Federico, Katia Todoerti, Bachisio Ziccheddu, Valentina Palermo, Arianna Giacomini, et al. Specific targeting of the KRAS mutational landscape in myeloma as a tool to unveil the elicited antitumor activity. Blood. 2021;138(18):1705-1720. PMID: 34077955. doi: 10.1182/blood.2020010572.
Rasmus Hansen, Ulf Peters, Anjali Babbar, Yuching Chen, Jun Feng, Matthew R Janes, et al. The reactivity-driven biochemical mechanism of covalent KRAS. Nat Struct Mol Biol. 2018;25(6):454-462. PMID: 29760531. doi: 10.1038/s41594-018-0061-5.
Piro Lito, Martha Solomon, Lian-Sheng Li, Rasmus Hansen, Neal Rosen Allele-specific inhibitors inactivate mutant KRAS G12C by a trapping mechanism. Science. 2016;351(6273):604-8. PMID: 26841430. doi: 10.1126/science.aad6204.
David S Hong, Marwan G Fakih, John H Strickler, Jayesh Desai, Gregory A Durm, Geoffrey I Shapiro, et al. KRAS. N Engl J Med. 2020;383(13):1207-1217. PMID: 32955176. doi: 10.1056/NEJMoa1917239.
James D Vasta, D Matthew Peacock, Qinheng Zheng, Joel A Walker, Ziyang Zhang, Chad A Zimprich, et al. KRAS is vulnerable to reversible switch-II pocket engagement in cells. Nat Chem Biol. 2022;18(6):596-604. PMID: 35314814. doi: 10.1038/s41589-022-00985-w.
Miles A Keats, John J W Han, Yeon-Hwa Lee, Chih-Shia Lee, Ji Luo A Nonconserved Histidine Residue on KRAS Drives Paralog Selectivity of the KRASG12D Inhibitor MRTX1133. Cancer Res. 2023;83(17):2816-2823. PMID: 37339170. doi: 10.1158/0008-5472.CAN-23-0592.
Teklab Gebregiworgis, Yoshihito Kano, Jonathan St-Germain, Nikolina Radulovich, Molly L Udaskin, Ahmet Mentes, et al. The Q61H mutation decouples KRAS from upstream regulation and renders cancer cells resistant to SHP2 inhibitors. Nat Commun. 2021;12(1):6274. PMID: 34725361. doi: 10.1038/s41467-021-26526-y.
Nicolas Bery, Sandrine Legg, Judit Debreczeni, Jason Breed, Kevin Embrey, Christopher Stubbs, et al. KRAS-specific inhibition using a DARPin binding to a site in the allosteric lobe. Nat Commun. 2019;10(1):2607. PMID: 31197133. doi: 10.1038/s41467-019-10419-2.
Padma Akkapeddi, Takamitsu Hattori, Imran Khan, Eliezra Glasser, Akiko Koide, Gayatri Ketavarapu, et al. Exploring switch II pocket conformation of KRAS(G12D) with mutant-selective monobody inhibitors. Proc Natl Acad Sci U S A. 2023;120(28):e2302485120. PMID: 37399416. doi: 10.1073/pnas.2302485120.
Kai Wen Teng, Steven T Tsai, Takamitsu Hattori, Carmine Fedele, Akiko Koide, Chao Yang, et al. Selective and noncovalent targeting of RAS mutants for inhibition and degradation. Nat Commun. 2021;12(1):2656. PMID: 33976200. doi: 10.1038/s41467-021-22969-5.
Nicolas Bery, Ami Miller, Terry Rabbitts A potent KRAS macromolecule degrader specifically targeting tumours with mutant KRAS. Nat Commun. 2020;11(1):3233. PMID: 32591521. doi: 10.1038/s41467-020-17022-w.
Elizaveta S Leshchiner, Andrey Parkhitko, Gregory H Bird, James Luccarelli, Joseph A Bellairs, Silvia Escudero, et al. Direct inhibition of oncogenic KRAS by hydrocarbon-stapled SOS1 helices. Proc Natl Acad Sci U S A. 2015;112(6):1761-6. PMID: 25624485. doi: 10.1073/pnas.1413185112.
Kobe Janssen, Filip Claes, Dido Van de Velde, Vanessa L Wehbi, Bert Houben, Yulia Lampi, et al. Exploiting the intrinsic misfolding propensity of the KRAS oncoprotein. Proc Natl Acad Sci U S A. 2023;120(9):e2214921120. PMID: 36812200. doi: 10.1073/pnas.2214921120.
Shady Sayed, Olga A Sidorova, Alexander Hennig, Martina Augsburg, Catherine P Cortés Vesga, Moustafa Abohawya, et al. Efficient Correction of Oncogenic KRAS and TP53 Mutations through CRISPR Base Editing. Cancer Res. 2022;82(17):3002-3015. PMID: 35802645. doi: 10.1158/0008-5472.CAN-21-2519.
Björn Kruspig, Tiziana Monteverde, Sarah Neidler, Andreas Hock, Emma Kerr, Colin Nixon, et al. The ERBB network facilitates KRAS-driven lung tumorigenesis. Sci Transl Med. 2018;10(446). PMID: 29925636. doi: 10.1126/scitranslmed.aao2565.
Claudia Tonelli, Astrid Deschênes, Victoria A Gaeth, Amanda Jensen, Nandan Vithlani, Melissa A Yao, et al. FGFR2 Abrogation Intercepts Pancreatic Ductal Adenocarcinoma Development. Cancer Res. 2025;85(11):1960-1977. PMID: 40170241. doi: 10.1158/0008-5472.CAN-24-4576.
Ryan B Corcoran, Katherine A Cheng, Aaron N Hata, Anthony C Faber, Hiromichi Ebi, Erin M Coffee, et al. Synthetic lethal interaction of combined BCL-XL and MEK inhibition promotes tumor regressions in KRAS mutant cancer models. Cancer Cell. 2013;23(1):121-8. PMID: 23245996. doi: 10.1016/j.ccr.2012.11.007.
Miriam Molina-Arcas, Christopher Moore, Sareena Rana, Febe van Maldegem, Edurne Mugarza, Pablo Romero-Clavijo, et al. Development of combination therapies to maximize the impact of KRAS-G12C inhibitors in lung cancer. Sci Transl Med. 2019;11(510). PMID: 31534020. doi: 10.1126/scitranslmed.aaw7999.
Ensar Halilovic, Qing-Bai She, Qing Ye, Raymond Pagliarini, William R Sellers, David B Solit, et al. PIK3CA mutation uncouples tumor growth and cyclin D1 regulation from MEK/ERK and mutant KRAS signaling. Cancer Res. 2010;70(17):6804-14. PMID: 20699365. doi: 10.1158/0008-5472.CAN-10-0409.
Jieqiong Wang, Kewen Hu, Jiawei Guo, Feixiong Cheng, Jing Lv, Wenhao Jiang, et al. Suppression of KRas-mutant cancer through the combined inhibition of KRAS with PLK1 and ROCK. Nat Commun. 2016;7:11363. PMID: 27193833. doi: 10.1038/ncomms11363.
Felix Dietlein, Bastian Kalb, Mladen Jokic, Elisa M Noll, Alexander Strong, Lars Tharun, et al. A Synergistic Interaction between Chk1- and MK2 Inhibitors in KRAS-Mutant Cancer. Cell. 2015;162(1):146-59. PMID: 26140595. doi: 10.1016/j.cell.2015.05.053.
Anurag Singh, Michael F Sweeney, Min Yu, Alexa Burger, Patricia Greninger, Cyril Benes, et al. TAK1 inhibition promotes apoptosis in KRAS-dependent colon cancers. Cell. 2012;148(4):639-50. PMID: 22341439. doi: 10.1016/j.cell.2011.12.033.
Aslamuzzaman Kazi, Shengyan Xiang, Hua Yang, Daniel Delitto, José Trevino, Rays H Y Jiang, et al. GSK3 suppression upregulates β-catenin and c-Myc to abrogate KRas-dependent tumors. Nat Commun. 2018;9(1):5154. PMID: 30514931. doi: 10.1038/s41467-018-07644-6.
Kun Li, Yanan Liu, Yi Ding, Zhengwei Zhang, Juanjuan Feng, Jiaxin Hu, et al. BCL6 is regulated by the MAPK/ELK1 axis and promotes KRAS-driven lung cancer. J Clin Invest. 2022;132(22). PMID: 36377663. doi: 10.1172/JCI161308.
Bing Yu, Stephen Swatkoski, Alesia Holly, Liam C Lee, Valentin Giroux, Chih-Shia Lee, et al. Oncogenesis driven by the Ras/Raf pathway requires the SUMO E2 ligase Ubc9. Proc Natl Acad Sci U S A. 2015;112(14):E1724-33. PMID: 25805818. doi: 10.1073/pnas.1415569112.
Kaja Kostyrko, Marta Román, Alex G Lee, David R Simpson, Phuong T Dinh, Stanley G Leung, et al. UHRF1 is a mediator of KRAS driven oncogenesis in lung adenocarcinoma. Nat Commun. 2023;14(1):3966. PMID: 37407562. doi: 10.1038/s41467-023-39591-2.
Carol Babij, Yihong Zhang, Robert J Kurzeja, Anke Munzli, Amro Shehabeldin, Manory Fernando, et al. STK33 kinase activity is nonessential in KRAS-dependent cancer cells. Cancer Res. 2011;71(17):5818-26. PMID: 21742770. doi: 10.1158/0008-5472.CAN-11-0778.
Tuoping Luo, Kristina Masson, Jacob D Jaffe, Whitney Silkworth, Nathan T Ross, Christina A Scherer, et al. STK33 kinase inhibitor BRD-8899 has no effect on KRAS-dependent cancer cell viability. Proc Natl Acad Sci U S A. 2012;109(8):2860-5. PMID: 22323609. doi: 10.1073/pnas.1120589109.
Edwin H Yau, Indrasena Reddy Kummetha, Gianluigi Lichinchi, Rachel Tang, Yunlin Zhang, Tariq M Rana Genome-Wide CRISPR Screen for Essential Cell Growth Mediators in Mutant KRAS Colorectal Cancers. Cancer Res. 2017;77(22):6330-6339. PMID: 28954733. doi: 10.1158/0008-5472.CAN-17-2043.
Arafath K Najumudeen, Fatih Ceteci, Sigrid K Fey, Gregory Hamm, Rory T Steven, Holly Hall, et al. The amino acid transporter SLC7A5 is required for efficient growth of KRAS-mutant colorectal cancer. Nat Genet. 2021;53(1):16-26. PMID: 33414552. doi: 10.1038/s41588-020-00753-3.
Kewen Hu, Kun Li, Jing Lv, Jie Feng, Jing Chen, Haigang Wu, et al. Suppression of the SLC7A11/glutathione axis causes synthetic lethality in KRAS-mutant lung adenocarcinoma. J Clin Invest. 2020;130(4):1752-1766. PMID: 31874110. doi: 10.1172/JCI124049.
Dana M Gwinn, Alex G Lee, Marcela Briones-Martin-Del-Campo, Crystal S Conn, David R Simpson, Anna I Scott, et al. Oncogenic KRAS Regulates Amino Acid Homeostasis and Asparagine Biosynthesis via ATF4 and Alters Sensitivity to L-Asparaginase. Cancer Cell. 2018;33(1):91-107.e6. PMID: 29316436. doi: 10.1016/j.ccell.2017.12.003.
Jihye Yun, Edouard Mullarky, Changyuan Lu, Kaitlyn N Bosch, Adam Kavalier, Keith Rivera, et al. Vitamin C selectively kills KRAS and BRAF mutant colorectal cancer cells by targeting GAPDH. Science. 2015;350(6266):1391-6. PMID: 26541605. doi: 10.1126/science.aaa5004.
Yunyi Bian, Guangyao Shan, Guoshu Bi, Zhijie Xu, Jiaqi Liang, Yuanliang Yan, et al. Targeting polyamine metabolism and ferroptosis enhances the efficacy of KRAS-targeted therapy depending on KEAP1 status. Nat Commun. 2025;16(1):9923. PMID: 41219185. doi: 10.1038/s41467-025-65441-4.
Guang Lei, Boyi Gan Iron out KRAS-driven cancer. J Exp Med. 2022;219(4). PMID: 35262627. doi: 10.1084/jem.20212166.
Walaa E Kattan, Junchen Liu, Dina Montufar-Solis, Hong Liang, Bhargavi Brahmendra Barathi, Ransome van der Hoeven, et al. Components of the phosphatidylserine endoplasmic reticulum to plasma membrane transport mechanism as targets for KRAS inhibition in pancreatic cancer. Proc Natl Acad Sci U S A. 2021;118(51). PMID: 34903667. doi: 10.1073/pnas.2114126118.
Juan Li, Yang Wang, Yue Luo, Yang Liu, Yong Yi, Jinsong Li, et al. USP5-Beclin 1 axis overrides p53-dependent senescence and drives Kras-induced tumorigenicity. Nat Commun. 2022;13(1):7799. PMID: 36528652. doi: 10.1038/s41467-022-35557-y.
Mandayam O Nandan, Beth B McConnell, Amr M Ghaleb, Agnieszka B Bialkowska, Hongmiao Sheng, Jinyi Shao, et al. Krüppel-like factor 5 mediates cellular transformation during oncogenic KRAS-induced intestinal tumorigenesis. Gastroenterology. 2008;134(1):120-30. PMID: 18054006.
Warapen Treekitkarnmongkol, Hiroshi Katayama, Deivendran Sankaran, Mei-Chee Tai, Sanchita Rauth, Hanxiao Chen, et al. RASH3D19 mediates RAS activation through a positive feedback loop in KRAS-mutant cancer. Nat Cell Biol. 2026;28(1):197-206. PMID: 41326795. doi: 10.1038/s41556-025-01816-5.
Marina Salmón, Ruth Álvarez-Díaz, Coral Fustero-Torre, Oksana Brehey, Carmen G Lechuga, Manuel Sanclemente, et al. Kras oncogene ablation prevents resistance in advanced lung adenocarcinomas. J Clin Invest. 2023;133(7). PMID: 36928090. doi: 10.1172/JCI164413.
Anna M Varghese, Maria A Perry, Joanne F Chou, Subhiksha Nandakumar, Daniel Muldoon, Amanda Erakky, et al. Clinicogenomic landscape of pancreatic adenocarcinoma identifies KRAS mutant dosage as prognostic of overall survival. Nat Med. 2025;31(2):466-477. PMID: 39753968. doi: 10.1038/s41591-024-03362-3.
Chunxiao Zhu, Xiaoqing Guan, Xinuo Zhang, Xin Luan, Zhengbo Song, Xiangdong Cheng, et al. Targeting KRAS mutant cancers: from druggable therapy to drug resistance. Mol Cancer. 2022;21(1):159. PMID: 35922812. doi: 10.1186/s12943-022-01629-2.
Hiromichi Ebi, Ryan B Corcoran, Anurag Singh, Zhao Chen, Youngchul Song, Eugene Lifshits, et al. Receptor tyrosine kinases exert dominant control over PI3K signaling in human KRAS mutant colorectal cancers. J Clin Invest. 2011;121(11):4311-21. PMID: 21985784. doi: 10.1172/JCI57909.
Yihsuan S Tsai, Mark G Woodcock, Salma H Azam, Leigh B Thorne, Krishna L Kanchi, Joel S Parker, et al. Rapid idiosyncratic mechanisms of clinical resistance to KRAS G12C inhibition. J Clin Invest. 2022;132(4). PMID: 34990404. doi: 10.1172/JCI155523.
Tadashi Manabe, Trever G Bivona Remodeling of the tumor/tumor microenvironment ecosystem during KRAS G12C inhibitor clinical resistance in lung cancer. J Clin Invest. 2022;132(4). PMID: 35166243. doi: 10.1172/JCI156891.
Federica Di Nicolantonio, Sabrina Arena, Josep Tabernero, Stefano Grosso, Francesca Molinari, Teresa Macarulla, et al. Deregulation of the PI3K and KRAS signaling pathways in human cancer cells determines their response to everolimus. J Clin Invest. 2010;120(8):2858-66. PMID: 20664172. doi: 10.1172/JCI37539.
Grace M Wang, Hong Yuen Wong, Hiroyuki Konishi, Brian G Blair, Abde M Abukhdeir, John P Gustin, et al. Single copies of mutant KRAS and mutant PIK3CA cooperate in immortalized human epithelial cells to induce tumor formation. Cancer Res. 2013;73(11):3248-61. PMID: 23580570. doi: 10.1158/0008-5472.CAN-12-1578.
W J McDaid, L Wilson, H Adderley, A Martinez-Lopez, M J Baker, J Searle, et al. The PI3K-AKT-mTOR axis persists as a therapeutic dependency in KRAS. Mol Cancer. 2024;23(1):253. PMID: 39533328. doi: 10.1186/s12943-024-02157-x.
Mounira Chalabi-Dchar, Stéphanie Cassant-Sourdy, Camille Duluc, Marjorie Fanjul, Hubert Lulka, Rémi Samain, et al. Loss of Somatostatin Receptor Subtype 2 Promotes Growth of KRAS-Induced Pancreatic Tumors in Mice by Activating PI3K Signaling and Overexpression of CXCL16. Gastroenterology. 2015;148(7):1452-65. PMID: 25683115. doi: 10.1053/j.gastro.2015.02.009.
Madelaine S Theardy, Mitsunobu Takeda, Alexey Sorokin, Shuaitong Chen, Zecheng Yang, Xiaofei Wang, et al. Therapeutic targeting of syndecan-1 axis overcomes acquired resistance to KRAS-targeted therapy in gastrointestinal cancers. Cell Rep Med. 2025;6(8):102253. PMID: 40713971. doi: 10.1016/j.xcrm.2025.102253.
O Alejandro Balbin, John R Prensner, Anirban Sahu, Anastasia Yocum, Sunita Shankar, Rohit Malik, et al. Reconstructing targetable pathways in lung cancer by integrating diverse omics data. Nat Commun. 2013;4:2617. PMID: 24135919. doi: 10.1038/ncomms3617.
Ken Fujimura, Huawei Wang, Felicia Watson, Richard L Klemke KRAS Oncoprotein Expression Is Regulated by a Self-Governing eIF5A-PEAK1 Feed-Forward Regulatory Loop. Cancer Res. 2018;78(6):1444-1456. PMID: 29321164. doi: 10.1158/0008-5472.CAN-17-2873.
Yafang Wang, Mingyue Yao, Cheng Li, Kexin Yang, Xiaolong Qin, Lansong Xu, et al. Targeting ST8SIA6-AS1 counteracts KRAS. Exp Hematol Oncol. 2023;12(1):105. PMID: 38104151. doi: 10.1186/s40164-023-00466-3.
Shasha Tao, Shue Wang, Seyed Javad Moghaddam, Aikseng Ooi, Eli Chapman, Pak K Wong, et al. Oncogenic KRAS confers chemoresistance by upregulating NRF2. Cancer Res. 2014;74(24):7430-41. PMID: 25339352. doi: 10.1158/0008-5472.CAN-14-1439.
Yuta Adachi, Kentaro Ito, Yuko Hayashi, Ryo Kimura, Tuan Zea Tan, Rui Yamaguchi, et al. Epithelial-to-Mesenchymal Transition is a Cause of Both Intrinsic and Acquired Resistance to KRAS G12C Inhibitor in KRAS G12C-Mutant Non-Small Cell Lung Cancer. Clin Cancer Res. 2020;26(22):5962-5973. PMID: 32900796. doi: 10.1158/1078-0432.CCR-20-2077.
Zhuxuan Li, Xueqian Zhuang, Chun-Hao Pan, Yan Yan, Rohit Thummalapalli, Jill Hallin, et al. Alveolar Differentiation Drives Resistance to KRAS Inhibition in Lung Adenocarcinoma. Cancer Discov. 2024;14(2):308-325. PMID: 37931288. doi: 10.1158/2159-8290.CD-23-0289.
Anupriya Singhal, Hannah C Styers, Jonathan Rub, Zhuxuan Li, Stefan R Torborg, Jung Yun Kim, et al. A Classical Epithelial State Drives Acute Resistance to KRAS Inhibition in Pancreatic Cancer. Cancer Discov. 2024;14(11):2122-2134. PMID: 38975873. doi: 10.1158/2159-8290.CD-24-0740.
Nathan L Tran, Jiewei Jiang, Min Ma, Gillian E Gadbois, Kevin C M Gulay, Alyssa L Verano, et al. ZBTB11 depletion targets metabolic vulnerabilities in KRAS inhibitor-resistant PDAC. Nat Chem Biol. 2026;22(2):260-273. PMID: 40789946. doi: 10.1038/s41589-025-01978-1.
Kirsten L Bryant, Clint A Stalnecker, Daniel Zeitouni, Jennifer E Klomp, Sen Peng, Andrey P Tikunov, et al. Combination of ERK and autophagy inhibition as a treatment approach for pancreatic cancer. Nat Med. 2019;25(4):628-640. PMID: 30833752. doi: 10.1038/s41591-019-0368-8.
Gamze Bildik, Joshua P Gray, Weiqun Mao, Hailing Yang, Rumeysa Ozyurt, Vivian R Orellana, et al. DIRAS3 induces autophagy and enhances sensitivity to anti-autophagic therapy in KRAS-driven pancreatic and ovarian carcinomas. Autophagy. 2024;20(3):675-691. PMID: 38169324. doi: 10.1080/15548627.2023.2299516.
Yunyi Bian, Guangyao Shan, Guoshu Bi, Jiaqi Liang, Zhengyang Hu, Qihai Sui, et al. Targeting ALDH1A1 to enhance the efficacy of KRAS-targeted therapy through ferroptosis. Redox Biol. 2024;77:103361. PMID: 39317105. doi: 10.1016/j.redox.2024.103361.
Fabienne Müller, Jonathan K M Lim, Christina M Bebber, Eric Seidel, Sofya Tishina, Alina Dahlhaus, et al. Elevated FSP1 protects KRAS-mutated cells from ferroptosis during tumor initiation. Cell Death Differ. 2023;30(2):442-456. PMID: 36443441. doi: 10.1038/s41418-022-01096-8.
Kyaw Z Thein, Amadeo B Biter, David S Hong Therapeutics Targeting Mutant KRAS. Annu Rev Med. 2021;72:349-364. PMID: 33138715. doi: 10.1146/annurev-med-080819-033145.
Tamara Isermann, Christine Sers, Channing J Der, Bjoern Papke KRAS inhibitors: resistance drivers and combinatorial strategies. Trends Cancer. 2025;11(2):91-116. PMID: 39732595. doi: 10.1016/j.trecan.2024.11.009.
Di Wu, Chunbin Zhu, Haoqi Pan, He Xu, Jin Xu, Yang Liu, et al. Integrated screens reveal that guanine nucleotide depletion, which is irreversible via targeting IMPDH2, inhibits pancreatic cancer and potentiates KRAS inhibition. Gut. 2026. PMID: 41494804. doi: 10.1136/gutjnl-2025-336235.
Frank McCormick KRAS as a Therapeutic Target. Clin Cancer Res. 2015;21(8):1797-801. PMID: 25878360. doi: 10.1158/1078-0432.CCR-14-2662.
Rafael G Amado, Michael Wolf, Marc Peeters, Eric Van Cutsem, Salvatore Siena, Daniel J Freeman, et al. Wild-type KRAS is required for panitumumab efficacy in patients with metastatic colorectal cancer. J Clin Oncol. 2008;26(10):1626-34. PMID: 18316791. doi: 10.1200/JCO.2007.14.7116.
William Pao, Theresa Y Wang, Gregory J Riely, Vincent A Miller, Qiulu Pan, Marc Ladanyi, et al. KRAS mutations and primary resistance of lung adenocarcinomas to gefitinib or erlotinib. PLoS Med. 2005;2(1):e17. PMID: 15696205.
Patrick J Roberts, Thomas E Stinchcombe, Channing J Der, Mark A Socinski Personalized medicine in non-small-cell lung cancer: is KRAS a useful marker in selecting patients for epidermal growth factor receptor-targeted therapy?. J Clin Oncol. 2010;28(31):4769-77. PMID: 20921461. doi: 10.1200/JCO.2009.27.4365.
Patrick J Roberts, Thomas E Stinchcombe KRAS mutation: should we test for it, and does it matter?. J Clin Oncol. 2013;31(8):1112-21. PMID: 23401440. doi: 10.1200/JCO.2012.43.0454.
Shao-Lai Zhou, Hao-Yang Xin, Rong-Qi Sun, Zheng-Jun Zhou, Zhi-Qiang Hu, Chu-Bin Luo, et al. Association of KRAS Variant Subtypes With Survival and Recurrence in Patients With Surgically Treated Intrahepatic Cholangiocarcinoma. JAMA Surg. 2022;157(1):59-65. PMID: 34730772. doi: 10.1001/jamasurg.2021.5679.
R Seth, S Crook, S Ibrahem, W Fadhil, D Jackson, M Ilyas Concomitant mutations and splice variants in KRAS and BRAF demonstrate complex perturbation of the Ras/Raf signalling pathway in advanced colorectal cancer. Gut. 2009;58(9):1234-41. PMID: 19474002. doi: 10.1136/gut.2008.159137.
Javier Torres-Jiménez, Javier Baena Espinar, Helena Bote de Cabo, María Zurera Berjaga, Jorge Esteban-Villarrubia, Jon Zugazagoitia Fraile, et al. Targeting KRAS. Drugs. 2024;84(5):527-548. PMID: 38625662. doi: 10.1007/s40265-024-02030-7.
Bennett A Caughey, John H Strickler Targeting KRAS-Mutated Gastrointestinal Malignancies with Small-Molecule Inhibitors: A New Generation of Breakthrough Therapies. Drugs. 2024;84(1):27-44. PMID: 38109010. doi: 10.1007/s40265-023-01980-8.
Fergus Keane, Joanne F Chou, Henry Walch, Joshua Schoenfeld, Anupriya Singhal, Darren Cowzer, et al. Precision medicine for pancreatic cancer: characterizing the clinicogenomic landscape and outcomes of KRAS G12C-mutated disease. J Natl Cancer Inst. 2024;116(9):1429-1438. PMID: 38702822. doi: 10.1093/jnci/djae095.
Zhongwei Mao, Hongying Xiao, Panpan Shen, Yu Yang, Jing Xue, Yunyun Yang, et al. KRAS(G12D) can be targeted by potent inhibitors via formation of salt bridge. Cell Discov. 2022;8(1):5. PMID: 35075146. doi: 10.1038/s41421-021-00368-w.
Martin Indarte, Roisin Puentes, Marco Maruggi, Nathan T Ihle, Geoffrey Grandjean, Michael Scott, et al. An Inhibitor of the Pleckstrin Homology Domain of CNK1 Selectively Blocks the Growth of Mutant KRAS Cells and Tumors. Cancer Res. 2019;79(12):3100-3111. PMID: 31040156. doi: 10.1158/0008-5472.CAN-18-2372.
Erick Riquelme, Carmen Behrens, Heather Y Lin, George Simon, Vassiliki Papadimitrakopoulou, Julie Izzo, et al. Modulation of EZH2 Expression by MEK-ERK or PI3K-AKT Signaling in Lung Cancer Is Dictated by Different KRAS Oncogene Mutations. Cancer Res. 2016;76(3):675-85. PMID: 26676756. doi: 10.1158/0008-5472.CAN-15-1141.
Silvestre Vicent, Ron Chen, Leanne C Sayles, Chenwei Lin, Randal G Walker, Anna K Gillespie, et al. Wilms tumor 1 (WT1) regulates KRAS-driven oncogenesis and senescence in mouse and human models. J Clin Invest. 2010;120(11):3940-52. PMID: 20972333. doi: 10.1172/JCI44165.
Eunyoung Park, Shaun Rawson, Anna Schmoker, Byeong-Won Kim, Sehee Oh, Kangkang Song, et al. Cryo-EM structure of a RAS/RAF recruitment complex. Nat Commun. 2023;14(1):4580. PMID: 37516774. doi: 10.1038/s41467-023-40299-6.
Zhi-Wei Zhou, Chiara Ambrogio, Asim K Bera, Qing Li, Xing-Xiao Li, Lianbo Li, et al. KRAS. Cancer Res. 2020;80(17):3719-3731. PMID: 32605999. doi: 10.1158/0008-5472.CAN-20-0448.
Zhenhao Fang, Ki-Young Lee, Ku-Geng Huo, Geneviève Gasmi-Seabrook, Le Zheng, Nadeem Moghal, et al. Multivalent assembly of KRAS with the RAS-binding and cysteine-rich domains of CRAF on the membrane. Proc Natl Acad Sci U S A. 2020;117(22):12101-12108. PMID: 32414921. doi: 10.1073/pnas.1914076117.
Peter Dietrich, Andreas Koch, Valerie Fritz, Arndt Hartmann, Anja Katrin Bosserhoff, Claus Hellerbrand Wild type Kirsten rat sarcoma is a novel microRNA-622-regulated therapeutic target for hepatocellular carcinoma and contributes to sorafenib resistance. Gut. 2018;67(7):1328-1341. PMID: 29275358. doi: 10.1136/gutjnl-2017-315402.
Hiroki Ozawa, Kazuki Takahashi, Tomoki Motegi, Justine Jacobi, Atrayee Bhattarchya, Keisuke Shigeta, et al. Targeting KRAS Inhibitor-Resistant Pancreatic Cancer with an MUC1-C Antibody-Drug Conjugate. Clin Cancer Res. 2025;31(24):5246-5260. PMID: 41086055. doi: 10.1158/1078-0432.CCR-25-2333.
Xiangyan Jiang, Tao Wang, Bin Zhao, Haonan Sun, Yuman Dong, Yong Ma, et al. KRAS. Cell Rep Med. 2025;6(2):101966. PMID: 39970873. doi: 10.1016/j.xcrm.2025.101966.
Marco H Hofmann, Daniel Gerlach, Sandra Misale, Mark Petronczki, Norbert Kraut Expanding the Reach of Precision Oncology by Drugging All KRAS Mutants. Cancer Discov. 2022;12(4):924-937. PMID: 35046095. doi: 10.1158/2159-8290.CD-21-1331.
Ying Li, Junfeng Zhao, Yintao Li New exploration of KRAS. Exp Hematol Oncol. 2025;14(1):39. PMID: 40097966. doi: 10.1186/s40164-025-00637-4.
Jie Yang, Qiao-Li Wang, Guan-Nan Wang, Jia-Cong Ye, Zi-Qian Li, Jing-Yun Wang, et al. A pan-KRAS degrader for the treatment of KRAS-mutant cancers. Cell Discov. 2024;10(1):70. PMID: 38937452. doi: 10.1038/s41421-024-00699-4.
Gongmin Zhu, Lijiao Pei, Di Ye, Qiulin Tang, Huanji Xu, Feng Bi Targeting KRAS in colorectal cancer immunotherapy: rationale, challenges and future directions. Biochim Biophys Acta Rev Cancer. 2025;1880(4):189382. PMID: 40614879. doi: 10.1016/j.bbcan.2025.189382.
Zhi Zhu, Hongqi Chen, Chao Feng, Lingjuan Chen, Congrong Ma, Zuqiang Liu, et al. Specific inhibitor to KRAS. J Immunother Cancer. 2025;13(7). PMID: 40707131. doi: 10.1136/jitc-2024-010514.
Misako Nagasaka, Bindu Potugari, Alexis Nguyen, Ammar Sukari, Asfar S Azmi, Sai-Hong Ignatius Ou KRAS Inhibitors- yes but what next? Direct targeting of KRAS- vaccines, adoptive T cell therapy and beyond. Cancer Treat Rev. 2021;101:102309. PMID: 34715449. doi: 10.1016/j.ctrv.2021.102309.
Jing Zhang, Jing Wang, Yangang Liu, Harwin Sidik, Ken H Young, Harvey F Lodish, et al. Oncogenic Kras-induced leukemogeneis: hematopoietic stem cells as the initial target and lineage-specific progenitors as the potential targets for final leukemic transformation. Blood. 2009;113(6):1304-14. PMID: 19066392. doi: 10.1182/blood-2008-01-134262.
Celine Yeh, Wungki Park, Rona Yaeger KRAS. Dev Cell. 2023;58(17):1515-1516. PMID: 37699334. doi: 10.1016/j.devcel.2023.08.020.
Qinglong Ma, Wenyang Zhang, Kongming Wu, Lei Shi The roles of KRAS in cancer metabolism, tumor microenvironment and clinical therapy. Mol Cancer. 2025;24(1):14. PMID: 39806421. doi: 10.1186/s12943-024-02218-1.
Lei Shi, Peter Magee, Matteo Fassan, Sudhakar Sahoo, Hui Sun Leong, Dave Lee, et al. A KRAS-responsive long non-coding RNA controls microRNA processing. Nat Commun. 2021;12(1):2038. PMID: 33795683. doi: 10.1038/s41467-021-22337-3.
Ci Yang, Hong-Xu Li, Hu Gan, Xin Shuai, Chen Dong, Wei Wang, et al. KRAS4B oncogenic mutants promote non-small cell lung cancer progression via the interaction of deubiquitinase USP25 with RNF31. Dev Cell. 2025;60(12):1768-1783.e5. PMID: 39952242. doi: 10.1016/j.devcel.2025.01.015.
Ling-Yu Zhu, Jun-Bo Yuan, Li Zhang, Chun-Xiao He, Xiao Lin, Bin Xu, et al. Loss of MLL Induces Epigenetic Dysregulation of Rasgrf1 to Attenuate Kras-Driven Lung Tumorigenesis. Cancer Res. 2022;82(22):4153-4163. PMID: 36098964. doi: 10.1158/0008-5472.CAN-22-1475.
Chen Liang, Si Shi, Mingyang Liu, Yi Qin, Qingcai Meng, Jie Hua, et al. PIN1 Maintains Redox Balance via the c-Myc/NRF2 Axis to Counteract Kras-Induced Mitochondrial Respiratory Injury in Pancreatic Cancer Cells. Cancer Res. 2019;79(1):133-145. PMID: 30355620. doi: 10.1158/0008-5472.CAN-18-1968.
Catherine Carrière, Elliott S Seeley, Tobias Goetze, Daniel S Longnecker, Murray Korc The Nestin progenitor lineage is the compartment of origin for pancreatic intraepithelial neoplasia. Proc Natl Acad Sci U S A. 2007;104(11):4437-42. PMID: 17360542.
Gad Singer, Robert Oldt, Yoram Cohen, Brant G Wang, David Sidransky, Robert J Kurman, et al. Mutations in BRAF and KRAS characterize the development of low-grade ovarian serous carcinoma. J Natl Cancer Inst. 2003;95(6):484-6. PMID: 12644542.
Tamihiro Kamata, Susan Giblett, Catrin Pritchard KRAS. Blood. 2017;130(4):514-526. PMID: 28550040. doi: 10.1182/blood-2017-02-770149.
Per Pfeiffer, Camilla Qvortrup KRAS. Lancet Oncol. 2022;23(1):10-11. PMID: 34919826. doi: 10.1016/S1470-2045(21)00652-5.
Khalil Choucair, Hafsa Imtiaz, Md Hafiz Uddin, Misako Nagasaka, Mohammad Najeeb Al-Hallak, Philip A Philip, et al. Targeting KRAS mutations: orchestrating cancer evolution and therapeutic challenges. Signal Transduct Target Ther. 2025;10(1):385. PMID: 41309533. doi: 10.1038/s41392-025-02473-8.
Wei Ye, Xin Lu, Yue Qiao, Wen-Bin Ou Activity and resistance to KRAS. Biochim Biophys Acta Rev Cancer. 2024;1879(3):189108. PMID: 38723697. doi: 10.1016/j.bbcan.2024.189108.
Krishnan K Mahadevan, Kathleen M McAndrews, Valerie S LeBleu, Sujuan Yang, Hengyu Lyu, Bingrui Li, et al. KRAS. Cancer Cell. 2023;41(9):1606-1620.e8. PMID: 37625401. doi: 10.1016/j.ccell.2023.07.002.
Dong Hoon Shin, Jeong Yeon Jo, Minyoung Choi, Kyung-Hee Kim, Young-Ki Bae, Sang Soo Kim Oncogenic KRAS mutation confers chemoresistance by upregulating SIRT1 in non-small cell lung cancer. Exp Mol Med. 2023;55(10):2220-2237. PMID: 37779142. doi: 10.1038/s12276-023-01091-0.
Marcus Fischer, Wan-Ching Yen, Ann M Kapoun, Min Wang, Gilbert O'Young, John Lewicki, et al. Anti-DLL4 inhibits growth and reduces tumor-initiating cell frequency in colorectal tumors with oncogenic KRAS mutations. Cancer Res. 2011;71(5):1520-5. PMID: 21193546. doi: 10.1158/0008-5472.CAN-10-2817.
Patricia S Hähnel, Birgit Enders, Daniel Sasca, Wynand P Roos, Bernd Kaina, Lars Bullinger, et al. Targeting components of the alternative NHEJ pathway sensitizes KRAS mutant leukemic cells to chemotherapy. Blood. 2014;123(15):2355-66. PMID: 24505083. doi: 10.1182/blood-2013-01-477620.
Xinyu Shi, Liling Hu, Yongshi Huang, Mei-Fang Zhang, Xingfeng Ying, Xiaoyi Yuan, et al. Integrative proteomic characterization of human lung adenocarcinoma with KRAS G12 mutations reveals molecular pathogenesis. J Adv Res. 2025. PMID: 40957478. doi: 10.1016/j.jare.2025.09.014.
Peter M K Westcott, Kyle D Halliwill, Minh D To, Mamunur Rashid, Alistair G Rust, Thomas M Keane, et al. The mutational landscapes of genetic and chemical models of Kras-driven lung cancer. Nature. 2015;517(7535):489-92. PMID: 25363767. doi: 10.1038/nature13898.
Jiayao Ma, Shenao Fu, Jun Tan, Ying Han, Yihong Chen, Xiangying Deng, et al. Mechanistic Foundations of KRAS-Driven Tumor Ecosystems: Integrating Crosstalk among Immune, Metabolic, Microbial, and Stromal Microenvironment. Adv Sci (Weinh). 2025;12(30):e02714. PMID: 40485603. doi: 10.1002/advs.202502714.
Yanyan Shi, Huiling Zheng, Tianzhen Wang, Shengpu Zhou, Shiqing Zhao, Mo Li, et al. Targeting KRAS: from metabolic regulation to cancer treatment. Mol Cancer. 2025;24(1):9. PMID: 39799325. doi: 10.1186/s12943-024-02216-3.
Jihye Yun, Carlo Rago, Ian Cheong, Ray Pagliarini, Philipp Angenendt, Harith Rajagopalan, et al. Glucose deprivation contributes to the development of KRAS pathway mutations in tumor cells. Science. 2009;325(5947):1555-9. PMID: 19661383. doi: 10.1126/science.1174229.
Rodrigo Romero, Volkan I Sayin, Shawn M Davidson, Matthew R Bauer, Simranjit X Singh, Sarah E LeBoeuf, et al. Keap1 loss promotes Kras-driven lung cancer and results in dependence on glutaminolysis. Nat Med. 2017;23(11):1362-1368. PMID: 28967920. doi: 10.1038/nm.4407.
Ivana Yen, Frances Shanahan, Mark Merchant, Christine Orr, Thomas Hunsaker, Matthew Durk, et al. Pharmacological Induction of RAS-GTP Confers RAF Inhibitor Sensitivity in KRAS Mutant Tumors. Cancer Cell. 2018;34(4):611-625.e7. PMID: 30300582. doi: 10.1016/j.ccell.2018.09.002.
Antonios N Gargalionis, Kostas A Papavassiliou, Athanasios G Papavassiliou KRAS. Trends Pharmacol Sci. 2025;46(5):389-391. PMID: 40234119. doi: 10.1016/j.tips.2025.03.009.
Frances A Shepherd, Benjamin Lacas, Gwénaël Le Teuff, Pierre Hainaut, Pasi A Jänne, Jean-Pierre Pignon, et al. Pooled Analysis of the Prognostic and Predictive Effects of TP53 Comutation Status Combined With KRAS or EGFR Mutation in Early-Stage Resected Non-Small-Cell Lung Cancer in Four Trials of Adjuvant Chemotherapy. J Clin Oncol. 2017;35(18):2018-2027. PMID: 28453411. doi: 10.1200/JCO.2016.71.2893.
Nikhil P Mankuzhy, Valentina Santo, Jacob Y Shin, Tafadzwa L Chaunzwa, Daphna Y Gelblum, Abraham J Wu, et al. Impact of KRAS Mutations and Co-Alterations on Outcomes in Stage III Non-Squamous Non-Small Cell Lung Cancer Treated with Chemoradiation and Immunotherapy. J Thorac Oncol. 2026. PMID: 41619904. doi: 10.1016/j.jtho.2026.103565.
Sarah A Best, Sheryl Ding, Ariena Kersbergen, Xueyi Dong, Ji-Ying Song, Yi Xie, et al. Distinct initiating events underpin the immune and metabolic heterogeneity of KRAS-mutant lung adenocarcinoma. Nat Commun. 2019;10(1):4190. PMID: 31519898. doi: 10.1038/s41467-019-12164-y.
Daowei Yang, Xinlei Sun, Rohan Moniruzzaman, Hua Wang, Citu Citu, Zhongming Zhao, et al. Loss of p53 and SMAD4 induces adenosquamous subtype pancreatic cancer in the absence of an oncogenic KRAS mutation. Cell Rep Med. 2024;5(9):101711. PMID: 39232498. doi: 10.1016/j.xcrm.2024.101711.
Achille Aouba KRAS. Blood. 2017;130(4):391-392. PMID: 28751357. doi: 10.1182/blood-2017-06-788455.
Yingying Wang, Youping Zhang, Hao Luo, Wei Wei, Wanting Liu, Weiwei Wang, et al. Identification of USP2 as a novel target to induce degradation of KRAS in myeloma cells. Acta Pharm Sin B. 2024;14(12):5235-5248. PMID: 39807309. doi: 10.1016/j.apsb.2024.08.019.
Hao-Jun Xiong, Hong-Qiang Yu, Jie Zhang, Lei Fang, Di Wu, Xiao-Tong Lin, et al. Elevated FBXL6 activates both wild-type KRAS and mutant KRAS. Mil Med Res. 2023;10(1):68. PMID: 38124228. doi: 10.1186/s40779-023-00501-8.
Ming Yuan, Chi Zhang, Shaopeng Chen, Shubiao Ye, Huashan Liu, Haoxian Ke, et al. PDP1 promotes KRAS mutant colorectal cancer progression by serving as a scaffold for BRAF and MEK1. Cancer Lett. 2024;597:217007. PMID: 38849010. doi: 10.1016/j.canlet.2024.217007.
Xingzhao Ji, Mingqiang Liu, Tianyi Zhang, Weiying Zhang, Fuyuan Xue, Qiang Wan, et al. KRAS/PI3K axis driven GTF3C6 expression and promotes LUAD via FAK pathway. J Adv Res. 2025;70:243-254. PMID: 38685529. doi: 10.1016/j.jare.2024.04.028.
Jing Yang, Changchun Hou, Huashan Wang, Edith A Perez, Hanh Chi Do-Umehara, Huali Dong, et al. Miz1 promotes KRAS-driven lung tumorigenesis by repressing the protocadherin Pcdh10. Cancer Lett. 2023;555:216025. PMID: 36538983. doi: 10.1016/j.canlet.2022.216025.
Yiran Li, Fan Huo, Liusheng Chen, Haiqi Wang, Jianzhuang Wu, Peiwen Zhang, et al. Protein-Targeted Glycan Editing on Living Cells Disrupts KRAS Signaling. Angew Chem Int Ed Engl. 2023;62(26):e202218148. PMID: 37103924. doi: 10.1002/anie.202218148.
Beatriz Mateo-Victoriano, Govindi J Samaranayake, Sheela Pokharel, Gracy Jenifer Sahayanathan, Christina Jayaraj, Clara I Troccoli, et al. Oncogenic KRAS addiction states differentially influence MTH1 expression and 8-oxodGTPase activity in lung adenocarcinoma. Redox Biol. 2025;82:103610. PMID: 40184641. doi: 10.1016/j.redox.2025.103610.
Man Li, Xuexin Yu, Yuanji Liu, Shuqin Ouyang, Long Wu, Xiaohong Chen, et al. KRAS/ABHD17C/ALOX15B Axis Promotes Pancreatic Cancer Progression via Ferroptosis Evasion. Adv Sci (Weinh). 2025;12(35):e04470. PMID: 40569151. doi: 10.1002/advs.202504470.
Soo-Yeon Lee, Hyun-Jong Eun, Ki-Young Lee Effector Binding Sequentially Alters KRAS Dimerization on the Membrane: New Insights Into RAS-Mediated RAF Activation. Adv Sci (Weinh). 2024;11(38):e2401530. PMID: 39138901. doi: 10.1002/advs.202401530.
Ken Takezawa, Isamu Okamoto, Kimio Yonesaka, Erina Hatashita, Yuki Yamada, Masahiro Fukuoka, et al. Sorafenib inhibits non-small cell lung cancer cell growth by targeting B-RAF in KRAS wild-type cells and C-RAF in KRAS mutant cells. Cancer Res. 2009;69(16):6515-21. PMID: 19638574. doi: 10.1158/0008-5472.CAN-09-1076.
Niels Smakman, Diana J M van den Wollenberg, Sjoerd G Elias, Takehiko Sasazuki, Senji Shirasawa, Rob C Hoeben, et al. KRAS(D13) Promotes apoptosis of human colorectal tumor cells by ReovirusT3D and oxaliplatin but not by tumor necrosis factor-related apoptosis-inducing ligand. Cancer Res. 2006;66(10):5403-8. PMID: 16707468.
Young Seok Cho, Hanhee Cho, Ha Rin Kim, Seong Jin Park, Joo Hye Yeo, Yoon Gun Ko, et al. Macropinocytosis-targeted peptide-docetaxel conjugate for bystander pancreatic cancer treatment. J Control Release. 2024;376:829-841. PMID: 39491626. doi: 10.1016/j.jconrel.2024.10.070.
Sophie M Ernst, Ronald van Marion, Peggy N Atmodimedjo, Evert de Jonge, Ron H J Mathijssen, Marthe S Paats, et al. Clinical Utility of Circulating Tumor DNA in Patients With Advanced KRAS. J Thorac Oncol. 2024;19(7):995-1006. PMID: 38615940. doi: 10.1016/j.jtho.2024.04.007.
免疫疗法
100 项与 CGX-1007 相关的药物交易
登录后查看更多信息
研发状态
10 条进展最快的记录, 后查看更多信息
登录
| 适应症 | 最高研发状态 | 国家/地区 | 公司 | 日期 |
|---|---|---|---|---|
| 癫痫 | 临床1期 | 美国 | - | |
| 癫痫 | 临床1期 | - | - | |
| 癫痫 | 临床1期 | - | - | |
| 神经变性 | 临床1期 | - | - | |
| 神经变性 | 临床1期 | - | - | |
| 神经变性 | 临床1期 | - | - | |
| 神经退行性疾病 | 临床1期 | 美国 | - | - |
| 疼痛 | 临床前 | 美国 | - |
登录后查看更多信息
临床结果
临床结果
适应症
分期
评价
查看全部结果
| 研究 | 分期 | 人群特征 | 评价人数 | 分组 | 结果 | 评价 | 发布日期 |
|---|
No Data | |||||||
登录后查看更多信息
转化医学
使用我们的转化医学数据加速您的研究。
登录
或

药物交易
使用我们的药物交易数据加速您的研究。
登录
或

核心专利
使用我们的核心专利数据促进您的研究。
登录
或

临床分析
紧跟全球注册中心的最新临床试验。
登录
或

批准
利用最新的监管批准信息加速您的研究。
登录
或

生物类似药
生物类似药在不同国家/地区的竞争态势。请注意临床1/2期并入临床2期,临床2/3期并入临床3期
登录
或
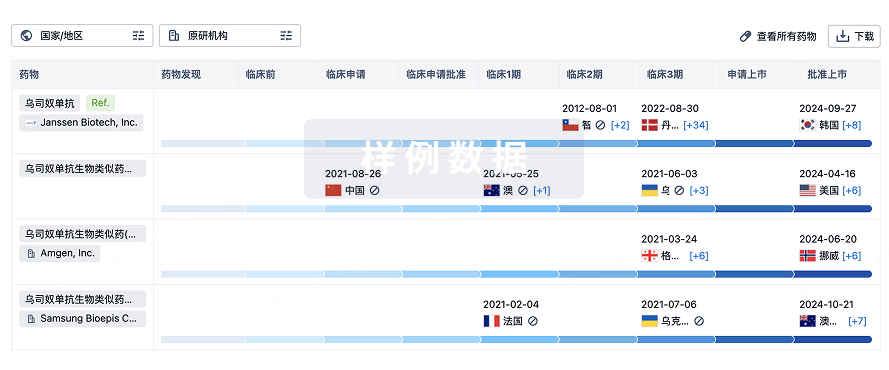
特殊审评
只需点击几下即可了解关键药物信息。
登录
或

生物医药百科问答
全新生物医药AI Agent 覆盖科研全链路,让突破性发现快人一步
立即开始免费试用!
智慧芽新药情报库是智慧芽专为生命科学人士构建的基于AI的创新药情报平台,助您全方位提升您的研发与决策效率。
立即开始数据试用!
智慧芽新药库数据也通过智慧芽数据服务平台,以API或者数据包形式对外开放,助您更加充分利用智慧芽新药情报信息。
生物序列数据库
生物药研发创新
免费使用
化学结构数据库
小分子化药研发创新
免费使用


