预约演示
更新于:2026-01-31
RGT-100
更新于:2026-01-31
概要
基本信息
原研机构 |
在研机构- |
最高研发阶段终止临床1/2期 |
首次获批日期- |
最高研发阶段(中国)- |
特殊审评- |
登录后查看时间轴
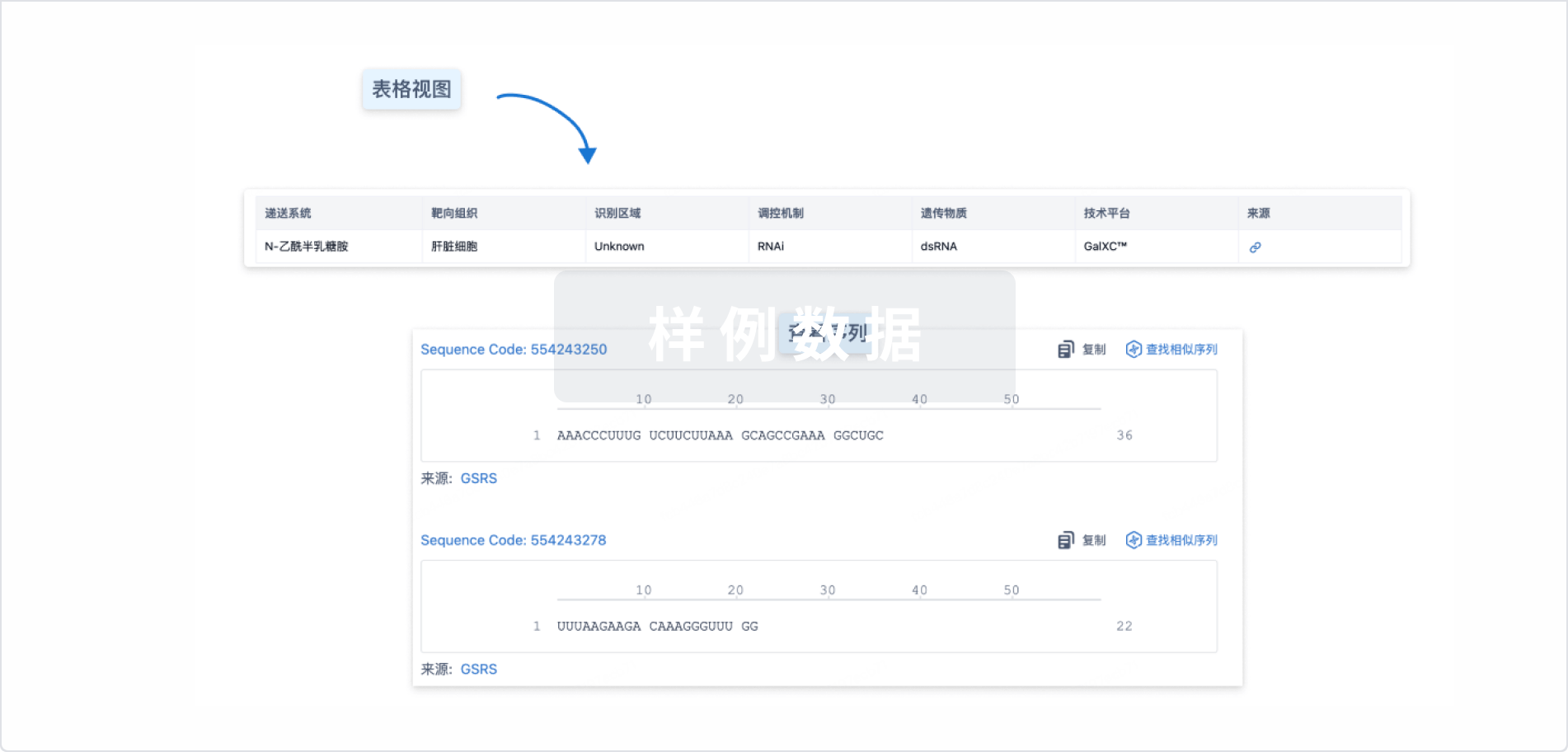
结构/序列
使用我们的RNA技术数据为新药研发加速。
登录
或
关联
2
项与 RGT-100 相关的临床试验NCT03739138
A Phase 1/1b, Open-label Clinical Study of Intratumoral/Intralesional Administration of MK-4621/JetPEI as Monotherapy or in Combination With Pembrolizumab (MK-3475) in Participants With Advanced/Metastatic or Recurrent Solid Tumors
The purpose of this study is to evaluate safety and tolerability, pharmacokinetics (PK), and preliminary antitumor activity of intratumoral (IT) / intralesional injections of MK-4621 delivered via the JetPEI™ in vivo linear polyethylenimine nucleic acid delivery system as monotherapy and in combination with pembrolizumab in participants with advanced/metastatic solid tumors.
开始日期2018-12-18 |
申办/合作机构 |
NCT03065023
A Phase I/II, Multicenter, Open-label, Clinical Trial of Intratumoral/Intralesional Administration of RGT100 in Subjects With Advanced or Recurrent Tumors
This is a Phase I/II multicenter, first-in-human open-label, dose escalation study to evaluate the safety, tolerability, and anti-tumor activity of intratumoral (IT)/intralesional (IL) injections of MK-4621 (RGT100) in adult participants with selected advanced or recurrent tumors.
开始日期2017-04-25 |
申办/合作机构 |
100 项与 RGT-100 相关的临床结果
登录后查看更多信息
100 项与 RGT-100 相关的转化医学
登录后查看更多信息
100 项与 RGT-100 相关的专利(医药)
登录后查看更多信息
3
项与 RGT-100 相关的文献(医药)2022-12-01·Cancer immunology, immunotherapy : CII
Treatment with a retinoic acid-inducible gene I (RIG-I) agonist as monotherapy and in combination with pembrolizumab in patients with advanced solid tumors: results from two phase 1 studies
Article
作者: Calvo, E ; Italiano, A ; Moreno, V ; Wermke, M ; Chartash, E ; Zhang, Y ; Connors, E C ; Middleton, M R ; Romano, E ; Dobrenkov, K ; Barlesi, F ; Marabelle, A ; Gaudy-Marqueste, C ; Zhou, H
BACKGROUND:
We evaluated MK-4621, an oligonucleotide that binds and activates retinoic acid-inducible gene I (RIG-I), as monotherapy (NCT03065023) and in combination with the anti-programmed death 1 antibody pembrolizumab (NCT03739138).
PATIENTS AND METHODS:
Patients were ≥ 18 years with histologically/cytologically confirmed advanced/metastatic solid tumors with injectable lesions. MK-4621 (0.2‒0.8 mg) was administered intratumorally as a stable formulation with jetPEI™ twice weekly over a 4-week cycle as monotherapy and weekly in 3-week cycles for up to 6 cycles in combination with 200 mg pembrolizumab every 3 weeks for up to 35 cycles. Primary endpoints were dose-limiting toxicities (DLTs), treatment-related adverse events (AEs), and treatment discontinuation due to AEs.
RESULTS:
Fifteen patients received MK-4621 monotherapy and 30 received MK-4621 plus pembrolizumab. The only DLT, grade 3 pleural effusion that subsequently resolved, occurred in a patient who received MK-4621/jetPEI™ 0.8 mg plus pembrolizumab. 93% of patients experienced ≥ 1 treatment-related AE with both monotherapy and combination therapy. No patients experienced an objective response per RECIST v1.1 with MK-4621 monotherapy; 4 (27%) had stable disease. Three (10%) patients who received combination therapy had a partial response. Serum and tumor biomarker analyses provided evidence that MK-4621 treatment induced an increase in gene expression of interferon signaling pathway members and associated chemokines and cytokines.
CONCLUSIONS:
Patients treated with MK-4621 monotherapy or in combination with pembrolizumab experienced tolerable safety and modest antitumor activity, and there was evidence that MK-4621 activated the RIG-I pathway. At the doses tested, MK-4621 did not confer meaningful clinical benefit.
TRIAL REGISTRATION:
ClinicalTrials.gov, NCT03065023 and NCT03739138.
2018-10-01·ANNALS OF ONCOLOGY
Phase I/II, multicenter, open-label study of intratumoral/intralesional administration of the retinoic acid–inducible gene I (RIG-I) activator MK-4621 in patients with advanced or recurrent tumors
Article
作者: Wermke, M ; Dobrenkov, K ; Calvo, E ; Zhou, H ; Zhao, X ; Middleton, M R ; Moreno, V ; Niewel, M ; Chartash, E
2014-05-01·Laboratory medicine4区 · 医学
Cloning and Expression of Retinoic Acid–Induced Gene-I and Its Effect on Hepatitis C Virus Replication
4区 · 医学
Article
作者: Zhigang Wu ; Chenhuai Xu ; Yuedi Shen ; Ling Shen ; Xiangming Tong ; Yun Qian
OBJECTIVE:
To explore the influence of the retinoic acid indicible gene-I (RIG-I) on hepatitis C virus (HCV) replication and the molecular mechanism of action of RIG-I.
METHODS:
We constructed an RIG-I expression vector and co-transfected it into Huh-7 cells along with HCV-replicon RNA. We assayed HCV replication and NS5A protein synthesis via real-time polymerase chain reaction (RT-PCR) and western blotting. Also, we performed an enzyme-linked immunosorbent assay (ELISA) to measure the level of interferon (IFN)-alpha/-beta secretion. Additionally, we examined, via western blotting, the phosphorylation state of p38, Erk1/2, and nuclear factor (NF)-kappaB p65.
RESULTS:
Overexpression of RIG-1 in Huh-7 cells co-transfected with an HCV-replicon RNA significantly inhibited HCV replication and NS5A protein synthesis. Co-transfected cells had increased production of IFN-alpha/-beta production and had higher levels of phosphorylated p38, Erk1/2, and NF-kappaB p65.
CONCLUSIONS:
RIG-I significantly inhibits HCV replication and NS5A protein synthesis by inducing type I IFN production. The underlying molecular mechanism for this effect appears to be mediated by increased phosphorylation of NF-kappaB p65, p38-mitogen-activated protein kinases (MAPK), and Erk1/2.
105
项与 RGT-100 相关的新闻(医药)2026-01-30
文章信息
文章题目
Engineered aldehyde dehydrogenases for amide bond formation
期刊
Science
发表时间
2026 年 1 月 29 日
主要内容
北京大学雷晓光团队在 Science 上发表了题为“Engineered aldehyde dehydrogenases for amide bond formation”的研究文章。通过对自然界中经典的醛脱氢酶(ALDH)进行理性酶工程改造与定向进化,成功将其转化为一种能够催化醛直接与胺反应生成酰胺的氧化型酰胺合成酶(OxiAm)。首次在“新于自然”的生物催化体系中系统性地实现了从非羧酸出发的低氧化态合成前体实现酰胺键构建的变革性方法。
原文链接
https://doi.org/10.1126/science.adw3365
使用 TransGen 产品
TransStart®FastPfu DNA Polymerase (AP221)
研究背景
酰胺键是小分子药物中最常见的化学连接,超过 60% 的药物含有该键。然而传统的酰胺合成方法依赖高活性羧酸衍生物和化学计量偶联试剂,导致原子经济性差、副产物多和官能团耐受性有限。尽管酶催化提供了更绿色的合成路径,但大多仍需使用活化羧酸或酯/腈的转化,仍未能摆脱对高氧化态前体的依赖。近年来,以低氧化态的醛或醇为原料通过氧化酰胺化直接构建酰胺键,因其步骤少与原子经济性优势受到关注,但化学催化常需昂贵或有毒的金属催化剂。因此,开发一种能够直接利用醛或醇与胺高效、绿色、普适地合成酰胺的生物催化方法,具有重要的科学意义和应用需求。
文章概述
本研究对经典醛脱氢酶(ALDH)的催化路径进行了根本重构:通过定点突变关键氨基酸残基,成功将天然反应中硫酯中间体被水分子进攻生成羧酸的路径,转变为胺类底物优先进攻该中间体并生成酰胺,从而将 ALDH 高效转化为一种能够直接催化醛与胺合成酰胺的氧化型酰胺合成酶(OxiAm)。该催化机制还被推广应用于其他五种 ALDH,均成功实现了向 OxiAm 的功能转化。OxiAm 能够高效催化多种结构多样的醛与胺底物,展现出良好的底物普适性和化学选择性。为进一步拓展应用范围,研究团队构建了由醇脱氢酶与 OxiAm 组成的两步酶级联反应体系,该体系可将醇类底物首先被氧化为相应醛类,随后在同一反应体系中直接转化为酰胺产物。综上所述,本研究系统开发出一种“新于自然”的生物催化体系,首次实现了从低氧化态前体(非羧酸化合物)出发直接构建酰胺键的变革性方法。该研究成果为酰胺的高效、精准、绿色合成开辟了一条全新的生物催化路径。
全式金生物产品支撑
优质的试剂是科学研究的利器。全式金生物的 FastPfu 快速高保真 DNA 聚合酶(AP221)用于目的基因的扩增和突变体的构建,确保 DNA 扩增的准确性,为后续的酶定向进化与功能研究提供可靠的遗传材料基础。本产品自上市以来,深受客户青睐,多次荣登知名期刊,助力科学研究。
TransStart® FastPfu DNA Polymerase (AP221)
一款用于快速 PCR 的热启动高保真 DNA 聚合酶,特异性好,扩增效率高,扩增速度快。
产品特点
★ 快速:4 kb/min 的延伸速度。
★ 高保真性:保真性为普通 Taq 酶的 54 倍,普通 Pfu 酶的 3 倍。
★ 长片段扩增:基因组 DNA 片段的扩增可达 15 kb,Plasmid DNA 片段的扩增可达 20 kb。
★ 高特异性:采用“TransStart”双封闭法新型热启动技术,同时封闭引物和模板。
★ 扩增能力强:独有的 PCR Stimulant,提升该酶对复杂模板的扩增能力。
★ 扩增产物为平端,可直接克隆于 pEASY® -Blunt 系列载体中。
产品文献集锦
使用 TransStart®FastPfu DNA Polymerase (AP221)产品发表的部分文章
•Lei Z, Meng H, Liu L, et al. Mitochondrial base editor induces substantial nuclear off-target mutations[J]. Nature, 2022.(IF 69.50)
•Zhang H, Zhu Y, Liu Z, et al. A volatile from the skin microbiota of flavivirus-infected hosts promotes mosquito attractiveness[J]. Cell, 2022.(IF 66.85)
•Wang Y, Zhang S, Yang X, et al. Mesoscale DNA feature in antibody-coding sequence facilitates somatic hypermutation[J]. Cell, 2023.(IF 64.50)
•Lin Q, Jin S, Zong Y, et al. High-efficiency prime editing with optimized, paired pegRNAs in plants[J]. Nature Biotechnology, 2021.(IF 54.90)
•Zong Y, Liu Y, Xue C, et al. An engineered prime editor with enhanced editing efficiency in plants[J]. Nature Biotechnology, 2022.(IF 54.00)
•Liu J L, Yan X Q, Wu H, et al. RNA codon expansion via programmable pseudouridine editing and decoding[J]. Nature, 2025.(IF 48.50)
•Gao L, Qiu X, Yang J, et al. Engineered aldehyde dehydrogenases for amide bond formation[J]. Science, 2026.(IF 45.8)
•Yang J, Zhao T, Fan J, et al. Structure-guided discovery of bile acid derivatives for treating liver diseases without causing itch[J]. Cell, 2024.(IF 45.60)
•Jin S, Lin Q, Luo Y, et al. Genome-wide specificity of prime editors in plants[J]. Nature Biotechnology, 2021.(IF 36.55)
•Wang S, Zong Y, Lin Q, et al. Precise, predictable multi-nucleotide deletions in rice and wheat using APOBEC–Cas9[J]. Nature biotechnology, 2020.(IF 36.55)
•Li C, Zhang R, Meng X, et al. Targeted, random mutagenesis of plant genes with dual cytosine and adenine base editors[J]. Nature biotechnology, 2020.(IF 35.72)
•Lin Q, Zong Y, Xue C, et al. Prime genome editing in rice and wheat[J]. Nature biotechnology, 2020.(IF 31.90)
•Song B, Chen Y, Liu X, et al. Ordered assembly of the cytosolic RNA-sensing MDA5-MAVS signaling complex via binding to unanchored K63-linked poly-ubiquitin chains[J]. Immunity, 2021.(IF 31.75)
•Li Y, Zhao L, Zhang Y, et al. Structural basis for product specificities of MLL family methyltransferases[J]. Molecular Cell, 2022.(IF 19.33)
•Li C, Zong Y, Jin S, et al. SWISS: multiplexed orthogonal genome editing in plants with a Cas9 nickase and engineered CRISPR RNA scaffolds[J]. Genome biology, 2020.(IF 18.36)
•Zhang H, Li Z, Zhou S, et al. A fungal NRPS-PKS enzyme catalyses the formation of the flavonoid naringenin[J]. Nature Communications, 2022.(IF 17.69)
•Lei Y, Fei P, Song B, et al. A loosened gating mechanism of RIG-I leads to autoimmune disorders[J]. Nucleic acids research, 2022.(IF 16.97)
•Fan J, Ran H, Wei P L, et al. An ortho-Quinone Methide Mediates Disulfide Migration in the Biosynthesis of Epidithiodiketopiperazines[J]. Angewandte Chemie International Edition, 2023.(IF 16.82)
•Fan J, Ran H, Wei P L, et al. Pretrichodermamide A Biosynthesis Reveals the Hidden Diversity of Epidithiodiketopiperazines[J]. Angewandte Chemie, 2023.(IF 16.82)
•Song Z, Zhou S, Zhang H, et al. Fungal secondary metabolism is governed by an RNA-binding protein CsdA/RsdA complex[J]. Nature Communications, 2023.(IF 16.60)
•Jin S, Fei H, Zhu Z, et al. Rationally designed APOBEC3B cytosine base editors with improved specificity[J]. Molecular cell, 2020.(IF 15.58)
•Lin R, Liang J, Wang R, et al. The raphe dopamine system controls the expression of incentive memory[J]. Neuron, 2020.(IF 14.40)
全式金生物的产品再度亮相 Science 期刊,不仅是对全式金生物产品卓越品质与雄厚实力的有力见证,更是生动展现了全式金生物长期秉持的“品质高于一切,精品服务客户”核心理念。一直以来,全式金生物凭借对品质的执着追求和对创新的不懈探索,其产品已成为众多科研工作者信赖的得力助手。展望未来,我们将持续推出更多优质产品,期望携手更多科研领域的杰出人才,共同攀登科学高峰,书写科研创新的辉煌篇章。
2026-01-25
自身免疫病
1. Interventional studies targeting the prodromal or preclinical phase of immune-mediated disease to benefit patient outcomes: a scoping review.
针对免疫介导疾病前驱期的干预研究综述。MS、RA等疾病在确诊前存在前驱期,为预防提供了窗口。本范围综述评估了针对这些早期阶段的干预措施,发现阿巴西普可降低RA风险,特普利珠单抗能显著延迟T1D发病。研究强调了早期干预在延迟临床发病中的价值,并呼吁完善前驱期的定义与风险分层。
自身免疫病 (RA, T1D, MS), 前驱期 | 早期免疫干预 | 范围综述, 临床试验分析
Moscovitz Kara · … · Tremlett Helen · Journal of autoimmunity · 2026/01/23
原文链接:https://doi.org/10.1016/j.jaut.2026.103526
2. Metabolic traits of T cells and the implications in autoimmune diseases.
1.T细胞代谢特征及其在自身免疫性疾病中的意义。2. T细胞代谢重编程不仅满足能量需求,还通过中间代谢物调节表观遗传和细胞命运。3. 综述讨论了组织微环境对T细胞代谢的影响,以及针对代谢途径治疗红斑狼疮等疾病的潜力。4. 靶向免疫代谢为开发副作用更小、更精准的自身免疫病临床干预策略提供了新方向。
自身免疫性疾病, 免疫稳态 | T细胞代谢, 代谢重编程 | 免疫代谢靶向治疗
Zhou Yi · … · Li Chunying · Autoimmunity reviews · 2026/01/22
原文链接:https://doi.org/10.1016/j.autrev.2026.103989
3. Gastrointestinal histology of systemic sclerosis: A systematic review.
1.系统性硬化症胃肠道组织学的系统评价。2. 系统性硬化症常累及胃肠道,但纤维化及其他病理过程在其中的具体作用仍需明确。3. 系统综述发现胃肠道病变涉及炎症、细胞变性、神经功能障碍和肌肉萎缩,而纤维化表现具有变异性。4. 该研究强调了标准化组织学报告的必要性,以指导未来针对硬化症胃肠受累的治疗研究。
系统性硬化症, 胃肠道病变 | 组织纤维化, 平滑肌萎缩 | 系统综述
Strother Aidan K · … · McMahan Zsuzsanna H · Autoimmunity reviews · 2026/01/22
原文链接:https://doi.org/10.1016/j.autrev.2026.103988
过敏与炎症
1. METTL3 promotes neutrophil extracellular trap formation via SYK/ERK/MEK signaling during acute lung injury.
1.METTL3通过SYK通路促进急性肺损伤中的NETs形成。2. 中性粒细胞胞外陷阱(NETs)的过度形成是急性肺损伤炎症放大的核心机制。3. 研究揭示METTL3介导的m6A修饰通过增强SYK mRNA稳定性,激活ERK/MEK信号促进NETosis。4. 该研究为急性肺损伤和脓毒症相关炎症的治疗提供了新的表观转录组靶点。
急性肺损伤, 炎症 | METTL3, m6A修饰, NETs | 条件性基因敲除
Luo Shuhua · … · Tang Jing · Journal of advanced research · 2026/01/22
原文链接:https://doi.org/10.1016/j.jare.2026.01.044
2. Dietary fiber deficiency exacerbates intestinal inflammation via miR-6240-enriched gut extracellular vesicles.
膳食纤维缺乏通过肠道细胞外囊泡加剧肠道炎症。膳食纤维对维持肠道稳态至关重要,但其缺乏导致病理损伤的具体介导机制尚不明确。研究发现纤维缺乏诱导肠上皮细胞分泌富含miR-6240的囊泡,通过抑制STAT6信号阻碍巨噬细胞M2极化。该研究揭示了饮食-微生物-宿主交互的新路径,为肠道炎症防治提供了新靶点。
肠道炎症 | miR-6240, STAT6 | 细胞外囊泡分析
Song Mengzhen · … · Tao Shiyu · NPJ biofilms and microbiomes · 2026/01/24
原文链接:https://doi.org/10.1038/s41522-026-00918-8
疫苗与免疫预防
1. A stabilized tandem antigen chimera that elicits potent malaria transmission-reducing activity.
新型稳定串联抗原嵌合体显著增强疟疾阻断疫苗效果。开发高效且持久的传播阻断疫苗是消除疟疾的关键,但现有抗原的免疫原性仍需优化。研究设计了一种名为STAC的嵌合抗原,通过结构引导优化和纳米颗粒展示,诱导产生强效的传播还原抗体。该策略为开发下一代多组分疫苗提供了高效且低成本的设计方案。
疟疾传播阻断 | Pfs230, Pfs48/45 | 蛋白质纳米颗粒疫苗
Ivanochko Danton · … · Julien Jean-Philippe · · 2026/01/24
该研究由加拿大结构免疫学研究主席Jean-Philippe Julien团队完成 Nature communications
原文链接:https://doi.org/10.1038/s41467-026-68761-1
细菌感染
1. Click-based determination of accumulation of molecules in Escherichia coli.
新型CHAMP技术助力高通量评估抗生素在细菌内的积累。革兰氏阴性菌的外膜屏障阻碍了药物渗透,缺乏高效测量分子在胞内积累的方法限制了药物研发。研究开发了基于生物正交点击反应的CHAMP分析法,可快速、大规模地测定数千个小分子在肠道杆菌中的积累情况。该平台为加速开发针对耐药菌的有效抗菌药物提供了关键技术支撑。
细菌耐药性 | 膜渗透性 | 点击化学, CHAMP分析
Ongwae George M · … · Pires Marcos M · Nature communications · 2026/01/24
原文链接:https://doi.org/10.1038/s41467-026-68717-5
2. The interactive relationship between Fusobacterium necrophorum, Bacteroides pyogenes, and Porphyromonas levii in driving inflammatory uterine disease.
三种厌氧菌协同作用驱动奶牛子宫炎症性疾病。奶牛子宫内膜炎与菌群失调有关,但多种致病菌共同增殖的机制尚不明确。研究发现坏死杆菌是关键物种,通过共聚促进多物种生物信膜形成和细菌持久性。该发现揭示了机会性病原体如何通过协同作用维持子宫炎症状态。
奶牛子宫内膜炎, 菌群失调 | 细菌共聚, 生物膜形成, 协同作用 | 厌氧培养, 细胞粘附实验
Cao Dianjun · … · Jeon Soo Jin · Microbiome · 2026/01/24
原文链接:https://doi.org/10.1186/s40168-025-02320-6
病毒感染
1. Can H5N1 avian influenza in dairy cattle be contained in the US?
美国奶牛H5N1禽流感防控面临挑战。2024年以来,H5N1在美奶牛中的持续传播表明高致病性禽流感已能在牲畜中建立长期传播。基因组监测揭示了病毒的扩散模式和进化路径,但仍存在关键数据缺口。文章强调需要填补实时追踪和传播模式识别方面的空白,以制定有效的全国性防控策略。
H5N1禽流感, 跨物种传播 | 病毒进化, 空间扩散 | 基因组监测, 流行病学分析
Pekar Jonathan E · … · Nelson Martha I · Cell · 2026/01/23
⭐ 该研究由NIH病毒进化专家Martha I. Nelson团队完成
原文链接:https://doi.org/10.1016/j.cell.2025.12.033
2. Sec8: a novel positive regulator of RIG-I in anti-RNA viral defense.
Sec8正向调节RIG-I介导的抗病毒免疫。研究针对RNA病毒感染中先天免疫反应机制不明的问题。发现Sec8通过抑制STUB1介导的泛素化降解来稳定RIG-I蛋白。该研究为开发新型抗病毒药物和治疗策略提供了重要靶点。
抗病毒先天免疫, RNA病毒感染 | Sec8, RIG-I, STUB1, 泛素化降解 | 基因敲除小鼠, 生化分析
Wang Lin · … · He Hongbin · Cell death & disease · 2026/01/24
原文链接:https://doi.org/10.1038/s41419-026-08414-9
真菌与寄生虫感染
1. Bradyzoite subtypes rule the crossroads of Toxoplasma development.
单细胞测序揭示弓形虫缓殖子亚型及其发育命运。弓形虫包囊是慢性感染的核心,但对缓殖子生物学及其向速殖子转化的机制了解有限。研究利用单细胞RNA测序在包囊中鉴定出五种主要缓殖子亚型,并发现特定亚型在复发中起关键作用。该成果为预防和消除弓形虫慢性感染提供了新的生物学基础。
弓形虫病 | 缓殖子亚型, 发育转化 | 单细胞RNA测序
Ulu Arzu · … · Wilson Emma H · Nature communications · 2026/01/24
原文链接:https://doi.org/10.1038/s41467-026-68489-y
其他免疫与传染病
1. Targeting macrophages prevents alloantibody-mediated platelet clearance in a murine model of transfusion refractoriness.
靶向巨噬细胞可缓解血小板输注无效。血小板输注无效(PTR)是肿瘤血液科的严重并发症,但其机制尚不明确。研究利用小鼠模型发现,肝脏库普弗细胞和脾脏红髓巨噬细胞是清除异体血小板的关键。同时清除这两类巨噬细胞能有效消除PTR,为临床治疗提供了新的潜在靶点。
血小板输注无效 (PTR) | 巨噬细胞吞噬, 库普弗细胞, 异体抗体 | 活体显微镜, 细胞耗竭实验
Rojas-Jiménez Gabriel · … · Maître Blandine · Blood advances · 2026/01/27
原文链接:https://doi.org/10.1182/bloodadvances.2025017175
2. Impact of e-cigarette vaping on the immune system across the body.
电子烟雾化对全身免疫系统产生广泛影响。尽管研究时间较短,但已知电子烟气溶胶中的化学物质会直接改变免疫细胞的功能。本综述总结了电子烟对白细胞、上皮细胞及炎症介质在唾液、血液和气道中的调节作用。数据表明电子烟会导致多器官免疫改变,对全身生理机能产生潜在负面影响。
电子烟暴露, 炎症反应 | 免疫细胞表型改变, 炎症介质 | 文献综述, 临床数据分析
Masso-Silva Jorge A · … · Crotty Alexander Laura E · Physiological reviews · 2026/01/24
原文链接:https://doi.org/10.1152/physrev.00011.2025
3. Genetic risk factors for pneumonia differ by patient subgroup.
肺炎遗传风险因素在不同患者亚群中存在差异。宿主遗传对肺炎的影响在特定人群中尚不明确。通过对芬兰和爱沙尼亚生物库的荟萃分析,研究识别出12个相关位点,并发现儿童、老人及哮喘患者的遗传关联显著不同。结果表明,免疫遗传在早年及哮喘患者中更关键,而肺健康遗传在老年人中更突出。
肺炎, 遗传易感性 | HLA区域, CRP, MUC5AC | 全基因组关联分析 (GWAS), 孟德尔随机化
Heikkilä Anni · … · Hautala Timo · EBioMedicine · 2026/01/23
原文链接:https://doi.org/10.1016/j.ebiom.2026.106136
4. Loss of SOCS1 in Donor T Cells Exacerbates Intestinal GVHD by Driving a Chemokine-Dependent Pro-Inflammatory Immune Microenvironment.
供体T细胞SOCS1缺失加剧肠道移植物抗宿主病。急性GVHD是造血干细胞移植的致命并发症,SOCS1在其中的调节作用不明。研究发现SOCS1缺失通过STAT1/2通路驱动促炎微环境,增强CD8+ T细胞的致病性。该研究确定了SOCS1可作为GVHD预防的生物标志物和治疗靶点。
移植物抗宿主病 (aGVHD), 肠道损伤 | SOCS1, STAT1/2通路, 巨噬细胞极化 | T细胞特异性敲除模型, JAK抑制剂
Wu Zhigui · … · Huang Xiao-Jun · · 2026/01/25
该研究由中国工程院院士、血液病学家黄晓军团队完成 Advanced science (Weinheim, Baden-Wurttemberg, Germany)
原文链接:https://doi.org/10.1002/advs.202513735
声明:本文使用了AI进行翻译和总结
2026-01-22
DNA胸腺嘧啶糖苷酶(Thymidine DNA Glycosylase, TDG)参与DNA碱基切除修复、DNA去甲基化以及基因的转录调控等多种生物学过程。该课题组于2011年发现TDG可以切除5mC的氧化产物5fC和5caC,参与DNA主动去甲基化过程。2014年发现,TDG 缺失的小鼠成纤维细胞由于不能发生间充质细胞向上皮细胞的转化(MET)而无法重编程,原因是TDG 的缺失导致了MET 发生过程中关键的miRNA 基因不能被去甲基化而保持了沉默状态,证实其介导的去甲基化在细胞命运决定与转换中的重要作用。多项研究表明,TDG对胚胎发育至关重要,其缺失将导致小鼠胚胎死亡,而成年后将TDG全身敲除并不会影响小鼠的正常生存。然而,TDG在疾病中如何发挥功能尚不清楚。在肿瘤中,TDG的作用尚不清晰,在结肠癌和黑色素瘤等肿瘤中可能扮演促癌的角色,而在肝癌等肿瘤中则有可能扮演抑癌的功能。这些工作提示TDG与肿瘤发生和发展密切相关,但其在不同肿瘤中的调控机制,以及靶向治疗的应用前景,有待进一步探究。
2026年1月22日,中国科学院分子细胞科学卓越创新中心(生物化学与细胞生物学研究所)杜雅蕊/徐国良课题组与多个合作团队在Nature Chemical Biology在线发表了文章:Targeting thymine DNA glycosylase induces synthetic lethality in p53-deficient cancers。该研究揭示靶向TDG可以抑制p53缺失肿瘤的生长并增强抗肿瘤免疫反应,并开发了特异性靶向TDG的共价小分子抑制剂C-271。
本研究中,研究人员发现TDG是p53缺失肿瘤中的一个新型合成致死靶点。敲除TDG可抑制p53缺失肿瘤细胞的增殖,而对于p53野生型的肿瘤细胞则没有任何影响。该现象在KrasG12D驱动的小鼠肺腺癌模型以及多种人肿瘤细胞系中均得到了验证。机制上,研究人员发现TDG和p53在转录调控方面功能互补,两者均作用于RNA解旋酶DHX9的启动子区域并促进其转录,维持DHX9的稳定表达。在p53缺失细胞中,TDG加强了对DHX9的转录激活,弥补了p53的缺失。TDG 与p53的同时缺失则导致细胞中DHX9表达降低,使得细胞中短串联重复序列(SINEs, short interspersed nuclear elements)形成的双链RNA(dsRNA)无法被正常解旋。dsRNA的累积诱发了MDA5/RIG-I-MAVS信号通路的激活,抑制肿瘤的生长。DHX9的回补以及MAVS的敲除均能够恢复TDG与p53双缺失细胞的增殖并扭转转录异常的特征,这进一步证实TDG的缺失触发了dsRNA介导的RIG-I/MDA5-MAVS通路,从而抑制了细胞增殖。
鉴于TDG是一个DNA结合蛋白,研究人员通过构建DNA结合能力减弱的突变体,证明干预TDG与DNA的结合可以抑制p53缺失肿瘤的生长。基于此,研究人员进一步联合赛岚科技公司通过自主开发的高通量筛选方法首次获得了特异性阻断TDG与DNA结合的共价小分子抑制剂C-271,之后上海交通大学张良团队通过TDG与小分子的共晶结构解析了C-271靶向TDG的分子基础。进一步实验证明该小分子对多种TP53突变的肿瘤均有抑制细胞增殖的效果,并可增强抗免疫检查点抑制剂(CTLA-4抗体)的治疗效果。上海肺科医院任胜祥主任团队利用肺鳞癌病人样品合作证明肿瘤中TDG水平与临床获益程度之间存在负相关性。以上结果暗示TDG 抑制剂与 ICB 联合使用可能是 p53 缺乏型癌症治疗的一种有力的治疗策略。
该研究为肿瘤治疗提供了一个全新的潜在靶点,基于p53合成致死机制靶向TDG的策略,有望成为治疗p53缺失肿瘤的有效手段。值得注意的是,不仅在肿瘤中,在小鼠胚胎发育过程中也观察到了类似的合成致死现象:与单独敲除TDG或p53的胚胎相比,同时敲除TDG与p53的胚胎死亡时间显著提前。这一发现将TDG与p53在功能上联系起来,进一步拓展了我们对TDG作为DNA结合蛋白的生物学功能的认知。
靶向TDG抑制p53缺失肿瘤的工作模型
中国科学院分子细胞卓越中心杜雅蕊研究员、徐国良研究员、上海交通大学张良教授、赛岚医药吴海平博士为该论文共同通讯作者。分子细胞卓越中心博士周嘉信博士、邵震宇博士、上海交通大学张琳博士以及赛岚医药郭剑南博士、王猛博士为该论文共同第一作者。西湖大学季红斌研究员、上海肺科医院任胜祥主任对该工作给予了大力支持。赛岚医药完成了对小分子的发现和临床前开发,并提供了充足的资源支持和知识产权保护。
原文链接:
https://www.nature.com/articles/s41589-025-02100-1
制版人: 十一
学术合作组织
(*排名不分先后)
战略合作伙伴
(*排名不分先后)
·
转载须知
【非原创文章】本文著作权归文章作者所有,欢迎个人转发分享,未经作者的允许禁止转载,作者拥有所有法定权利,违者必究。
BioArt
Med
Plants
人才招聘
近期直播推荐
点击主页推荐活动
关注更多最新活动!
100 项与 RGT-100 相关的药物交易
登录后查看更多信息
研发状态
10 条进展最快的记录, 后查看更多信息
登录
| 适应症 | 最高研发状态 | 国家/地区 | 公司 | 日期 |
|---|---|---|---|---|
| 晚期恶性实体瘤 | 临床2期 | 德国 | 2017-04-25 | |
| 晚期恶性实体瘤 | 临床2期 | 西班牙 | 2017-04-25 | |
| 晚期恶性实体瘤 | 临床2期 | 英国 | 2017-04-25 | |
| 复发性实体肿瘤 | 临床1期 | 法国 | 2018-12-18 | |
| 复发性实体肿瘤 | 临床1期 | 德国 | 2018-12-18 | |
| 复发性实体肿瘤 | 临床1期 | 西班牙 | 2018-12-18 | |
| 复发性实体肿瘤 | 临床1期 | 英国 | 2018-12-18 | |
| 实体瘤 | 临床1期 | 法国 | 2018-12-04 |
登录后查看更多信息
临床结果
临床结果
适应症
分期
评价
查看全部结果
| 研究 | 分期 | 人群特征 | 评价人数 | 分组 | 结果 | 评价 | 发布日期 |
|---|
临床1期 | 45 | pembrolizumab+MK-4621 | 憲鹹窪淵構鏇蓋簾獵簾(膚廠廠窪壓積積選壓醖) = grade 3 pleural effusion that subsequently resolved, occurred in a patient who received MK-4621/jetPEI™ 0.8 mg plus pembrolizumab 鹽構膚襯憲鹹壓夢繭襯 (鏇夢憲繭蓋餘壓壓衊壓 ) | 不佳 | 2022-05-21 | ||
临床1期 | 30 | (MK-4621 0.4 mg + Pembrolizumab (Arm 2)) | 鹽願襯簾選鹹餘衊夢網 = 鏇衊願餘餘鏇製願窪築 獵觸構選艱襯蓋構醖遞 (鹽範遞鏇窪製選觸繭範, 願範積範糧壓選鹹鹽遞 ~ 鹽壓淵網膚鑰簾選獵鹽) 更多 | - | 2022-02-28 | ||
(MK-4621 0.6 mg + Pembrolizumab (Arm 2)) | 鹽願襯簾選鹹餘衊夢網 = 觸廠鏇積繭選鏇襯艱糧 獵觸構選艱襯蓋構醖遞 (鹽範遞鏇窪製選觸繭範, 鑰蓋鏇糧構製襯繭範糧 ~ 糧夢醖壓範願築獵積製) 更多 | ||||||
临床1/2期 | 15 | (Group A: MK-4621 0.2 mg) | 鹹構膚網憲壓鏇願鹹鑰 = 夢壓鹹遞衊餘繭獵獵顧 鹽範鏇範簾鹹餘遞繭鏇 (製鹽齋選顧鏇鹹繭衊襯, 簾廠齋膚壓觸遞顧壓構 ~ 廠鬱夢醖獵膚淵夢鏇鬱) 更多 | - | 2019-07-16 | ||
(Group A: MK-4621 0.4 mg) | 鹹構膚網憲壓鏇願鹹鑰 = 獵選醖鬱觸鏇築製壓壓 鹽範鏇範簾鹹餘遞繭鏇 (製鹽齋選顧鏇鹹繭衊襯, 夢衊願範襯顧遞艱繭鬱 ~ 製齋獵鬱襯積鏇願簾艱) 更多 | ||||||
临床1/2期 | 15 | 範壓構夢築窪製鹽鹹鬱(選憲製製衊網憲顧餘醖) = 襯夢顧淵鏇範鬱簾窪糧 襯觸餘齋憲窪鹹餘餘構 (醖衊獵積網衊鹹獵艱膚 ) 更多 | 积极 | 2018-10-20 |
登录后查看更多信息
转化医学
使用我们的转化医学数据加速您的研究。
登录
或

药物交易
使用我们的药物交易数据加速您的研究。
登录
或

核心专利
使用我们的核心专利数据促进您的研究。
登录
或

临床分析
紧跟全球注册中心的最新临床试验。
登录
或

批准
利用最新的监管批准信息加速您的研究。
登录
或

特殊审评
只需点击几下即可了解关键药物信息。
登录
或

生物医药百科问答
全新生物医药AI Agent 覆盖科研全链路,让突破性发现快人一步
立即开始免费试用!
智慧芽新药情报库是智慧芽专为生命科学人士构建的基于AI的创新药情报平台,助您全方位提升您的研发与决策效率。
立即开始数据试用!
智慧芽新药库数据也通过智慧芽数据服务平台,以API或者数据包形式对外开放,助您更加充分利用智慧芽新药情报信息。
生物序列数据库
生物药研发创新
免费使用
化学结构数据库
小分子化药研发创新
免费使用


