预约演示
更新于:2025-05-07
MT1
更新于:2025-05-07
基本信息
别名 ALG1、ALG1 chitobiosyldiphosphodolichol beta-mannosyltransferase、Asparagine-linked glycosylation protein 1 homolog + [12] |
简介 Mannosyltransferase that operates in the biosynthetic pathway of dolichol-linked oligosaccharides, the glycan precursors employed in protein asparagine (N)-glycosylation. The assembly of dolichol-linked oligosaccharides begins on the cytosolic side of the endoplasmic reticulum membrane and finishes in its lumen. The sequential addition of sugars to dolichol pyrophosphate produces dolichol-linked oligosaccharides containing fourteen sugars, including two GlcNAcs, nine mannoses and three glucoses. Once assembled, the oligosaccharide is transferred from the lipid to nascent proteins by oligosaccharyltransferases. Catalyzes, on the cytoplasmic face of the endoplasmic reticulum, the addition of the first mannose residues to the dolichol-linked oligosaccharide chain, to produce Man1GlcNAc(2)-PP-dolichol core oligosaccharide. Man1GlcNAc(2)-PP-dolichol is a substrate for ALG2, the following enzyme in the biosynthetic pathway. |
关联
3
项与 MT1 相关的药物作用机制 MT1 inhibitors [+1] |
在研适应症 |
非在研适应症- |
最高研发阶段临床前 |
首次获批国家/地区- |
首次获批日期1800-01-20 |
US20230052740
专利挖掘靶点 |
作用机制- |
在研适应症 |
非在研适应症- |
最高研发阶段药物发现 |
首次获批国家/地区- |
首次获批日期1800-01-20 |
CN118546239
专利挖掘100 项与 MT1 相关的临床结果
登录后查看更多信息
100 项与 MT1 相关的转化医学
登录后查看更多信息
0 项与 MT1 相关的专利(医药)
登录后查看更多信息
2,303
项与 MT1 相关的文献(医药)2025-12-01·Molecular Biology Reports
Cellular signalling of melatonin and its role in metabolic disorders
Review
作者: Tripathy, Snehasis ; Bhattamisra, Subrat Kumar
2025-08-01·Tissue and Cell
METTL3-mediated m6A modification of MT1G inhibits papillary thyroid carcinoma cell growth and metastasis via Wnt/β-catenin pathway
Article
作者: Wang, Xueyu ; Liu, Jie ; Yao, Lei ; Wang, Kun ; Chen, Yating
2025-06-01·Toxicology Reports
Oxidative damage to DNA, expression of Mt-1, and activation of repair mechanisms induced by vanadium trioxide in cultures of human lymphocytes
Article
作者: Alcántara-Mejía, V A ; Álvarez-Barrera, L ; Mateos-Nava, R A ; Rodríguez-Mercado, J J ; Bahena-Ocampo, I U ; Beltrán-Flores, A A ; Bonilla-González, E ; Santiago-Osorio, E
11
项与 MT1 相关的新闻(医药)2025-05-02
01[68Ga]Ga-FL-031、[225Ac]Ac-FL-031生长抑素受体2(SSTR2)在神经内分泌肿瘤(NET)和小细胞肺癌(SCLC)等实体瘤中高表达,是放射性核素诊疗一体化药物(RDC)的理想靶点。本研究基于自主研发的UniRDC™技术平台开发了新型SSTR2靶向分子FL-031,并系统评估其诊疗同位素[68Ga]Ga/[225Ac]Ac标记物的临床前特性。通过高效液相色谱(radio-HPLC)检测显示,FL-031与68Ga、225Ac的标记放射化学纯度均>98%,比活度分别达18.5 MBq/nmol和185 kBq/nmol,符合诊疗应用标准。在SCLC-21H荷瘤模型中,3.7 MBq [68Ga]Ga-FL-031 PET/CT显像显示给药后1小时和3小时的肿瘤摄取显著高于正常器官(SUVmax=5.2 vs 周围组织<1.8)。体内分布实验证实,[225Ac]Ac-FL-031在AR42J NET模型呈现高效且持久的肿瘤滞留(72小时滞留率>60%ID/g),同时实现正常器官快速清除(48小时肾脏残留<5%ID/g)。更重要的是,37 kBq剂量组在SSTR2阳性模型中展现出显著抑瘤效应(肿瘤体积缩小72%,p<0.001),且未观察到体重下降或血液学毒性。该研究首次证实FL-031诊疗配对在SSTR2阳性肿瘤中兼具精准显像能力([68Ga]Ga-FL-031)与α核素靶向治疗潜力([225Ac]Ac-FL-031),为推进临床转化奠定了重要基础。AACR; Cancer Res 2025;85(8_Suppl_1): nr 583.相关往期链接:PSMA和SSTR2双靶新核药重磅来袭!203/212Pb靶向SSTR2新核药![203/212Pb]Pb-VMT-α-NET的首次临床研究!新核药[68Ga]Ga-DATA5m-LM4的首个临床研究!新核素螯合剂及探针Tb-Crown-TATE!02[68Ga]Ga-R54CXCR4作为趋化因子受体在血液肿瘤及实体瘤转移灶中高表达,是精准诊断与疗效预测的重要靶点。本研究针对现有CXCR4显像剂对实体瘤灵敏度不足的瓶颈,开发了新型PET示踪剂[68Ga]Ga-R54(专利号PCT/EP2020/087792)。在荷瘤裸鼠模型中,皮下移植的NCI-H69(小细胞肺癌)和H460(非小细胞肺癌)肿瘤在注射后1小时均显示特异性摄取(分别为12.2±1%ID/g和2±0.4%ID/g),且通过冷R54竞争实验证实其CXCR4靶向性(摄取分别降低90%和60%)。与[18F]-FDG相比,[68Ga]Ga-R54在H69模型中肿瘤摄取显著更高(12.2±1 vs 3.75±0.6%ID/g, p<0.001)。基于临床前数据,研究者设计了遵循ICH M3(R2)指南的首个人体微剂量临床试验(R4-FOUR-PET,EU CT:2024-513303-14-00)。该单中心、开放性研究计划纳入5-10例CXCR4高表达的晚期实体瘤(包括肺癌、乳腺癌等)患者,重点评估[68Ga]Ga-R54的药代动力学特性,并与[18F]-FDG进行显像对比,同时分析SUVmax与免疫组化CXCR4表达的相关性。GLP毒理实验显示,大鼠静脉注射1000倍临床等效剂量后14天内未见明显毒性,证实其安全性。目前试验已获伦理批准,正在意大利IRCCS-Pascale核医学科开展患者入组。本研究为CXCR4靶向诊疗一体化策略的临床应用提供了关键转化证据。AACR; Cancer Res 2025;85(8_Suppl_1): nr 676.相关往期链接:CXCR4靶向探针99mTc-PentixaTec的首个临床实验!CXCR4靶向PET探针!靶向CXCR4和整合素αvβ3的胰腺癌显像肽异二聚体的研制与评价优化linker减少CXCR4 PET探针的肾脏摄取肿瘤学中的CXCR4靶向诊疗方法03[68Ga]Ga-NNS309、[177Lu]Lu-NNS309成纤维细胞激活蛋白(FAP)在大多数上皮性肿瘤微环境的癌相关成纤维细胞(CAFs)中高表达,而在正常组织中表达受限,成为泛癌种靶向治疗的理想靶点。[177Lu]Lu-NNS309作为一种新型FAP靶向放射性药物,在胰腺导管腺癌(PDAC)和非小细胞肺癌(NSCLC)等临床前模型中显示出显著的肿瘤滞留与抗肿瘤活性。本研究(NCT06562192)为首个人体I期、开放标签、多中心试验,旨在评估[177Lu]Lu-NNS309在PDAC、NSCLC、乳腺癌(BC)及结直肠癌(CRC)患者中的安全性、耐受性及初步疗效。研究分为剂量递增与扩展两阶段。入组标准包括经治的局部晚期或转移性患者,且所有靶病灶(依据RECIST 1.1)需经[68Ga]Ga-NNS309 PET/CT确认显像阳性。剂量递增阶段采用基于EWOC原则的贝叶斯模型指导,综合安全性、耐受性、辐射剂量学及药效学数据确定推荐剂量(RD)。患者接受每周期单次给药,探索6周和4周两种给药方案。剂量扩展计划纳入约20例PDAC、20例NSCLC、30例HR+/HER2- BC(导管型与小叶型各15例)及24例三阴性BC患者。主要终点为安全性评估及RD确立,次要终点包括客观缓解率、药代动力学、肿瘤/器官辐射剂量学及[68Ga]Ga-NNS309显像特性验证。目前研究已启动剂量递增阶段入组,其设计为FAP靶向泛癌治疗策略的临床转化提供了关键范式,未来数据将推动放射性配体疗法在实体瘤中的广泛应用。AACR; Cancer Res 2025;85(8_Suppl_2): nr CT112.相关往期链接:FAP环肽核药[18F]AlF-FAP-NUR与[18F]FDG的头对头对比研究!OncoFAP核药叠叠乐新FAPI核药改善肿瘤剂量和靶本比!恶性胸膜间皮瘤与诊断核药68Ga-FAPI46首个64Cu标记的FAP靶向环肽FIH研究!04[225Ac]Ac-Macropa-(T/P)-PEG6-DM1针对HER2阳性乳腺癌对曲妥珠单抗及T-DM1的耐药难题,本研究开发了α核素偶联药物[225Ac]Ac-Macropa-(曲妥珠+PEG6-DM1)与[225Ac]Ac-Macropa-(帕妥珠+PEG6-DM1),并系统评估其协同疗效。通过构建曲妥珠单抗耐药(HCC1954,HER2高表达)及T-DM1/曲妥珠双耐药(JIMT-1,HER2中表达)小鼠模型,研究显示:抗体-药物偶联物(ADC)联用组在细胞内的内化效率显著高于单药(p<0.0001)。双模态显像([89Zr]Zr-DFO-T-PEG6-DM1 PET/[67Cu]Cu-DOTA-P-PEG6-DM1 SPECT)证实,放射性偶联物(ADR)在荷瘤模型呈现高特异性摄取(HCC1954肿瘤摄取率较非靶组织高8.6倍,JIMT-1高4.3倍)。毒理学评估表明,健康Balb/C小鼠间隔10天分次注射ADC与ADR组合,20天内血液学及生化指标均未出现显著毒性。在HCC1954模型中,ADR治疗组(22天)与ADC组(29天)均实现完全缓解(CR);而在JIMT-1耐药模型中,ADR组治疗60天后肿瘤体积降至11.5±4.4 mm³(3/8 CR),显著优于ADC组(无CR)。机制分析提示,策略通过协同阻断HER2二聚化结构域,增强核素靶向递送效率,并加速非靶组织清除(肝脏残留降低42%)。该研究首次证实α核素标记的抗HER2策略可克服现有靶向治疗耐药,为晚期乳腺癌提供全新解决方案。AACR; Cancer Res 2025;85(8_Suppl_1): nr 581.相关往期链接:蓝纳成新核药[225Ac]Ac-LNC1011正式亮相!212Pb席卷全球核素资本市场,正面宣战王牌核素225Ac,凭什么?212Pb与225Ac(一):核素性质与核资源储备ASCO 2024:225Ac-JNJ-6420临床I期碳酸酐酶Ⅸ(CA9)靶向的225Ac治疗核药!05SADA-PRIT针对恶性阑尾上皮肿瘤(AC)腹膜复发治疗困境,本研究创新性应用自组装-解组装双特异性抗体(SADA)预靶向放射免疫疗法(PRIT),通过靶向GPA33抗原实现α(225Ac)与β(177Lu)核素的高效递送。在皮下(SQ)及腹腔(IP)移植的AC患者来源异种移植(PDX)模型中,α-PRIT组(2×74 kBq 225Ac)与β-PRIT组(3×111 MBq 177Lu)分别将中位生存期(MS)从对照组的21天显著延长至70天和68天(p<0.0001)。尤为突出的是,IP模型中所有α-PRIT治疗小鼠(10/10)在100天观察期内保持无瘤生存,显著优于对照组的MS 42天。毒性分析显示,治疗组仅出现可逆性体重下降(<10%)及短暂白细胞减少(1-2级),组织病理学仅见轻度肾小管变性,未发现剂量限制性毒性。免疫组化证实26例AC样本及4种PDX模型均高表达GPA33、B7H3及HER2,为后续多靶点SADA抗体鸡尾酒疗法克服肿瘤异质性提供了理论依据。该研究首次验证SADA-PRIT在AC中的卓越疗效与安全性,为罕见肿瘤精准放疗开辟了新路径。AACR; Cancer Res 2025;85(8_Suppl_1): nr 2150.相关往期链接:通过[177Lu]Lu-PSMA证明Raion-TLC/HPLC分析的优化、临床实施和比较06[225Ac]Ac-Macropa-MUC-16针对MUC-16(CA125)靶点在80%上皮性卵巢癌(EOC)及65%胰腺导管腺癌(PDAC)中的高表达特性,本研究开发了新型α核素偶联药物[225Ac]Ac-Macropa-MUC-16,并系统评估其疗效与安全性。通过构建MUC-16高/中/低表达的EOC及PDAC患者来源异种移植(PDX)模型,研究显示:在SW1990 PDAC细胞系模型中,两次13 kBq剂量治疗组实现16.67%完全缓解(CR),其余83.33%肿瘤生长抑制≥50天(对照组33天进展至终点)。对于高/中表达MUC-16的PDAC及EOC PDX模型,治疗组CR率达100%,显著优于对照组中位生存期(PDAC 36天,EOC 47天)。低表达EOC模型虽未达CR,但第12天和21天的肿瘤生长抑制率仍达94.5±6.8%和84.7±11%。生物分布研究表明,药物在肿瘤组织滞留时间长达72小时(肝/脾清除半衰期<24小时),OLINDA剂量学模型预估肿瘤吸收剂量达12.5 Gy/MBq。安全性评估显示,两次15 kBq剂量虽引起轻度肝脾病理改变,但血液学及生化指标未见异常。该研究首次证实MUC-16表达水平是α靶向疗效的关键预测因子,为CA125阳性肿瘤的精准治疗提供了重要依据。AACR; Cancer Res 2025;85(8_Suppl_1):Abstract nr 567.相关往期链接:卵巢癌诊疗核药huAR9.607[161Tb]Tb-DOTA-N4MU01Nectin-4作为钙离子非依赖性的免疫球蛋白样细胞黏附分子,在三阴性乳腺癌(TNBC)及非小细胞肺癌(NSCLC)中异常高表达。本研究首次报道了161Tb标记的抗Nectin-4抗体([161Tb]Tb-DOTA-N4MU01)在TNBC及NSCLC模型中的诊疗潜力。通过稳定转染构建Nectin-4高表达的小鼠同源TNBC(4T1.Nectin-4、E0771.Nectin-4)及NSCLC(LLC.Nectin-4、CMT167.Nectin-4)细胞系,验证抗体结合亲和力(≤11 nM)及内化效率(流式细胞术与活细胞成像证实DOTA偶联不影响内化活性,p>0.999)。161Tb标记率达95%以上,药代动力学显示其血浆清除半衰期为120±20小时。在荷瘤模型中,SPECT/CT显像显示CMT167.Nectin-4与4T1.Nectin-4肿瘤在给药24小时后肿瘤/肌肉摄取比值分别达7.7和6.7。疗效实验表明:对于免疫检查点阻断疗法(ICBT)耐药的4T1.Nectin-4模型,2×5 MBq剂量组实现20%完全缓解(CR),中位生存期显著优于对照组(p≤0.0021);而抗PD-L1单药组无显著疗效(p=0.1166)。在侵袭性E0771.Nectin-4、CMT167.Nectin-4、LLC.Nectin-4及人源MDA-MB-468模型中,2×5 MBq剂量组均表现出显著肿瘤抑制(p≤0.0017-0.0316)及生存获益。安全性评估显示,两次5 MBq给药后28天内未观察到血液学或生化毒性。该研究首次证实161Tb标记的Nectin-4靶向疗法可克服ICBT耐药,为TNBC及NSCLC提供了新型诊疗一体化策略。AACR; Cancer Res 2025;85(8_Suppl_1): nr 584.AACR; 相关往期链接:Nectin-4靶向新核药:68Ga-FZ-NR-1的临床首秀!Nectin-4的PET新探针首次人体研究重磅来袭!靶向Nectin-4 ADC药物欧洲获批上市161Tb-AMTG 和 177Lu-AMTG的对比研究JNM:161Tb标记的PSMA核药重磅来袭!08[[177Lu]Lu-PSMA-I&T本研究通过构建RM1-PGLS去势抵抗性前列腺癌(PSMA稳定转染小鼠模型)同源移植瘤模型,首次揭示[177Lu]Lu-PSMA-I&T核素治疗诱导的免疫应答特征。荷瘤C57BL/6小鼠接受单次60 MBq [[177Lu]Lu-PSMA-I&T或5 Gy外照射(EBRT)后第7天,NanoString PanCancer免疫组学分析显示:核素治疗组显著激活109个差异表达基因(DEGs),其中98.2%呈上调趋势。单样本基因集富集分析(ssGSEA)鉴定出24条免疫相关通路活化,包括I型干扰素信号(NES=2.3)、抗原提呈(NES=2.1)及NK细胞介导细胞毒性(NES=1.9)等关键通路。免疫浸润分析显示巨噬细胞(p=0.004)、中性粒细胞(p=0.008)及CD56dim NK细胞(p=0.012)显著增加。核心发现聚焦于趋化因子Cxcl10的枢纽作用——在核素治疗组(log2FC=2.1, p=0.0059)与EBRT组(log2FC=2.3, p=0.0025)均呈现最显著上调。蛋白质互作网络分析显示,Cxcl10在两组分别以71(第1位)和124(第3位)条互作边数成为核心节点,调控包括STAT1、IRF7等关键免疫分子。体外实验证实,RM1-PGLS细胞经1.2 MBq/ml [[177Lu]Lu-PSMA-I&T(等效5 Gy照射)处理后,Cxcl10 mRNA表达于第7天达峰值(较基线升高4.1倍,p<0.001),而蛋白分泌峰出现在第2天(ELISA检测浓度提升3.8倍,p<0.05),第4-6天恢复基线水平。该发现提示肿瘤细胞具有独立于免疫微环境的Cxcl10分泌能力。本研究阐明[[177Lu]Lu-PSMA-I&T通过激活Cxcl10为核心的免疫网络重塑肿瘤微环境,为联合免疫检查点抑制剂等策略提供了分子机制层面的理论支撑。Cancer Res 2025;85(8_Suppl_1): nr 591.相关往期链接:19F/177Lu-rhPSMA-7.3和177Lu-PSMA I&T的较量!PET成像核素133La:225Ac和135La治疗类核素的soul mate!首个133La-PSMA-I&T!首次人体临床前评估:89Zr标记的PSMA-617和PSMA-I&T的药代动力学、PET成像和剂量测定。09[68Ga]Ga、[177Lu]Lu-BCY25286膜型基质金属蛋白酶1(MT1-MMP)作为肿瘤侵袭转移的关键调控因子,在非小细胞肺癌、膀胱癌等多种恶性肿瘤中高表达且与不良预后相关。本研究基于噬菌体展示技术开发了MT1-MMP特异性双环肽BCY25286,并实现其诊疗同位素标记(68Ga/177Lu)。临床前研究显示,[68Ga]Ga-BCY25286标记纯度>99%,对MT1-MMP具有高亲和力(7.2±1.6 nM)及72小时酶解稳定性。在荷瘤(MT1-MMP+ HT1080)裸鼠模型中,该示踪剂1小时肿瘤摄取达10.6±1.1%ID/g,肿瘤/肌肉比值为8.3,显著优于MT1-MMP阴性模型(p<0.001)。首个人体应用案例为65岁晚期肺腺癌患者,[68Ga]Ga-BCY25286 PET/CT显像(同情用药)显示原发灶及转移灶(经活检证实)均呈高摄取,与[18F]FDG显像结果高度一致。值得注意的是,骨转移灶SUVmax值较[18F]FDG提高42%(9.8 vs 6.9),而淋巴结转移灶摄取相当(SUVmax=6.2 vs 6.0)。药代动力学显示示踪剂主要通过肾脏排泄,60分钟时肾脏滞留量为15.3%ID/g。该研究首次证实MT1-MMP靶向显像在临床中的可行性,为基于该靶点的精准诊疗策略提供了关键转化证据。AACR; Cancer Res 2025;85(8_Suppl_1):Abstract nr 4601.相关往期链接:双环肽偶联物BT8009类似物:DOTA conjugated bicycle peptides叠加线粒体毒性的新PSMA靶向核药!非内化的ADC抗体药物偶联物!针对肿瘤微环境的放射性核素诊疗综述:针对多种癌症的个性化治疗方法10225Ac-SSO110SSO110(DOTA-JR11)作为生长抑素受体2(SSTR2)拮抗剂,相较于激动剂DOTATATE具有更高的靶点结合位点覆盖率和更长的肿瘤滞留时间,在临床前研究中展现出显著治疗优势。本研究系统评估了SSO110标记不同放射性核素(225Ac、212Pb、161Tb、177Lu)在SCLC(NCI-H69模型)及胰腺癌(AR42J模型)中的疗效差异。在NCI-H69 SCLC模型中,单次给药225Ac-SSO110(30 kBq,相当于人体剂量7.3 MBq)即可诱导肿瘤完全缓解(CR)及长期生存,显著优于225Ac-DOTATATE(42 kBq组中位生存期33天,p<0.001)。177Lu-SSO110(20 MBq)与161Tb-SSO110(20 MBq)虽有效,但均未达到CR。值得注意的是,212Pb-SSO110因SSO110长半衰期(>7天)与短半衰期核素(212Pb T1/2=10.6小时)匹配不佳,疗效不及177Lu-SSO110。在AR42J胰腺癌模型中,225Ac-SSO110(37 kBq)实现100%生存率,组织病理显示肿瘤细胞有丝分裂指数降低85%、坏死区域增加3倍,而同等剂量225Ac-DOTATATE组中位生存仅33天。机制分析揭示,SSO110的长效肿瘤滞留特性与长半衰期核素(如225Ac T1/2=9.92天)形成协同效应,使肿瘤吸收剂量提升2.4倍。基于177Lu-SSO110的LuSato1临床试验积极数据及225Ac-SSO110的卓越临床前活性,全球多中心I/II期试验(针对广泛期SCLC及Merkel细胞癌)已启动,旨在探索α核素靶向治疗与免疫检查点抑制剂的联合潜力。AACR; Cancer Res 2025;85(8_Suppl_1):Abstract nr 575.小核始终秉持客观中立原则,不发布主观评述或倾向性引导,内容严格援引公开信源,建议读者结合原文及多方信息祛魅思考,独立研判。●笔记:核药在ADC巨头第一三共研发进程中扮演的角色●Richard Zimmermann与Thomas Beyer共话核药行业格局●新型99mTc标探针用于黑色素瘤显像●Dr. Simone Krebs最新JNM:IL13Rα2靶向的免疫PET显像声明:本文观点仅代表小核本人,视角局限,欢迎批评指正;如需转载,请务必注明文章作者和来源。如有其它问题,请在本平台留言或联系xiaoheshuoheyao777@163.com,小核将在第一时间处理。内容编辑 | 小核排版设计 | 小核媒体合作 | 科研宣传 | 转载开白xiaoheshuoheyao777(小核微信)
AACR会议
2025-03-25
·精准药物
赖氨酸乙酰化为 BET溴结构域(BET bromodomain)蛋白的表观遗传读取域创建了对接位点,而这些蛋白已成为特异基因转录的主要调节剂。BET 抑制剂历经了泛 BET 抑制剂,到选择性的第二代抑制剂,以及高效的双功能和双重抑制剂。
1. 背景介绍
ω-氮赖氨酸残基的乙酰化是染色质结构的关键调节因子,该过程的失调与多种疾病的发展有关。组蛋白乙酰化是表观遗传密码的主要组成部分,控制基因进入转录机制的可及性,因此它受到两个酶家族作用的严格控制:“写入”乙酰化标记的组蛋白乙酰转移酶 (HAT) ,或“擦除”这些修饰的组蛋白去乙酰化酶 (HDAC)。HDAC 已成为主要的药物靶点,凸显了控制异常转录作为治疗策略的重要性。
乙酰化标记由溴结构域家族的表观遗传读取结构域特异性识别,该家族构成了人类蛋白质组中 61 个不同的结构保守的蛋白质相互作用结构域。 BET 家族由人类的四个成员(BRD2、BRD3、BRD4 和 BRDT)组成,每个成员都含有两个 N 端溴结构域(BD1、BD2)。将 pTEF 募集到转录活性的组蛋白乙酰化基因启动子上,在控制基础转录机制方面发挥着重要作用。重要的是,BET 蛋白是控制特异性转录的关键调节因子,它通过介导(超级)增强子中的蛋白质相互作用来控制特异性转录。 十多年前,人们发现了有效的溴结构域抑制剂,并且有望控制包括不可药用转录因子(如致癌基因 MYC)等致病基因家族,这促使人们开发出各种 BET 抑制剂,并针对该靶标家族开展临床研究。本文,总结了BET 抑制剂的最新进展及潜力。
pan-BET选择性抑制剂
2005 年,已鉴定出首批与溴结构域 (BD) 的乙酰赖氨酸 (KAc) 结合位点结合的骨架,其靶向乙酰赖氨酸转移酶 PCAF 的溴结构域,但在鉴定出首批强效且具有选择性的抑制剂后才开始迅速发展。3-methyltriazolothienodiazepines和methyltriazolobenzodiazepines是首批强效且具有选择性的泛 BET 抑制剂。结构研究表明,这些抑制剂与 BET 溴结构域的乙酰赖氨酸结合位点结合(图 1a)。三唑环充当乙酰赖氨酸模拟部分,通过模拟乙酰赖氨酸羰基与保守的天冬酰胺残基(BRD4 BD1 中的 N140)形成的氢键,以及与保守的酪氨酸(BRD4 BD1 中的 Y97)形成的水介导氢键。BET 溴结构域中还保留了一个结构水网络,从 Y97 延伸到乙酰赖氨酸结合位点和 ZA 通道的底部(图 1b)。3-methyltriazolothienodiazepines已被广泛用于设计溴结构域抑制剂,通常是通过对溶剂暴露区域进行修改(图 1c)。基于片段的发现和计算机模拟揭示了乙酰赖氨酸模拟片段的多样性,这表明 BET 溴结构域具有很好可成药性。 含有二甲基异恶唑核心的片段产生了多种新型泛 BET 抑制剂,包括有效的工具分子,如 iBET151 和 PLX51107、亚纳摩尔抑制剂 HJB97、以及 I-BET282E和 CPI-0610。此外,其他五元环体系,包括 4-methyl 1,2,3 triazoles和 2-thiazolidinones。
结构域选择性抑制剂
人 BET 溴结构域的高度序列同源性使结构域特异性抑制剂的开发具有挑战性,但最近的研究表明,通过小分子靶向第一个 BD1或第二个 BD2 溴结构域是可行的。RVX-208 与 BET 溴结构域的共晶体结构表明,RVX-208 与溶剂暴露残基相互作用,这些残基在 BD2 溴结构域内是保守的,但在 BD1 溴结构域中有所不同。
在最近开发的 BD2 选择性抑制剂 ABBV-744(图 2a、b)中,已经探索了这些残基变异,特别是BRD2 中的 H433、 D160、P430 和 K158。反过来,通过优化与 BD1 独特的 BC 环中天冬氨酸-赖氨酸残基的相互作用,实现了 BD1 选择性。葛兰素史克公司证明,可以利用针对 BD1 和 BD2 之间不同的残基来开发高选择性的 BD1 和 BD2 抑制剂 (图 2c、d)。
BD1 和 BD2 选择性抑制剂显示出显著的表型差异和对基因转录的不同影响。BD1 选择性抑制剂对癌细胞系和 mRNA 转录具有与泛 BET 抑制剂相似的广泛活性。相比之下,ABBV-744 仅在某些癌细胞中表现出抗增殖作用。例如,在前列腺癌细胞系中,ABBV-744 选择性地将 BRD4 从含 AR 的超增强子中置换出来,从而抑制了 AR 依赖性转录。然而,使用高 BD2 选择性抑制剂 iBET-BD2 并未观察到从启动子或增强子区域的置换,该抑制剂对改变预先存在的基因表达程序的影响很小,但阻断了驱动快速基因表达程序诱导的刺激反应。
双功能活性(BET+激酶) 抑制剂
BET 溴结构域是激酶抑制剂的常见脱靶物。两种高度多样化的靶标类别之间令人惊讶的交叉反应表明乙酰赖氨酸模拟部分在类药分子中很常见,这凸显了溴结构域出色的成药性。虽然临床激酶抑制剂(如 PLK1 抑制剂 BI-2536)的 BET 活性明显较弱,但随后对这些双重抑制剂进行优化,产生了具有平衡双靶标活性的化合物或由这些双重活性化合物衍生的选择性 BET 抑制剂。一些激酶抑制剂的 BET 活性也表明,这种双重活性可以合理设计用于治疗严重依赖两种靶标的疾病。这种想法导致了例如 ALK-BET 或 JAK-BET 双重抑制剂的出现。
在胰腺导管腺癌 (PDAC) 等难以治疗的癌症中,BET 和 HDAC 靶点类别之间存在协同作用,以此理性设计了第二类的双重抑制剂。将 HDAC 抑制剂 Vorinostat 和泛 BET 抑制剂 JQ1 结合起来,在 PDAC 细胞系中表现出协同作用,诱导细胞凋亡,并抑制 KRAS 突变小鼠的癌前病变,显著减少体内肿瘤体积,并且没有复发的迹象。从机制层面上讲,在 PDAC 中经常被沉默的 p57 (Cdkn1c) 在用两种抑制剂处理的细胞中协同去抑制。
事实上,早期概念验证抑制剂(如 JQ1 衍生物与 I 类 HDAC 抑制剂 CI994 (TW9))在体外表现出显著的协同作用,可抑制胰腺癌细胞生长。因此,BET 抑制剂与其他类别靶标的合理组合,可以显著降低体内实现显著抗肿瘤活性所需的 BET 抑制剂剂量。
双结构域抑制剂
BET 蛋白中存在两个相邻的溴结构域,这表明可以开发双功能抑制剂 (biBET) 来同时靶向 BD1 和 BD2。通过简单地将两个 JQ1 分子与基于 PEG 的连接体连接而开发的。与相应的单价抑制剂相比,所得抑制剂 MT1 的效力提高了约 100 倍。类似的设计思路促成了 MS654,它显示出对乳腺癌中 BRD4 的持续抑制。药代动力学优化使 AZD5153(图 3a,临床II期,AML)具有出色的体内效力。然而,AZD5153 中使用两种不同的乙酰赖氨酸模拟部分,3-methoxy-triazolo pyridazine和1,3 dimethylpiperazine-2-one,也导致溴结构域家族中存在一些脱靶活性。
第二类双功能抑制剂提高了 BET 家族的选择性,它们利用手柄与 E3 连接酶结合,将靶标募集到泛素蛋白酶体系统中,导致其药物诱导的降解。第一个 BET PROTAC,称为 dBET1,使用泛 BET 抑制剂 JQ1 和沙利度胺衍生物作为 E3 连接酶手柄开发,导致有效和选择性降解白血病细胞中表达的所有 BET 家族成员(BRD2-4)并强烈降解 MYC 水平。有趣的是,与 VHL 配体 VH032 连接 JQ1 的 PROTAC:MZ1(图 3b),其显示 BRD4选择性降解。该结构得到三元复合物(VHL-MZ1-BRD4)结构的理性解释。结果表明将 E3 募集到靶标并,与接头的优化导致靶标特异性降解,该策略可能允许降解具有组织特异性功能的其他 BET 异构体。组织或疾病特异性 E3 的配体的选择将导致 BET 家族中个别成员的限制性降解。然而,需要开发这些 E3 的新配体。
临床研究
BET 抑制剂对多种癌症的疗效进行临床研究。除了 NMC(一种罕见的癌症,其睾丸特异性蛋白 NUT 与 BRD3 或 BRD4 发生重排)外,大多数临床试验并未针对特定的分子亚型,这表明需要进行生物标志物研究,以确定决定抑制剂敏感性和耐药性的标志物。
由于其高度依赖于 NUT-BRD3/4 融合致癌基因,因此人们对使用泛 BET 抑制剂治疗这种侵袭性癌症的期望很高。然而,虽然在一些 NMC 患者中观察到部分反应,但用 BET 抑制剂治疗这种癌症并未达到基于令人信服的临床前数据的预期,原因仍有待阐明。
可能由于剂量限制性副作用(例如血小板减少、贫血、中性粒细胞减少、胃肠道事件和疲劳)导致患者无法达到治疗所需的剂量。BD2 选择性抑制剂或降低抑制剂剂量的联合疗法可能会产生更强劲的临床反应,尤其是当针对导致 BET 抑制剂耐药的途径时。
此外,AZD5153 等双功能抑制剂现已进入临床试验(NCT03205176,临床II期,2021-03-08),分子降解剂领域的快速进展使得针对 PROTAC 的 BET 抑制剂也有望很快进入临床研究。
BET溴结构域家族是非常火热的研发方向,特别是RBD4,以此开发了许多抑制剂。泛BET抑制剂的首次临床数据显示,剂量限制毒性、适度的抗肿瘤活性。除了开发更有效的抑制剂/降解剂,明确生物作用机制,未来还特别需要明确特异的临床适应症和生物标志物。PMID: 35462054
声明:发表/转载本文仅仅是出于传播信息的需要,并不意味着代表本公众号观点或证实其内容的真实性。据此内容作出的任何判断,后果自负。若有侵权,告知必删!
长按关注本公众号
粉丝群/投稿/授权/广告等
请联系公众号助手
觉得本文好看,请点这里↓
申请上市临床结果临床2期
2025-01-16
·VIP说
PREFACE
前言
2023年之前,核药还只是诺华和拜耳两家MNC重点谋划的方向,但在随后的两年,诸多MNC开始通过一系列合作、并购、投资强势入局,核药赛道热度不断飙升。
因此在JPM
2025上,各大MNC和biotech的核药布局与进展值得关注。
01
拜耳,核药领域先行者
拜耳作为较早布局核药赛道的MNC,早在2009年就与Algeta合作开发Xofigo,并于2013年获批上市,用于治疗晚期骨转移型去势抵抗性前列腺癌。
Xofigo上市后,销售额在2017年达到峰值(4.08亿欧元),后续开始逐年下滑。尽管Xofigo并没有如愿成为一款重磅炸弹,但是拜耳也从未停止对于核药赛道新疗法的布局。
根据拜耳本次JPM上的演讲PPT,2024年前三季度放射学板块整理营收达到15亿欧元,同比增长7%,表现亮眼。
在研管线方面,目前拜耳有两款核药正处于临床1期阶段,分别是BAY
3546828和BAY
3563254,从核素的选择上可以看出拜耳倾向于α核素,两款核药采用的核素均为225AC。
02
诺华,核药赛道王者
诺华2024年Q3收入同比增长10%,核心营业利润同比增长20%,2024年内三次上调收入预期,这样的表现在一众MNC中已是非常亮眼的存在。
如此结果得益于诺华多点开花、均衡增长的策略,其中在核药赛道的落子便是诺华迈出的关键一步,目前诺华手握两款RDC(核素偶联药物)Lutathera和Pluvicto,2024年前三季度分别贡献了5.34亿美元以及10.41亿美元销售额。
根据诺华在本次JPM上的演讲PPT,目前支撑诺华创新研发的共有五大技术平台,RLT(放射性配体疗法)便是其中之一,主要在肿瘤和免疫领域发力。
重磅RDC药物Pluvicto是诺华未来业绩增长的核心之一,预计销售峰值将超过50亿美元。在适应症拓展方面,预计在2025年获批pre-taxane进军1L
mCRPC,mHSPC的临床数据也预计将在2025年读出。
此外诺华还储备了多款核药管钱。如靶向GRPR的Lu-NeoB,目前正在探索乳腺癌等适应症,预计将在2026年读出数据;FXX489(177Lu-NNS309),靶点尚未公布;采用α核素以及靶向PSMA的Ac-PSMA-617和Ac-PSMA-R2,正在探索前列腺癌适应症的潜力,前者预计在2025H1进入临床3期,后者预计在2026年读出数据;以及AAA614(177Lu-FAP-2286),目前已经处于治疗实体瘤的2期临床,用于诊断的68Ga-FAP-2286也在同步开发中。
03
BMS:重金杀入核药领域
随着Pluvicto的起势,BMS也开始入局核药。2023年末以约250%的溢价收购了RayzeBio,从而囊获了RYZ101、RYZ801等创新靶向核药,以及GMP工厂和技术平台。
但BMS这笔收购后续研发并不太顺利,由于核素的短缺,BMS在2024年6月初暂停了RYZ101在研3期临床试验的患者入组,后续在解决了核素的供应问题后,BMS也是重启了这项临床试验。
根据BMS在本次JPM上的演讲PPT,在重金入局后,核药已经成为了BMS肿瘤板块四大重点投资方向之一。
BMS预计将在2025年读出RYZ101
1L治疗ES-SCLC的早期数据,在2026年读出RYZ101
2L+治疗GRP-NETs的注册临床试验数据。
此外,RYZ801目前正在探索治疗HCC适应症,并且在本次JPM召开前夕,BMS从艾博兹医药处,重新获取了RYZ801在大中华区开发和商业化的独家权力。
04
阿斯利康:
从合作到收购,切入核药赛道
阿斯利康在正式切入核药赛道前,先通过合作进行了试水。2020年11月,阿斯利康与Fusion
Pharmaceuticals达成了一笔总金额为4500万美元的合作,开发下一代基于α粒子的核药。
通过合作,阿斯利康看到了合作伙伴Fusion旗下Fast-Clear
linker的潜力,因而在核药升温之际,以最直接的方式,24亿美元收购Fusion,正式切入核药赛道,在获得技术平台的同时,还囊获了FPI-2265、FPI-1434、FPI-2068和FPI-2059四款在研核药。
在收购Fusion后的不久,阿斯利康再次投资了核药CDMO公司Nucleus
RadioPharma,开始全链条布局核药。
根据阿斯利康在本次JPM上的演讲PPT,RDC和ADC在未来将替代全身性放化疗,成为完成“2030愿景(2030年营收增至800亿美元)”的关键一部分。
目前AZ共有两款重点研发的核药FPI-2265和AZD2068。FPI-2265是一款采用α核素Ac-225的靶向PSMA的RDC,目前正在探索mCRPC适应症,已经启动2/3期临床。AZD2068(FPI-2068)同样采用的是α核素Ac-225,靶向c-MET和EGFR,目前处于临床1期。
05
Bicycle:拜耳、诺华的合作伙伴
除了多家MNC在本次JPM上分享了核药的内容外,还有一家与MNC达成深度合作的核药biotech也分享了公司的谋篇布局——专注于开发双环肽的Bicycle
therapeutics(由于Bicycle没有公开JPM演讲PPT,以下截图来自Bicycle在JPM召开前一日公开的deck)。
双环肽分子集抗体、小分子药物及肽类的特性于一身,具有与抗体类似的亲和性和精确的靶向特异性;同时,由于它们分子量较小,使得其能够快速深入地渗透组织,从而实现从组织内部靶向病灶;其肽类的性质则提供了“可调控”的药物动力学半衰期和肾脏清除途径,从而避免了其他药物形式中常见的肝脏和胃肠道毒性。
基于以上特性,Bicycle获得了诸多MNC的青睐,包括与诺华、拜耳在内的MNC达成了开发基于双环肽核药的合作。
在研发管线方面,Bicycle布局了多款双环肽放射性偶联药物(BRC)。
Bicycle此前公布了MT1-MMP
BRC的首个人体成像数据,验证了MT1-MMP作为抗肿瘤新靶点的潜力,以及双环肽分子将放射性同位素递送至肿瘤的能力。目前,Bicycle已经选择了将EphA2作为第二个BRC药物靶点。
并购临床3期临床1期放射疗法临床2期
分析
对领域进行一次全面的分析。
登录
或
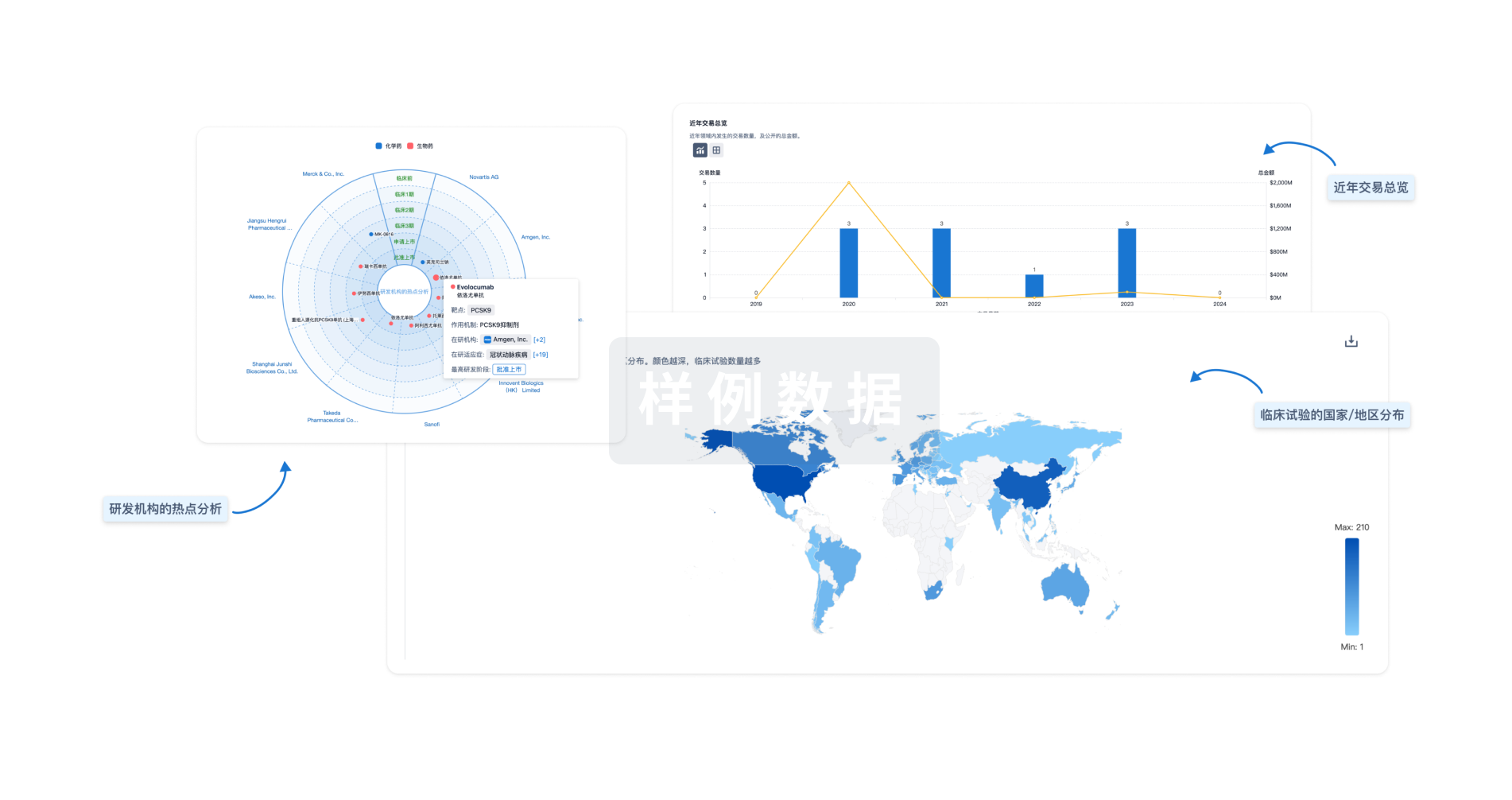
生物医药百科问答
全新生物医药AI Agent 覆盖科研全链路,让突破性发现快人一步
立即开始免费试用!
智慧芽新药情报库是智慧芽专为生命科学人士构建的基于AI的创新药情报平台,助您全方位提升您的研发与决策效率。
立即开始数据试用!
智慧芽新药库数据也通过智慧芽数据服务平台,以API或者数据包形式对外开放,助您更加充分利用智慧芽新药情报信息。
生物序列数据库
生物药研发创新
免费使用
化学结构数据库
小分子化药研发创新
免费使用




