预约演示
表观因子KMT2C/D突变三阴性乳腺癌脑转移的新机制
引言
乳腺癌是最常见的癌症,也是全球女性癌症相关死亡的主要原因,根据雌激素和孕激素受体以及人类表皮生长因子受体2(HER2)的表达,可将其分为激素受体阳性、HER2阳性或三阴性乳腺癌 (TNBC) 三种。这之中属TNBC远端转移的风险最高,预后最差,大约30%的转移性TNBC患者发生脑转移,总生存期最短【1】。
有研究发现,与原发乳腺肿瘤相比,组蛋白赖氨酸N-甲基转移酶2C(KMT2C)在远端转移中更常发生突变。同时,KMT2C和KMT2D在乳腺癌脑转移中也经常发生突变,提示了功能上的相关性。KMT2C和KMT2D可编码组蛋白H3赖氨酸4(H3K4)甲基转移酶,催化未甲基化的H3K4位点发生甲基化,形成单甲基化(H3K4me1)。H3K4me1e可在活性或准备状态的增强子和启动子区域富集,并能够招募其他组蛋白修饰酶,例如组蛋白H3K27的乙酰转移酶(例如P300)和去甲基化酶(例如KDM6A),对于基因表达的调控至关重要。KDM6A和KMT2C或KMT2D是SET1相关蛋白表观遗传调控复合体(COMPASS)的组成部分,并且KDM6A的结合依赖于KMT2C的存在,而与其催化活性无关。KMT2D与COMPASS复合体其他组分的结合也与其酶活性域无关。因此,KMT2C和KMT2D被认为是COMPASS复合体的核心蛋白,缺乏它们中的任何一个都会影响表观遗传景观【2,3】。然而,目前KMT2C/D突变如何影响表观遗传和转录组景观以促进肿瘤发生尚不清楚,哪些潜在途径可能驱动KMT2C或KMT2D缺陷肿瘤的转移也未可知。
近日,来自美国丹娜-法伯癌症研究所的Kornelia Polyak团队在Nature Cell Biology上在线发表了文章Loss of Kmt2c or Kmt2d drives brain metastasis via KDM6A-dependent upregulation of MMP3,发现在非转移性TNBC小鼠模型中,Kmt2c或Kmt2d的缺失会导致转移(尤其是脑转移),揭示了KMT2C或KMT2D缺失通过KDM6A依赖的方式促进MMP3的表达,从而驱动TNBC的脑转移的新机制,不仅在表观遗传层面为TNBC的治疗提供了新的视角,也为开发针对脑转移的靶向治疗策略提供了潜在靶点。

Preview
来源: 生物探索
本文研究人员发现,KMT2C和KMT2D是TNBC中最常见的突变基因,可分别在8.0和8.7%的肿瘤中检测到。与匹配的原发性TNBC相比,KMT2C和KMT2D的表达在远端转移中显著降低,提示KMT2C/D功能的丧失在转移中起作用。为了研究Kmt2c和Kmt2d缺乏的后果,研究人员在小鼠的乳腺脂肪垫(MFP)中注射带有H2B-mCherry标签的非转移性小鼠乳腺肿瘤细胞系168FARN,发现虽然原位肿瘤生长不受影响,但在注射了Kmt2c或Kmt2d基因敲除(KO)168FARN细胞的小鼠中,肝脏、肺和大脑中出现了mCherry+的微转移病灶。与匹配的原发肿瘤相比,转移瘤中Kmt2c和Kmt2d的表达普遍较低,表明它们确实来源于KO细胞。重要的是,Kmt2d KO细胞中没有Kmt2c的代偿性上调,反之亦然。
随后,研究人员对MFP注射的168FARN来源的原发性肿瘤进行了单细胞RNA测序(scRNA-seq)。根据mCherry和上皮标记物Krt8的表达识别肿瘤上皮细胞,并用已知的细胞类型特异性标记物来注释基质细胞,结果发现Kmt2c和Kmt2d KO的肿瘤细胞与野生型(WT)明显分离。差异表达基因(DGE)分析发现,与WT相比,两种KO肿瘤细胞中Ly6a、Bst2、Ifi27l2a和Stat1表达显著上调,暗示了一种通过潜在的干扰素信号激活而产生的促炎环境。与此同时,对非上皮细胞进行亚聚类和注释,并根据基因型对它们进行分组,发现KMT2C和KMT2D的缺失对肿瘤微环境中不同类型的细胞亚群有特定的影响,但Kmt2c或Kmt2d缺失相关的免疫抑制可能不是其转移表型的主要驱动因素。
为了剖析Kmt2c或Kmt2d缺失引起的细胞内在变化,研究人员首先关注了作为KMT2C和KMT2D的直接靶点的组蛋白。定量组蛋白质谱分析显示,与WT相比,两种KO细胞的组蛋白修饰相似,KMT2C或KMT2D基因敲除会导致H3K4me1和H3K27me3水平显著降低,而H3K27ac水平显著升高。通过染色质免疫共沉淀测序(ChIP-seq)技术,研究人员发现Kmt2c KO和Kmt2d KO细胞中H3K4me1和H3K27ac的峰值有显著的增加或减少,而H3K27me3的峰值变化不大。KDM6A——H3K27me3去甲基化酶,是KMT2C/D COMPASS复合物的一部分,KDM6A-ChIP-seq结果显示,与WT细胞相比,KO细胞中的峰值显著增加,且峰丢失很少。与此同时,RNA-seq分析显示,与WT细胞相比,Kmt2c KO和Kmt2d KO细胞存在大量DGE,这些基因表达的变化更多是独特的,而不是两者之间共享的,但都影响了上皮-间充质转换(EMT)的平衡。值得一提的是,即使在没有EMT的情况下,也能观察到Kmt2c和Kmt2d缺失促进转移的作用。进一步地利用结合与表达靶标分析(BETA)整合RNA-seq和ChIP-seq数据,研究人员发现在Kmt2c KO和Kmt2d KO细胞中,增加的H3K4me1、H3K27ac和KDM6A峰与上调基因显著相关,而丢失的H3K4me1和H3K27ac峰分别与下调基因显著相关,H3K4me1, H3K27ac和KDM6A具有相似的靶基因调控。这些结果提示KMT2C/D的缺失可通过影响染色质状态来调控基因表达,进而可能影响肿瘤的转移,而KDM6A可能是KMT2C/D缺失相关转录组变化的介质。
为了识别Kmt2c和Kmt2d KO细胞转移表型的共同表观遗传调控驱动因素,研究人员整合了H3K4me1和H3K27ac ChIP-seq中显著增加的峰以及在癌细胞RNA-seq和scRNA-seq中显著上调的基因,结合实验验证,证实基质金属蛋白酶3(Mmp3)是唯一重叠的基因。更重要的是,对临床样本的分析发现,与WT肿瘤相比,MMP3在KMT2C突变的TNBC中表达明显更高,并且 MMP3高表达肿瘤的KMT2C突变频率明显更高。进一步地利用shRNA在Kmt2c和Kmt2d KO细胞中下调Mmp3的表达,并将其注射到小鼠体内,结果表明,Mmp3的下调显著减少了Kmt2c和Kmt2d KO细胞的脑转移。已有的MMP抑制剂临床试验表明,其毒性高且疗效低。不过,庆幸的是,研究人员发现,通过shRNA或药理学抑制KDM6A能够减少Mmp3的表达,并在小鼠模型中减少Kmt2c和Kmt2d KO细胞的脑转移,表明通过靶向KDM6A间接抑制MMP3可以阻止KMT2C或KMT2D突变肿瘤的转移。此外,研究人员还发现,KDM6A的抑制对小鼠是耐受的,这表明KDM6A抑制剂可能在临床上对TNBC患者具有潜在的应用价值。
综上所述,本研究阐明了KMT2C/D突变导致的染色质重塑和组蛋白修饰变化,通过KDM6A间接影响MMP3的表达,从而诱导TNBC远端转移的分子机制。同时证明通过抑制KDM6A的活性,可以减少MMP3的表达,并可预防由KMT2C/D突变引起的脑转移(图1),不仅增进了人们对TNBC转移机制的理解,也为开发新的治疗策略提供了有价值的信息。
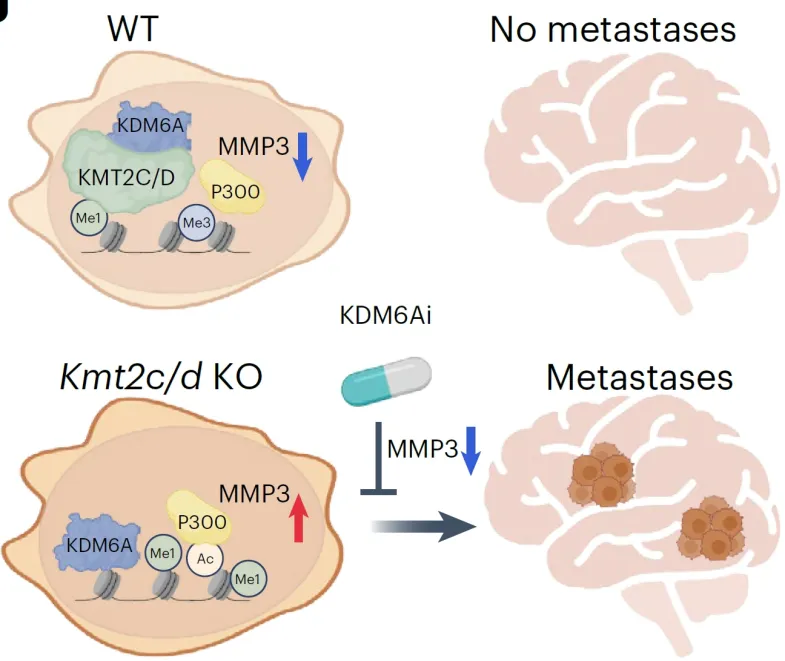
Preview
来源: 生物探索
参考文献
1. Darlix, A. et al. Impact of breast cancer molecular subtypes on the incidence, kinetics and prognosis of central nervous system metastases in a large multicentre real-life cohort. Br. J. Cancer 121, 991–1000 (2019).
2. Morgan, A. J., Giannoudis, A. & Palmieri, C. The genomic landscape of breast cancer brain metastases: a systematic review. Lancet Oncol. 22, e7–e17 (2021).
3. Sze, C. C. & Shilatifard, A. MLL3/MLL4/COMPASS family on epigenetic regulation of enhancer function and cancer. Cold Spring Harb. Perspect. Med. 6, a026427 (2016).
https://doi.org/10.1038/s41556-024-01446-3
内容来源于网络,如有侵权,请联系删除。
机构
-药物
生物医药百科问答
全新生物医药AI Agent 覆盖科研全链路,让突破性发现快人一步
立即开始免费试用!
智慧芽新药情报库是智慧芽专为生命科学人士构建的基于AI的创新药情报平台,助您全方位提升您的研发与决策效率。
立即开始数据试用!
智慧芽新药库数据也通过智慧芽数据服务平台,以API或者数据包形式对外开放,助您更加充分利用智慧芽新药情报信息。


